I TEST GENETICI ED IL LABORATORIO DI GENETICA: DAI TEST CLASSICI AI TEST DI NUOVA GENERAZIONE
|
|
|
- Corrado Boscolo
- 8 anni fa
- Visualizzazioni
Transcript
1 I TEST GENETICI ED IL LABORATORIO DI GENETICA: DAI TEST CLASSICI AI TEST DI NUOVA GENERAZIONE Metodi per l analisi di mutazioni specifiche Metodi generali per identificare la presenza di mutazioni in un dato gene. Roma 10 Giugno 2013
2 Biomarcatori Quando si parla di biomarcatore generalmente si intendono molecole, proteine, DNA, enzimi e quant altro possa avere un significato predittivo, prognostico, diagnostico I biomarcatori possono servire a: - ad identificare gli stadi precoci e sub clinici della malattia; - nella diagnosi di malattie; - a stratificare il rischio; - nel monitorare la progressione della malattia o la risposta alla terapia; - nella scelta del percorso terapeutico (se farmacologico oppure di altro tipo).
3 From bench to bedside: i criteri che devono essere soddisfatti prima che un biomarcatore possa passare dalla ricerca alla clinica 1. il biomarcatore può essere facilmente misurato dal clinico? Questo significa che devono essere disponibili test accurati e riproducibili da poter eseguire in qualsiasi parte del mondo e chiaramente a costi ragionevoli; 2. il biomarcatore aggiunge nuove informazioni a quelle già esistenti? La risposta a questo quesito deve provenire da evidenze forti e consistenti tra il biomarcatore e la malattia, validate in diversi studi di popolazione; 3. il biomarcatore deve essere di utilità per il clinico nel management del paziente (ad es. deve essere utile per indicare particolari strategie di trattamento nella cura dei pazienti).
4 Characteristics of a Detection System A good detection system should have 3 qualities: Sensitivity Specificity Simplicity Sensitivity means that the test must be able to detect very small amounts of target even in the presence of other molecules. Specificity: the test yields a positive result for the target molecule only. Simplicity: the test must be able to run efficiently and inexpensively on a routine basis.
5 Molecular Genetic Testing Disease Genes Identified Molecular Tests Offered in North America Molecular Tests Offered In Ontario nlm.nih.gov
6 Malattie mendeliane Malattie multifattoriali Ereditarietà Trasmissione mendeliana Autosomica recessiva Autosomica dominante X-linked Familiarità Malattia è più comune tra i parenti di un individuo affetto che nella popolazione in generale Transmissione nonmendeliana Rare Fibrosi cistica Sordità Distrofia muscolare di Duchenne Ipercolesterolemia familiare Comuni Ipertensione Asma Diabete mellito Malattia trombo-embolica Infarto miocardio Depressione Cefalea Osteoporosi Patologia tiroidea
7 Metodi per l analisi di mutazioni specifiche 1-PCR with restriction enzyme coupled analysis (RFLP). 2- PCR Allele specifiche (ARMS, SSP-PCR, SNAPShot ). 3-Oligonucleotide ligation assay, Dot Blot, Reverse Dot Blot. 4-OLA 5- Real time PCR
8 PCR - REAZIONE a CATENA della POLIMERASI E IL mezzo per produrre in vitro grandi quantità di una specifica sequenza di DNA, partendo da DNA estremamente complesso. 1 1 x 10 9 PCR DNA Il principio a cui si ispira la PCR è la replicazione del DNA: REPLICAZIONE Nuovo filamento Primer a RNA PCR DNA stampo 5 DNA polimerasi DNA stampo DNA polimerasi termostabile Nuovo filamento Primer oligonucleotidico Primers a RNA Nuovo filamento
9 I step primer 2 complementarietà al primer 2 II step 5 3 regione di interesse i filamenti vengono separati ed i primers si appaiano i primers si estendono Le FASI della PCR 3 5 primer 1 complementarietà al primer 1 i filamenti vengono separati ed i primers si appaiano III step 5 3 complementarietà al primer i filamenti vengono separati ed i primers si appaiano complementarietà al primer i primers si estendono i primers si estendono Frammenti desiderati (quelli di lunghezza variabile non sono mostrati) 3 5 filamenti di lunghezza variabile filamenti di lunghezza omogenea E così via 5 3
10 I primi 4 cicli della PCR in dettaglio Frammento desiderato 2 ciclo 3 ciclo 4 ciclo Amplificazione esponenziale 1 ciclo 35 ciclo DNA stampo Numero di copie di DNA contenenti il frammento desiderato 2 1 = = = = Numero di copie di DNA della corretta lunghezza contenenti il frammento desiderato
11 COMPONENTI di una REAZIONE di PCR Stampo DNA a doppio filamento Primers Oligonucleotidi complementari a regioni dei filamenti opposti che fiancheggiano la sequenza DNA bersaglio Deossiribonucleotidi trifosfati Miscela equimolare di datp, dttp, dgtp, dctp Tampone contenente cloruro di magnesio Lo ione Mg 2+ è essenziale per il funzionamento dell enzima Enzima Tradizionalmente viene usata la Taq polimerasi, enzima termostabile estratto dal batterio termofilo Thermus aquaticus
12 I FATTORI CRITICI della PCR Non utilizzare quantità eccessive di stampo, primers e/o dntp al fine di evitare amplificazioni non specifiche. Dosare accuratamente la [Mg 2+ ] in funzione della quantità di stampo, primers e dntp utilizzata Mg 2+ è indispensabile al funzionamento della Taq polimerasi, ma è chelato da stampo, primers e dntp. Se in eccesso causa una perdita di fedeltà da parte dell enzima. Selezionare l enzima più idoneo alle proprie necessità Emivita alla temperatura di denaturazione Processività Capacità di correzione degli errori
13 STRATEGIE per AUMENTARE la SPECIFICITA PCR CLASSICA TOUCHDOWN PCR Den. iniziale 3-5 min 94 C 1 ciclo Den. iniziale 3-5 min 94 C 1 ciclo Den. 30 sec 94 C Ann. 30 sec T m (-5 C) All. 1 min/kb 72 C 30 cicli Den. 30 sec 94 C Ann. 30 sec T m (+5 C) -1 C/ciclo All. 1 min/kb 72 C 10 cicli All. finale 7 min 72 C 1 ciclo Den. 30 sec 94 C Ann. 30 sec T m (-5 C) All. 1 min/kb 72 C 20 cicli Mantenimento 4 C All. finale 7 min 72 C 1 ciclo Mantenimento 4 C
14 STRATEGIE per AUMENTARE la SPECIFICITA HOT START Perché realizzare una PCR hot start? Durante l allestimento della reazione di PCR o durante il riscaldamento iniziale del campione si possono verificare situazioni di appaiamento non specifico fra primers e stampo. Fra 40 e 50 C la Taq polimerasi ha un efficiente attività polimerasica e può estendere i primers non correttamente appaiati. La PCR hot start previene la formazione di questi prodotti non specifici in quanto un componente chiave della miscela di reazione (enzima o MgCl 2 ) viene aggiunto dopo lo step di denaturazione iniziale. NOVITA : Enzimi Hot start Gocce di cera NESTED PRIMER PCR Procedura: L amplificazione è realizzata con un set di primers Il prodotto è riamplificato con un set di primers interni ai precedenti I a PCR pr. nested II a PCR pr. nested 3 I prodotti non specifici amplificati nella I a PCR non saranno amplificati nella II a 3 5
15 EF su gel di agarosio Principio Le molecole di DNA sono cariche negativamente e migrano in un campo elettrico verso il polo positivo con velocità inversamente proporzionali alle loro dimensioni (in paia di basi= pb). Concentrazione di agarosio ( %) Viene scelta in funzione della grandezza delle molecole da separare: alte concentrazioni sono utilizzate per piccoli frammenti di DNA e viceversa. Visualizzazione bande Mediante etidio bromuro (intercalante degli a. nucleici, emette fluorescenza arancio-rossa quando illuminato con luce ultravioletta). Valutazione risultati La grandezza dei frammenti di DNA viene valutata per confronto con marker di peso molecolare (frammenti di DNA a lunghezza nota) che vengono fatti correre sullo stesso gel. 15
16 Migrazione del DNA su gel Le molecole lineari di dsdna migrano a velocità inversamente proporzionali al logaritmo in base 10 del numero di paia di basi. Fattori che influenzano la migrazione Peso molecolare Concentrazione di agarosio Conformazione DNA Voltaggio applicato Intercalanti Tampone di elettroforesi
17 - mw mw + Amplificati 1-3: 143 pb 4-5: 375 pb 7-9: 2000 pb Condizioni sperimentali Gel: 2% agarosio Tampone: tris, EDTA, borato, ph V, 30 min Colorazione:etidio bromuro 17
18 Digestione con enzimi di restrizione RFLP - Gli enzimi di restrizione sono endonucleasi di origine batterica in grado di riconoscere specifiche sequenze di 4-8 nucleotidi e di tagliare in quelle posizioni il DNA. Mutazioni che aboliscono un sito di taglio Mutazioni che creano un nuovo sito di taglio - Mutazioni che modificano un sito di restrizione possono essere riconosciute direttamente mediante elettroforesi dopo digestione con l enzima di restrizione appropriato. 18
19 Sistema di restrizione-modificazione Negli anni 50 si notò che una preparazione di batteriofagi fatta crescere in un determinato ceppo di E.coli non era in grado di infettare con efficienza le cellule di E.coli di un altro ceppo. Nel 1965 Arber scoprì che il DNA del fago preparato ad es. nel ceppo C di E.coli era rapidamente degradato dopo il suo ingresso nel ceppo K. Il ceppo K restringe (inibisce) la crescita di fagi prodotti in E.coli di ceppo C. L analisi di un DNA di fago capace di resistere alla degradazione, rivelò, una decina di anni più tardi, la presenza di alcune basi metilate al suo interno. La degradazione del DNA del fago è dovuta all esistenza di un sistema di restrizione/modificazione controllato dalla cellula ospite per proteggersi dall introduzione di sequenze di DNA esogeno
20 Enzimi di restrizione Il primo enzima di restrizione fu isolato nel 1968 dal ceppo K di E. coli, ma il sito di taglio non fu identificato. Nel 1970, Smith et al. isolarono un enzima di restrizione dal ceppo Rd del batterio Haemophilus influenziae che fu chiamato HindII, che riconosce la sequenza a 6 pb: 5 - GT(T/C) (G/A)AC CA(A/G) (C/T)TG -5 L enzima non era in grado di tagliare il genoma di H. influenziae ma era capace di tagliare in oltre 40 punti il genoma del fago T7. Nel 1972 fu isolato un altro enzima di restrizione dal ceppo RY13 di E. coli, chiamato EcoRI, che riconosce la sequenza a 6 pb: 5 - GAATTC CTTAAG -5 Da allora sono stati isolati più di 900 enzimi di restrizione da circa 230 specie batteriche
21
22
23
24
25
26 26
27 27
28 I globuli rossi (o eritrociti o emazie) sono il componente principale del sangue. sono corpiccioli rotondi, di grandezza microscopica, tutti eguali, ripieni di una sostanza liquida: l emoglobina, che dà il colore rosso al sangue e che ha il compito di portare l ossigeno a tutte le cellule. La molecola emoglobinica è composta da 4 catene globiniche diverse tra loro per l ordinamento delle molecole di aminoacidi che le compongono. si distinguono: catene a (alfa), b (beta), d (delta), g (gamma). Famiglia dei geni della globina nell uomo e altri primati
29 LE EMOGLOBINE UMANE NORMALI VI SONO TRE TIPI DI EMOGLOBINA UMANA NORMALE: L Hb A CHE È PROPRIA DELL INDIVIDUO DOPO LA NASCITA, COSTITUISCE IL 98% DI TUTTA L EMOGLOBINA ED È COMPOSTA DA 2 CATENE GLOBINICHE a E 2 b; L Hb A 2, CHE È ANCH ESSA PRESENTE SOLO DOPO LA NASCITA, È COMPOSTA DA 2 CATENE a E 2 d, E COSTITUISCE IL RESTANTE 2%; L EMOGLOBINA FETALE (Hb F) CHE È PROPRIA DEL PERIODO DELLA VITA INTRAUTERINA ED È FORMATA DA 2 CATENE a E 2 g.
30 Diagnosi di Hb S: b6 Glu Val 180 pb 201 pb 256 pb 381 pb 88 pb 256 pb b A b S mutazione: codon b6 A - amplificato: 725 pb - enzima restrizione: Cvn I - abolizione sito di taglio T mw mw: marker di peso molecolare AA: soggetto normale AS: eterozigote per la mutazione SS: omozigote per la mutazione 30 Boehm,1989
31 Rivelazione del gene dell anemia falciforme mediante la tecnica degli RFLP applicata ai prodotti di PCR a) Amplificazione mediante PCR della porzione del gene della globina b contenente il tratto mutato. b) Digestione dei prodotti di PCR con l enzima CvnI. c) Pattern elettroforetico: AA condizione omozigotica per il gene wt; AS condizione eterozigotica; SS condizione omozigotica per il gene CCTGAGGAG (wt) CCTGTGGAG (mutante) CvnI riconosce la sequenza CCTNAGG. L endonucleasi di restrizione mostra 3 siti di taglio sulla sequenza wt e 2 sulla sequenza mutata (anemia falciforme).
32 Diagnosi della variante Mediterranea della G6PD - Mutazione: nt 563 C T - Amplificato: 547 pb - Enzima: Mbo II - Creazione di un sito di taglio Normale 377, 119, 26, 25 Frammenti dopo digestione (pb) Mutato omozigote 277, 119, 100, 26, 25 Mutato eterozigote 377, 277, 119, 100, 26, 25 32
33 Amplificazione allele-specifica ARMS (Amplification Refractory Mutation System) - Reazione di PCR nella quale vengono utilizzati separatamentemente primers complementari alla sequenza normale od a quella mutata. A: primer normale B: primer mutato Normale: amplificato solo con A Mutato omo: amplificato solo con B Mutato etero: amplificato con A e B - La presenza degli amplificati viene evidenziata mediante elettroforesi su gel di agarosio e colorazione con bromuro di etidio. 33
34 Advantages Rapid, reliable and non-isotopic. Single tube multiplexing is possible with the more recent fluorescent ARMS. Disadvantages The original format did not distinguish between homozygotes and heterozygotes, except for the F508del mutation. However, fluorescent ARMS can distinguish between homozygotes and heterozygotes. Questa metodica identifica sostituzioni nucleotidiche e piccole delezioni o inserzioni. La tecnica consiste nell'amplificazione del DNA mediante PCR utilizzando oligonucleotidi complementari alla sequenza normale e mutata. Si eseguono pertanto due amplificazioni per campione, una con primers normali ed un'altra con primers mutati. In presenza di mutazioni allo stato omozigote l' amplificazione avverrà solo con con primers mutati e viceversa. I soggetti eterozigoti per la mutazione amplificheranno il loro DNA in entrambe le condizioni. Advantages 34
35 Una variante a questa tecnica è rappresentata dalla ARMS multipla che utilizza più primers mutati in un unica reazione. Questa tecnica consente di studiare un soggetto per più mutazioni ed è utilizzata correntemente per l'analisi molecolare della talassemia e della fibrosi cistica.
36 PCR allele-specifica (SSP-PCR) reazioni di PCR allele-specifica (SSP-PCR) in cui, per ogni reazione, vengono utilizzati un primer Forward (F) e due Reverse (R1 e Rtag) marcati utilizzando il fluorocromo 6-FAM, specifici per i 2 alleli della variante da identificare. Tutti i primers Rtag, che differiscono dal primer R1 solo per la base da discriminare all estremità 3, vengono disegnati legando alla sua specifica sequenza una coda nucleotidica per aumentare le dimensioni dell amplicone e permettere di discriminare i due alleli nella corsa elettroforetica.
37
38
39
40 Genotyping by SNaPshot Multiplex Genotyping method based on a single-base extension, that is, each probe binds to complementary DNA template in the presence of fluorescently labeled ddntps and AmpliTaq DNA Polymerase. Polymerase extends probe by one base, adding one ddntp to 3 end. Separation of nucleotide peaks occurs on ABI instrument.
41 ELECTROPHEROGRAM OF FOUR MULTIPLEXED SNPs IN ABCG2-- SHOWING HOMOZYGOTES BLUE=G GREEN=A RED=T BLACK=C ORANGE =STD STD 35T/T 40G/G 45G/G 50T/T STD BLUE=G GREEN=A RED=T BLACK=C ORANGE =STD STD 35C/C 40A/A 45A/A 50C/C STD Note: Because of influence of dye on mobility shift of the DNA fragments, reported sizes differ by about 5 bases from actual sizes of probes. 5
42 ELECTROPHEROGRAM OF FOUR MULTIPLEXED SNPs IN ABCG2 -- SHOWING HETEROZYGOTES BLUE=G GREEN=A RED=T BLACK=C ORANGE =STD STD C T G A G A C T STD BLUE=G GREEN=A RED=T BLACK=C ORANGE =STD STD C T G A G A C T STD 6
43 ThromboFil Genotipizzazione molecolare del Fattore V di Leiden, Fattore II, MTHFR C677T, MTHFR A1298C attraverso Multiplex PCR / elettroforesi capillare Mutazione Gene Localizzazione Variazione Nucleotidica Variazione Aminoacidica G1691A Fattore V Esone 10 G-1691 A Arg-506 Glu G20210A Fattore II 3' UTR G A 3' Splice Mut. C677T MTHFR Esone 4 C-677 T Ala Val A1298C MTHFR Esone 8 A-1298 C Glu Ala
44 Ibridazione con oligonucleotidi allele-specifici ASO probe (allele-specific oligonucleotide probe) - La presenza di una mutazione viene riconosciuta facendo reagire il DNA amplificato con sonde oligonucleotidiche complementari alla sequenza mutata o normale (separatamente). - Solo in presenza del 100% di omologia tra le sequenze si avrà l ibridazione della sonda con il DNA. La tecnica viene realizzata mediante: 1. Dot blot 2. Reverse dot blot 44
45 Dot blot Amplificazione DNA Denaturazione Immobilizzazione DNA su filtro nylon/cellulosa + Oligonucleotidi di sintesi marcati ( 32 P) complementari sequenza normale e mutata Ibridazione con la sequenza complementare di DNA Esposizione filtro a lastra radiografica autoradiografia Se la rezione di ibridazione è avvenuta si avrà la comparsa di una macchia 1 macchia con oligo mutato: omozigote per la mutazione 1 macchia con oligo normale: normale 2 macchie con oligo normale e mutato: eterozigote per la mutazione 45
46 Esempio di analisi mediante dot blot Diagnosi prenatale per b-talassemia major ASO ASO Madre Padre Controllo negativo Controllo positio Figlia Feto (Portatore eterozigote) Cimentato con oligo mutato Cimentato con oligo normale Boehm,
47 2. Reverse dot blot (RDB) Esempio di test per la rivelazione di mutazioni b-talassemiche Oligonucleotidi complementari alle sequenze normali (N) e mutate (M) per 8 diverse mutazioni per la b-talassemia, sono immobilizzati su pozzetti di una micropiastra. Amplificazione DNA con primers biotinilati Denaturazione 47
48 Schema della reazione (nel caso di un campione normale) Oligonucleotidi allele-specifici immobilizzati nei pozzetti Ibridazione con DNA biotinilato del campione. Lavaggio Aggiunta coniugato SA-HRP. Lavaggio Aggiunta del substrato H 2 O 2 e TMB Lettura a 450 nm SA-HRP: streptavidina-perossidasi TMB: tetrametilbenzidina 48
49 Risultati La rivelazione dell avvenuta ibridazione attraverso una reazione enzimatico-colorimetrica (colore giallo), indica quali sequenze (nomali o mutate) sono presenti nel campione. 49
50
51
52
53
54
55
56 REAL-TIME PCR quantificazione dell espressione genica identificazione del prodotto di amplificazione specifico rispetto a prodotti aspecifici analisi simultanea di due campioni nella stessa reazione individuazione di mutazioni puntiformi
57 AMOUNT OF DNA AMOUNT OF DNA Log[DNA] PCR quantitativa Real-Time Resa teorica della PCR: 2 n Il prodotto (P) incrementa esponenzialmente con il numero di cicli di PCR (n) Il prodotto di PCR dipende da T, numero di copie di template di partenza P=(2) n T fase esponenziale PCR CYCLE NUMBER plateau plateau fase esponenziale PCR CYCLE NUMBER Resa effettiva: effetto plateau Il processo di duplicazione non procede all infinito, esso è limitato da: Quantità dei primers Attività della Taq polimerasi Reannealing dei filamenti Raggiunto il plateau non si osserva più un incremento nei prodotti
58 PCR product Il plateau non dipende dal numero iniziale di molecole stampo T Anche se la quantità iniziale di template è la medesima, il plateau è raggiunto in tempi diversi ed in cicli diversi Cycle Utilizzare i dati ottenuti durante la fase esponenziale in cui il prodotto di PCR è proporzionale al template iniziale. Questo è reso possibile mediante il rilevamento di una fluorescenza che è proporzionale al prodotto di PCR. La fluorescenza, durante ogni ciclo di amplificazione, può essere rilevata utilizzando uno strumento quantitativo e dei marcatori fluorescenti il cui accumulo segue la stessa cinetica della reazione di PCR.
59 La Real-Time misura l'amplificazione in tempo reale durante la fase esponenziale della PCR, quando cioè l'efficienza di amplificazione è influenzata minimamente dalle variabili di reazione, permettendo di ottenere risultati molto più accurati rispetto alla PCR tradizionale "end point. 1. halogen tungsten lamp 2a. excitation filters 2a. excitation filters 4. sample plate 2b. emission filters 3. intensifier 5. ccd detector Il segnale di fluorescenza può essere generato da: 1) coloranti fluorescenti che si intercalano nel DNA a doppio filamento; 2) da sonde fluorescenti sequenza-specifiche. Incremento di fluorescenza Curva di amplificazione Cicli di PCR
60 Nella Real-Time PCR, i primi cicli, in cui non è misurabile la variazione nel segnale della fluorescenza, definiscono un importante parametro: la linea base (baseline) della curva. Linea di base Ct Linea soglia Un aumento della fluorescenza oltre la linea base indica il rilevamento del prodotto di PCR in fase di accumulo. Un secondo parametro importante è la linea-soglia: tale linea, parallela alla linea di base, deve tagliare le curve dei campioni nella loro fase di crescita esponenziale. La curva di amplificazione di ogni campione taglia la linea-soglia in un punto, chiamato ciclo-soglia (threshold cycle) definibile come il numero del ciclo (o frazione di questo numero) in cui la curva di amplificazione del campione in fase esponenziale taglia la linea-soglia.
61 Il diagramma del logaritmo delle quantità iniziali di DNA nei confronti dei valori di ciclo-soglia è una retta. Ciò significa che, se nella reazione erano presenti campioni a quantità nota di DNA, standards ( ), dai quali viene ricavata una retta di riferimento, diventa possibile calcolare la quantità di DNA iniziale presente in campioni oggetto di studio ( ). S1 S2
62 SYBR GRENN All inizio dell amplificazione le molecole di colorante non sono legate e producono una fluorescenza molto debole. Infatti il SYBR GRENN legato al dsdna, se eccitato, fluoresce con un intensità 100 volte maggiore di quando è libero in soluzione. Dopo l annealing dei primers, si legano poche molecole fluorescenti alla doppia elica. Il colorante si lega al solco minore del DNA. Durante l elongazione le molecole di colorante si legano al DNA neosintetizzato. Se la reazione è monitorata continuamente si ha un aumento di fluorescenza in realtime.
63 Il fatto che SYBR GREEN si leghi al dsdna senza specificità di sequenza fa si che si debba verificare che il segnale ottenuto non sia dovuto a prodotti aspecifici o dimeri di primers. Per questo motivo alla fine dell amplificazione si fa una curva di melting o di dissociazione. La curva di melting consiste in una rampa di temperatura che parte al di sotto del punto di melting dei prodotti di amplificazione e gradualmente aumenta fino ad oltrepassare la Tm degli stessi. Ciò permette di distinguere i vari prodotti di PCR sulla base della temperatura relativa di dissociazione, che dipende dalla lunghezza della sequenza e dal contenuto in G/C. Quando i 2 filamenti si separano la fluorescenza decresce velocemente. Il software rielabora le variazioni di fluorescenza, Relative Fluorescence Units, (RFU) in funzione del tempo (T) secondo la seguente (-d(rfu)/dt) espressione: Il picco rappresenta la melting temperature (Tm). prodotti specifici prodotti aspecifici
64 Analisi infezione Citomegalovirus Diagnosi tradizionale: I metodi tradizionali per la rilevazione e l identi ficazione del CMV comprendono analisi sierologiche per il rilevamento di anticorpi, isolamento del virus mediante colture cellulari e microscopia elettronica. Diagnosi molecolare: oggi si possono ottenere risultati rapidi e con un a maggiore sensibilità mediante tecniche di amplificazione del DNA che permettono il rilevamento qualitativo del virus direttamente da campioni di plasma e urine. Mediante l amplificazione del DNA in Real Time è possibile inoltre determinare il titolo virale all interno dell organismo permettendo di monitorare la persistenza dell infezione, ad esempio durante una terapia, e il rischio di complicazioni dell infezione stessa.
65 PCR VANTAGGI: Costi ridotti Amplificazione specifica DNA virale SVANTAGGI: Tempi relativamenti lunghi (4 ore) Limite di risoluzione a 1*10 3 copie
66 RT-PCR CMV + > 1-10 copie/ CMV + < 1-10 copie/ l l CMV - Negative Control VANTAGGI: Tempistica analisi 1,5 ore Alta risoluzione Consente quantificazione virale SVANTAGGI: Tecnica costosa
67 Donor (reporter) Acceptor (quencher) Un alternativa più specifica dei coloranti fluorescenti (SYBER GRENN) sono le sonde sequenza-specifiche marcate con uno o più fluorofori; in questo caso il segnale fluorescente è prodotto solo se nella reazione è presente il bersaglio specifico (amplicone). Le più diffuse sono: 1) Sonde di idrolisi (o sonde Taqman) 2) Molecular beacons 3) Sonde di ibridizzazione 4) Scorpions Le sonde sequenza-specifiche basano la loro funzione sul FRET (Fluorescence Resonance Energy Transfer). Il FRET è un trasferimento di energia distanza-dipendente tra due fluorofori adiacenti che avviene se: - le molecole di colorante fluorescente accettore e donatore sono vicine - lo spettro di assorbimento dell accettore si sovrappone allo spettro di emissione del donatore
68 Zona di sovrapposizione dello spettro di emissione del donatore con lo spettro di assorbimento dell accettore Esistono due tipi principali di FRET: FRET che porta all emissione di fluorescenza (il segnale fluorescente emesso dal donatore (reporter) eccitato è assorbito dal accettore (quencher) che lo riemette sotto forma di segnale fluorescente. Sonde di ibridizzazione. FRET che porta al quenching fluorescente (il segnale fluorescente emesso dal donatore (reporter) eccitato è assorbito dall accettore (quencher) che lo riemette sotto forma di vibrazioni, calore o bassa fluorescenza. Sonde Taqman, molecular beacons e scorpions.
69 SONDE di IBRIDAZIONE Prodotto di amplificazione Oligo 1 Fluoresceina Oligo 2 LC Red 640 Due sonde oligonucleotidiche sequenza-specifiche, marcate con coloranti diversi, ibridizzano con la sequenza bersaglio sul filamento di DNA amplificato in un arrangiamento testa-coda che consente l emissione della fluorescenza. DENATURAZIONE Non viene rilevata fluorescenza: il colorante donatore è eccitato dalla sorgente ed emette energia in grado di eccitare l accettore solo se la distanza fra i due è pari a 1-5 nucleotidi Colorante donatore Colorante accettore Trasferimento Eccitazione Emissione 1-5 nucleotidi ANNEALING Viene registrata la massima fluorescenza L AUMENTO DELLA FLUORESCENZA E REGISTRATO A 640 nm
70 -df/dt Fluorescenza ANALISI di MUTAZIONI PUNTIFORMI Sfrutta differenze nella T m di Sonde di Ibridazione perfettamente legate al DNA bersaglio o il cui legame è destabilizzato dalla presenza di basi non appaiate. Sonda 1 marcata con Fluoresceina Sonda 2 marcata con LC Red 640 gene selvatico omozigote wildtype omozigote mutante eterozigote La Sonda 1 si ibridizza alla parte della sequenza bersaglio mutata La Sonda 2 si ibridizza alla parte della sequenza bersaglio non mutata Temperatura gene mutato Al termine della PCR si analizza la curva di melting: se è presente una mutazione, la T m dell ibrido sarà più bassa che in assenza di mutazione Temperatura
71 MULTIPLEX PCR Consente l analisi simultanea di due campioni nella stessa reazione STRATEGIA: Due coppie di Sonde di Ibridazione, ciascuna specifica per un prodotto di amplificazione, vengono marcate con un diverso colorante accettore che emette la fluorescenza a differenti lunghezze d onda. Fluorecseina LC Red 640 Fluorecseina LC Red 705 Canale nm Canale nm Il segnale emesso viene letto simultaneamente attraverso due canali di rilevamento fluorimetrico. Eccitazione Trasferimento Emissione
72 Sonde Taqman Le sonde di idrolisi o sonde Taqman sono costituite da oligonucleotidi marcati in 5' con un fluoroforo reporter (donatore) e in 3', o internamente, con un quencher (accettore); vengono disegnate in modo che si leghino ad una sequenza specifica del bersaglio compresa tra i primers forward e reverse. Le sonde intatte non emettono fluorescenza perché la vicinanza del quencher al reporter induce una soppressione della fluorescenza del reporter a causa del FRET. Nella fase di estensione dei primers, la sonda Taqman, complementare alla sequenza dell'amplicone, si lega al prodotto PCR e, quando viene raggiunta dalla polimerasi, subisce l'idrolisi da parte dello stesso enzima (attività 5 3 esonucleolitica). A questo punto, il fluoroforo (dye), allontanato dal quencher, se eccitato, emette fluorescenza. No fluorescenza Emissione di fluorescenza
73
74
75 Fattore V Di Laiden Fluorescenza in FAM Fluorescenza in Joe
76
77 Molecular Beacons Sono una variante delle sonde Taqman. Hanno una struttura a forcina con le sequenze dello stelo che sono complementari tra loro e la sequenza dell ansa che è complementare ad una sequenza specifica presente sul DNA bersaglio. Alle estremità i due bracci hanno un fluoroforo e un quencher estremamente vicini. Quando la Molecular Beacon si lega alla sequenza bersaglio lo stem si apre facendo aumentare la distanza tra fluoroforo e quencher; in questo modo non c è più trasferimento di energia dall una all altro e si ha un aumento della fluorescenza. Emissione di fluorescenza
78 Methods of mutation scanning (when we do not know where is our mutation)
79 Methods of mutation scanning (when we do not know where is our mutation) Sequencing (Maxam Gilbert,Sanger, Pyro, NGS); Single-strand conformational analysis; Detecting mismatches or heteroduplex DNA molecules (DGGE, DHPLC); Detecting of deletions (MLPA);
80
81 Single strand conformation polymorphism SSCP E basato sulla differenza di mobilità elettroforetica tra DNA mutato a singolo filamento rispetto a quello non mutato Sostituzione di un solo nucleotide Diversa conformazione 3D di ssdna Diversa mobilità elettroforetica 81
82 Amplificazione del DNA (PCR) Denaturazione DNA amplificato DNA a singolo filamento Elettroforesi su gel di acrilammide colorazione con sali di argento Bande in posizione anomale Norm Omo Etero 82
83 SSCP A: normale B: eterozigote 83
84 . SSCP for detection of point mutations in the LDL receptor gene. We have performed analyses of single- strand conformation polymorphisms (SSCP) of the promoter region and the translated parts of the 18 exons of the low-density Iipoprotein receptor (LDLR) gene. DNA from 20 unrelated familial hypercholesterolemla (FH) patients was studied. Four different running conditions were used for the nondenaturing gel electrophoresis to systematically evaluate how differences in the running conditions affect the sensitivity of the assay.
85
86 Denaturing gradient gel electrophoresis DGGE E basata sul differente comportamento di denaturazione del DNA mutato rispetto a quello normale DNA Presenza di una mutazione Alterazione delle caratteristiche di denaturazione del dsdna Diversa mobilità elettroforetica su gel denaturante 86
87 -La temperatura di denaturazione (Tm, o la concentrazione di denaturante ad essa equivalente) dipende dalla composizione nucleotidica ed è peculiare per ogni frammento di DNA. Frammento di DNA a doppia elica Dominio 1 Tm maggiore Aumento della concentrazione di denaturante Frammento parzialmente denaturato Dominio 2 Tm minore - A basse concentrazioni di denaturante il DNA migra come molecola a doppia elica in funzione delle sue dimensioni - Raggiunta la zona del gel a concentrazione di denaturante uguale al suo Tm, il DNA assume una conformazione ramificata (parziale apertura della doppia elica) e rallenta la sua velocità di migrazione. - La presenza di una mutazione può alterare il Tm del frammento di DNA e di conseguenza la sua mobilità. La variazione nella Tm può essere valutata su gel con gradiente di denaturante. 87
88 DGGE Amplificazione del DNA (PCR) Elettroforesi su gel di acrilammide denaturante gradiente di urea/formamide Il DNA mutato ha caratteristiche di denaturazione differenti rispetto al normale e può essere distinto sulla base della sua diversa mobilità Norm Omo Etero 88
89 Formazione di molecole eteroduplici Viene effettuata per aumentare il potere di risoluzione della DGGE quando la migrazione del DNA mutato è simile a quello del normale - L amplificato viene denaturato e poi sottoposto ad un lento processo di riappaiamento. Queste condizioni favoriscono la formazione di eteroduplici. Mutazione eterozigote Molecole omoduplici Dovute all appaiamento tra i due filamenti normali e tra i due filamenti mutati Molecole eteroduplici Dovute all appaiamento di un filamento normale con uno mutato Normale od omozigote: formazione di un unica molecola omoduplex Eterozigote: formazione di 4 molecole 2 omoduplex (riappaiamento dei 2 filamenti normali e mutati) 2 eteroduplex (formati dalle due combinazioni possibili tra due filamenti normali e due mutati) 89
90 - Gli eteroduplex migrano sempre più lentamente dei corrispondenti omoduplex in quanto il misappaiamento in essi contenuto determina un effetto destabilizzante nella molecola che si aprirà più facilmente qualora sottoposta a denaturazione Urea bassa conc. Urea alta conc AA: normale Aa: eterozigote per la mutazione aa: omozigote 90
91 DGGE A: eterozigote per la mutazione B: normale 91
92
93 HPLC 93
94 RP-HPLC E basato sul principio della ripartizione in fase inversa e separa i Frammenti di DNA sulla base delle loro dimensioni. Colonna C18: fase stazionaria a base polimerica (stirenedivenilbenzene) derivatizzata con catene alchiliche a 18 atomi di carbonio. Ha caratteristiche idrofobiche. Fase mobile: contiene solvente organico (acetonitrile) e un accoppiante ionico (TEAA, trietilammonioacetato). Eluizione: mediante gradiente di acetonitrile. L acetonitrile rompe il legame TEAA-DNA consentendone l eluizione. I frammenti più corti (minor numero di legami DNA-TEAA) vengono eluiti prima rispetto ai frammenti più lunghi che richiedono, per essere eluiti, una maggiore percentuale di acetonitrile nella fase mobile. 94
95 Interazione tra fase stazionaria e DNA La fase stazionaria (C18) è neutra ed idrofobica. L agente accoppiante (TEAA) è una molecola bifunzionale con un estremità idrofobica ed una polare con carica positiva. C18 TEAA Il TEA si lega al DNA (carico negativamente) facendo da ponte e favorendo l interazione DNA-C18. 95
96 Separazione di frammenti di DNA di diverse dimensioni 80 pb 587 pb 96
97 HPLC denaturante (DHPLC) La separazione viene effettuata in condizioni parzialmente denaturanti, cioè termostatando la colonna ad una determinata temperatura (temp di quasi denaturazione). In presenza di una mutazione la denaturazione e l eluizione del frammento sarà diversa rispetto al non mutato. Alla temp di quasi denaturazione gli eteroduplici mostrano una parziale denaturazione in corrispondenza del misappaiamento mentre gli omoduplici, alla stessa temperatura, sono ancora nella forma di doppia elica. Gli eteroduplici hanno un comportamento cromatografico diverso rispetto all omoduplice non mutato e generalmente vengono eluiti più rapidamente. La presenza di una mutazione si evidenzia con la comparsa nel cromatogramma di picchi supplementari rispetto al normale. 97
98 mvolts 12.5 Eteroduplici Omoduplici o C 53 o C 54 o C 55 o C 56 o C 57 o C 58 o C Minuti Cromatogrammi di un campione eterozigote per una mutazione ottenuti a diverse temperature. Si nota la differente risoluzione degli eteroduplici in base alla temperatura. Minutes 98
99 Profili di eluizione in presenza di mutazioni wt wt 99
100 DGGE - CP: controllo positivo - wt: campione normale - C, N, O: campioni eterozigoti per la mutazione DHPLC 100
101
102
103 MLPA Probe design Synthetic oligo M13 derived oligo bp bp 1 A 1 B 2 A 2 B 3 A 3 B 45 A 45 B Primer X Target Specific Stuffer Sequence Primer Y sequence Each probe consists of two parts: one synthetic oligonucleotide and one phage M13 derived single-stranded DNA fragment For each probe there is a target specific sequence that can be ligated when correctly hybridized to its target. All probes have the same PCR primer sequences at their ends. The nonhybridizing stuffer sequence of each probe has a different length and sequence enabling separation by electrophoresis
104 MLPA Reaction #1 First, genomic DNA is completely denatured by heat The MLPA probe mix is added and allowed to hybridize to their respective target overnight
105 MLPA Reaction #2 The hybridized probes are ligated with the thermo-stable Ligase-65
106 MLPA Reaction #3 Only perfectly hybridized probes are ligated
107 MLPA Reaction #4 A universal primer pair is used to amplify all ligated probes Finally the probes are separated by sequence type electrophoresis
108 Separation and quantification by capillary electrophoresis Each peak is the amplification product of a specific probe. Samples are compared to a control sample. A difference in relative peak height or peak area indicates a copy number change of the probe target sequence
109 MLPA and Neuromuscular Disorders Several types of inherited neuromuscular disorder are due to deletions or duplications of specific genes. Among these, Dystrophinopaties (Duchenne Muscular Dystrophy, DMD, and Becker Muscular Dystrophy, BMD), Spinal Muscular Atrophy (SMA), Charcot Marie Thoot (CMT) disease and Hereditary Neuropathy with liability to Pressure Palsies (HNPP) represent a large portion of all mendelian neuromuscular disease for which genetic testing is routinely carried out for diagnostic purposes, for the identification of healthy carriers and for the evaluation of the recurrence risk. For this reason, MLPA assay represents a powerful tool for the study of these different conditions.
110 Current applications on MLPA Detection of aneuploidy of chromosomes 13, 18, 21, X and Y Detection of large chromosomal deletions or duplications: DiGeorge syndrome, Williams syndrome, Spinal Muscular Atrophy (SMA), subtelomeric regions etc Detection of gains and losses of genes in cancer tissues: Her2-neu (ERBB2), TP53, MYC etc Detection of deletions / duplications of single genes and exons: RHD, BRCA1 and 2, MSH2, MLH1, VHL, SHOX, MECP2, APC and NF2 etc Copy number of all CFTR exons Copy number of all 79 DMD exons in 2 MLPA reactions MS-MLPA kits (PWS/AS) Mediterranean Fever Tumor genetics
111
112
113
114 Use of the MLPA Assay in the Molecular Diagnosis of Gene, Copy Number Alterations in Human Genetic DiseasesInt. J. Mol. Sci. 2012, 13, ;
115 DNA Sequencing
116 DNA sequencing Determination of nucleotide sequence Two similar methods: 1. Maxam and Gilbert method 2. Sanger method They depend on the production of a mixture of oligonucleotides labeled either radioactively or fluorescein, with one common end and differing in length by a single nucleotide at the other end This mixture of oligonucleotides is separated by high resolution electrophoresis on polyacrilamide gels and the position of the bands determined
117 Maxam-Gilbert Walter Gilbert Harvard physicist Knew James Watson Became intrigued with the biological side Became a biophysicist Allan Maxam
118 Maxam-Gilbert sequencing - modifications
119 Maxam and Gilbert Method Chemical degradation of purified fragments (chemical degradation) The single stranded DNA fragment to be sequenced is end-labeled by treatment with alkaline phosphatase to remove the 5 phosphate It is then followed by reaction with P-labeled ATP in the presence of polynucleotide kinase, which attaches P labeled to the 5 terminal The labeled DNA fragment is then divided into four aliquots, each of which is treated with a reagent which modifies a specific base 1. Aliquot A + dimethyl sulphate, which methylates guanine residue 2. Aliquot B + formic acid, which modifies adenine and guanine residues 3. Aliquot C + Hydrazine, which modifies thymine + cytosine residues 4. Aliquot D + Hydrazine + 5 mol/l NaCl, which makes the reaction specific for cytosine The four are incubated with piperidine which cleaves the sugar phosphate backbone of DNA next to the residue that has been modified
120 Maxam-Gilbert Technique Principle Chemical Degradation of Pyrimidines Pyrimidines (C, T) are damaged by hydrazine Piperidine cleaves the backbone 2 M NaCl inhibits the reaction with T
121 Maxam-Gilbert sequencing - summary
122 Advantages/disadvantages Maxam-Gilbert sequencing Requires lots of purified DNA, and many intermediate purification steps Relatively short readings Automation not available (sequencers) Remaining use for footprinting (partial protection against DNA modification when proteins bind to specific regions, and that produce holes in the sequence ladder) In contrast, the Sanger sequencing methodology requires little if any DNA purification, no restriction digests, and no labeling of the DNA sequencing template
123 Fred Sanger, 1958 Was originally a protein chemist Made his first mark in sequencing proteins Made his second mark in sequencing RNA 1980 dideoxy sequencing Sanger Method
124 Sanger Method in-vitro DNA synthesis using terminators, use of dideoxinucleotides that do not permit chain elongation after their integration DNA synthesis using deoxy- and dideoxynucleotides that results in termination of synthesis at specific nucleotides Requires a primer, DNA polymerase, a template, a mixture of nucleotides, and detection system Incorporation of di-deoxynucleotides into growing strand terminates synthesis Synthesized strand sizes are determined for each dideoxynucleotide by using gel or capillary electrophoresis Enzymatic methods
125 Dideoxynucleotide PPP O 5 CH2 O BASE 3 no hydroxyl group at 3 end prevents strand extension
126 3 CCGTAC 5 primer 5 3 dntp ddatp ddttp ddctp ddgtp GGCA GGCAT A T C G GGC G GG GGCATG
127 Sanger Method Sequencing Gel
128
129 Dalla cycle sequencing all elettroforetogramma...
130 Le emissioni fluorescenti vengono captate da un rilevatore e le informazioni vengono integrate e trasformate in picchi di colore diverso, con aree proporzionali all intensità di emissione.
131 Sequenziamento automatico Terminatori BigDye Sistemi a trasferimento di energia a singola molecola, costituiti da accettore e donatore legati da un linker Vantaggi: Segnale omogeneo, basso rumore di fondo, luminosità maggiore, facilità interpretativa per A e G attigue D A
132 Mutazione di un singolo nucleotide
133 Mutazioni per inserzione o delezione di nucleotidi in eterozigosi
134 Pyrosequencing
135 Principle of Pyrosequencing Addition of dntps is performed one at a time. thio triphosphate (datp-alphas) is used as a substitute for the natural (datp), as natural datp is not good for luciferase
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Real Time PCR. La PCR Real Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico.
 eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Metodi di analisi mutazionale
 Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PRINCIPALI APPLICAZIONI DELLA PCR. PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b) PRINCIPALI TIPI DI PCR a)
 EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
ABI-7700 User Bulletin #5
 ABI-7700 User Bulletin #5 1. halogen tungsten lamp 2b. emission filters 3. intensifier 5. ccd detector 350,000 pixels 2a. excitation filters 4. sample plate www.biorad.com 3 Real-time qpcr - La fluorescenza
ABI-7700 User Bulletin #5 1. halogen tungsten lamp 2b. emission filters 3. intensifier 5. ccd detector 350,000 pixels 2a. excitation filters 4. sample plate www.biorad.com 3 Real-time qpcr - La fluorescenza
Diagnosi genetiche molecolari
 Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine
 Training Course 2010 Stem Cell Differentiation Napoli, 9-12 Novembre Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine Cristina D
Training Course 2010 Stem Cell Differentiation Napoli, 9-12 Novembre Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine Cristina D
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
La farmacogeneticanella gestione del paziente con tumore colorettale, l esperienza traslazionaleal CRO di Aviano
 SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
Metodica fu ideata nel 1983
 Metodica fu ideata nel 1983 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi
Metodica fu ideata nel 1983 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi
ANALISI DI MUTAZIONI PUNTIFORMI NON NOTE ANALISI DI SEQUENZA.
 ANALISI DI MUTAZIONI PUNTIFORMI NON NOTE Margherita Vinciguerra U.O. Ematologia II Laboratorio per lo Studio e la Diagnosi Molecolare Prenatale di Talassemia A.O. V. Cervello, Palermo. Analisi di sequenza
ANALISI DI MUTAZIONI PUNTIFORMI NON NOTE Margherita Vinciguerra U.O. Ematologia II Laboratorio per lo Studio e la Diagnosi Molecolare Prenatale di Talassemia A.O. V. Cervello, Palermo. Analisi di sequenza
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
PCR. (Reazione a catena della polimerasi) ESTRAZIONE del DNA da gel di AGAROSIO. Corso di INGEGNERIA GENETICA, Prof. Renato Fani,
 PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Protocollo Crime Scene Investigation
 Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Biotecnologie Mediche. Cinzia Di Pietro 0953782055 dipietro@unict.it http://www.bgbunict.it/
 Biotecnologie Mediche Cinzia Di Pietro 0953782055 dipietro@unict.it http://www.bgbunict.it/ Biotecnologie Mediche Tecnologie da applicare per la salute dell uomo Biotecnologie Mediche Prevenzione Diagnostica
Biotecnologie Mediche Cinzia Di Pietro 0953782055 dipietro@unict.it http://www.bgbunict.it/ Biotecnologie Mediche Tecnologie da applicare per la salute dell uomo Biotecnologie Mediche Prevenzione Diagnostica
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di. DNA umano. 22-26 giugno 2009
 Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3. Criteri di identificazione
 Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Tecniche di sequenziamento del DNA
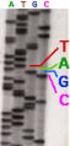 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D
 Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D Spettrofotometro cuvetta monocromatore rivelatore
Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D Spettrofotometro cuvetta monocromatore rivelatore
Genotipizzazione virale
 Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Principali tecniche di base
 Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione (Southern blotting) PCR (Polymerase Chain Reaction) (Sequenziamento) Tecniche di base Enzimi di di restrizione
Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione (Southern blotting) PCR (Polymerase Chain Reaction) (Sequenziamento) Tecniche di base Enzimi di di restrizione
Lezione XLI-XLII martedì 17-1-2012
 Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
8-06-2010. BLOTTING degli acidi nucleici: RT-PCR SOUTHERN BLOTTING NORTHERN BLOTTING. BLOTTING delle proteine: NORTHERN BLOTTING SOUTHERN BLOTTING
 8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
PROGETTO DNA chiavi in mano. Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi
 PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR. Dott. Paolo Cascio
 LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Orga Bio Human S.r.l. DIREZIONE E UFFICI: Via Amsterdam 75, 00144 Roma Tel/fax: 06 99701675
 Orga Bio Human S.r.l. DIREZIONE E UFFICI: Via Amsterdam 75, 00144 Roma Tel/fax: 06 99701675 SEDE LEGALE: Via Helsinki 21, 00144 Roma Tel. 06 97998273; Fax 06 52246148 e-mail: orgabiohuman@fastwebnet.it
Orga Bio Human S.r.l. DIREZIONE E UFFICI: Via Amsterdam 75, 00144 Roma Tel/fax: 06 99701675 SEDE LEGALE: Via Helsinki 21, 00144 Roma Tel. 06 97998273; Fax 06 52246148 e-mail: orgabiohuman@fastwebnet.it
PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay)
 Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
La trasmissione dei caratteri ereditari. Le leggi di Mendel (1882-1884)
 La trasmissione dei caratteri ereditari Le leggi di Mendel (1882-1884) Le leggi di Mendel studiano la trasmissione di caratteri qualitativi prodotti da un singolo gene Procedimento sperimentale di Mendel
La trasmissione dei caratteri ereditari Le leggi di Mendel (1882-1884) Le leggi di Mendel studiano la trasmissione di caratteri qualitativi prodotti da un singolo gene Procedimento sperimentale di Mendel
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo
 C.R.A. Consiglio per la Ricerca in Agricoltura Centro di ricerca per la genomica Fiorenzuola d Arda (Piacenza) SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo Tutor: Dott. Gianni TACCONI Studente:
C.R.A. Consiglio per la Ricerca in Agricoltura Centro di ricerca per la genomica Fiorenzuola d Arda (Piacenza) SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo Tutor: Dott. Gianni TACCONI Studente:
Maria Antonietta Lepore. Principali tecniche di biologia molecolare clinica
 Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Elettroforesi. Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita.
 Elettroforesi Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita. A qualunque ph diverso dal pi le proteine hanno una carica netta quindi,
Elettroforesi Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita. A qualunque ph diverso dal pi le proteine hanno una carica netta quindi,
Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici
 "Focus su sicurezza d'uso e nutrizionale degli alimenti" 21-22 Novembre 2005 Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici Simona
"Focus su sicurezza d'uso e nutrizionale degli alimenti" 21-22 Novembre 2005 Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici Simona
Progetto della classe II C
 Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
METODI ALTERNATIVI. PCR digitale per la rilevazione e quantificazione. assoluta del DNA
 METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
PCR (Polymerase Chain Reaction)
 PCR (Polymerase Chain Reaction) Metodo enzimatico estremamente rapido e semplice per produrre una quantità illimitata di copie della sequenza di un singolo gene Sometime a good idea comes to yow when you
PCR (Polymerase Chain Reaction) Metodo enzimatico estremamente rapido e semplice per produrre una quantità illimitata di copie della sequenza di un singolo gene Sometime a good idea comes to yow when you
PRODA Istituto di Diagnostica Clinica
 Test genetici per evidenziare il rischio di trombolfilia Il Fattore V della coagulazione è un cofattore essenziale per l attivazione della protrombina a trombina. La variante G1691A, definita variante
Test genetici per evidenziare il rischio di trombolfilia Il Fattore V della coagulazione è un cofattore essenziale per l attivazione della protrombina a trombina. La variante G1691A, definita variante
Quantitative Real-time PCR
 Quantitative Real-time PCR Tecnica che consente la simultanea amplificazione e quantificazione del DNA target AMPLIFICAZIONE: prevede essenzialmente gli step di una classica PCR QUANTIFICAZIONE: effettuata
Quantitative Real-time PCR Tecnica che consente la simultanea amplificazione e quantificazione del DNA target AMPLIFICAZIONE: prevede essenzialmente gli step di una classica PCR QUANTIFICAZIONE: effettuata
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
Note Tecniche al LightCycler Selezione di sonde di ibridazione per LightCycler. Olfert Landt e Andreas Nitsche, TIB MOLBIOL, Berlino
 Note Tecniche al LightCycler Selezione di sonde di ibridazione per LightCycler Olfert Landt e Andreas Nitsche, TIB MOLBIOL, Berlino 1. Introduzione Scopo di questa nota La detezione di amplicon specifici
Note Tecniche al LightCycler Selezione di sonde di ibridazione per LightCycler Olfert Landt e Andreas Nitsche, TIB MOLBIOL, Berlino 1. Introduzione Scopo di questa nota La detezione di amplicon specifici
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona
 Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
SINTESI DELL RNA. Replicazione. Trascrizione. Traduzione
 SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
Quantificazione di. legionella pneumophila. mediante metodo biomolecolare
 Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
Principi dello screening raccomandati dalla World Health Organization
 EMOGLOBINE: DIAGNOSTICA, STANDARDIZZAZIONE, PROSPETTIVE Approcci diagnostici e problematiche in relazione ad uno screening neonatale Giovanni Ivaldi Laboratorio di Genetica - Settore Microcitemia Ospedali
EMOGLOBINE: DIAGNOSTICA, STANDARDIZZAZIONE, PROSPETTIVE Approcci diagnostici e problematiche in relazione ad uno screening neonatale Giovanni Ivaldi Laboratorio di Genetica - Settore Microcitemia Ospedali
Variazioni del genoma possono determinare:
 Variazioni del genoma possono determinare: Insorgenza di malattie genetiche Differente predisposizione a determinate patologie Differente risposta a specifiche terapie Differente risposta a stress ambientali,
Variazioni del genoma possono determinare: Insorgenza di malattie genetiche Differente predisposizione a determinate patologie Differente risposta a specifiche terapie Differente risposta a stress ambientali,
REPLICAZIONE DEL DNA
 REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
ZytoLight SPEC ERG Dual Color Break Apart Probe
 ZytoLight SPEC ERG Dual Color Break Apart Probe IVD Dispositivo medico-diagnostico in vitro Produttore: ZytoVision GmbH Codice: Z-2138-200 (0.2ml).. Per l identificazione della traslocazione che coinvolge
ZytoLight SPEC ERG Dual Color Break Apart Probe IVD Dispositivo medico-diagnostico in vitro Produttore: ZytoVision GmbH Codice: Z-2138-200 (0.2ml).. Per l identificazione della traslocazione che coinvolge
Metodiche analitiche. Anna Maria Eleuteri
 Metodiche analitiche per la rilevazione i di mutazioni i Anna Maria Eleuteri Analisi del DNA dopo PCR Analisi dei prodotti di amplificazione - Elettroforesi su gel di agarosio Metodi per lo studio di mutazioni
Metodiche analitiche per la rilevazione i di mutazioni i Anna Maria Eleuteri Analisi del DNA dopo PCR Analisi dei prodotti di amplificazione - Elettroforesi su gel di agarosio Metodi per lo studio di mutazioni
Verifiche qualitative dell RNA e cdna in ambito dei trials BCR/ABL e AML translocation programme. Massimo Degan, CRO Aviano (PN)
 e cdna in ambito dei trials BCR/ABL e AML translocation programme Massimo Degan, CRO Aviano (PN) perchè è importante verificare l RNA La verifica della qualità dell RNA nei saggi diagnostico-molecolari
e cdna in ambito dei trials BCR/ABL e AML translocation programme Massimo Degan, CRO Aviano (PN) perchè è importante verificare l RNA La verifica della qualità dell RNA nei saggi diagnostico-molecolari
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE
 LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
ENZIMI DI RESTRIZIONE
 ENZIMI DI RESTRIZIONE La scoperta degli enzimi di restrizione e modificazione Intorno agli anni 50 si notò che talvolta l introduzione in E.coli di DNA esogeno, proveniente da un diverso ceppo di E.coli,
ENZIMI DI RESTRIZIONE La scoperta degli enzimi di restrizione e modificazione Intorno agli anni 50 si notò che talvolta l introduzione in E.coli di DNA esogeno, proveniente da un diverso ceppo di E.coli,
Thalassaemias. Thalassaemia Hb Variants. A group of blood deseases caused by a genetically determined. alteration in globin synthesis
 Approccio diagnostico delle Varianti Hb Giovanni Ivaldi Laboratorio di Genetica Umana - Settore Microcitemia Ospedali Galliera - Genova A group of blood deseases caused by a genetically determined reduction
Approccio diagnostico delle Varianti Hb Giovanni Ivaldi Laboratorio di Genetica Umana - Settore Microcitemia Ospedali Galliera - Genova A group of blood deseases caused by a genetically determined reduction
GENETICA seconda parte
 GENETICA seconda parte I cromosomi sono lunghe molecole di una sostanza l acido desossiribonucleico. DNA Il DNA è una lunga catena fatta da due lunghi fili avvolti su se stessi a doppia elica. Sembra una
GENETICA seconda parte I cromosomi sono lunghe molecole di una sostanza l acido desossiribonucleico. DNA Il DNA è una lunga catena fatta da due lunghi fili avvolti su se stessi a doppia elica. Sembra una
Aggiornamenti in ambito genetico
 10 Congresso Nazionale sulla Sindrome di Cornelia de Lange 1-4 novembre 2012 - Gabicce Aggiornamenti in ambito genetico Cristina Gervasini Genetica Medica Dip. Scienze della Salute Università degli Studi
10 Congresso Nazionale sulla Sindrome di Cornelia de Lange 1-4 novembre 2012 - Gabicce Aggiornamenti in ambito genetico Cristina Gervasini Genetica Medica Dip. Scienze della Salute Università degli Studi
Alberto Viale I CROMOSOMI
 Alberto Viale I CROMOSOMI DA MENDEL ALLA GENETICA AL DNA ALLE MUTAZIONI I cromosomi sono dei particolari bastoncelli colorati situati nel nucleo delle cellule. Sono presenti nelle cellule di ogni organismo
Alberto Viale I CROMOSOMI DA MENDEL ALLA GENETICA AL DNA ALLE MUTAZIONI I cromosomi sono dei particolari bastoncelli colorati situati nel nucleo delle cellule. Sono presenti nelle cellule di ogni organismo
Lezioni di biotecnologie
 Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
U.O.C. di Epatologia Clinica e Biomolecolare. Unità di misura. Repertorio. 200 ml 10.000 U. 500 test
 U.O.C. di Epatologia Clinica e Biomolecolare Lotto N. DESCRIZIONE PRODOTTO Quantità annua richiesta Unità di misura Repertorio CND Codice Prodotto, Confezione Offerta e nome commerciale Prezzo unitario
U.O.C. di Epatologia Clinica e Biomolecolare Lotto N. DESCRIZIONE PRODOTTO Quantità annua richiesta Unità di misura Repertorio CND Codice Prodotto, Confezione Offerta e nome commerciale Prezzo unitario
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Immagine di una sequenza ben riuscita. Sequences troubleshooting
 Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Controllo ufficiale degli OGM nel settore agroalimentare
 Controllo ufficiale degli OGM nel settore agroalimentare Ugo Marchesi Istituto Zooprofilattico Sperimentale Lazio e Toscana Centro di Referenza Nazionale per la ricerca di OGM ugo.marchesi@izslt.it ORGANISMI
Controllo ufficiale degli OGM nel settore agroalimentare Ugo Marchesi Istituto Zooprofilattico Sperimentale Lazio e Toscana Centro di Referenza Nazionale per la ricerca di OGM ugo.marchesi@izslt.it ORGANISMI
Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta)
 Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta) E il termine generico che indica una serie di tecniche di separazione di molecole simili in
Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta) E il termine generico che indica una serie di tecniche di separazione di molecole simili in
PRODA Istituto di Diagnostica Clinica
 PRODA Istituto di Diagnostica Clinica Sezione di Citogenetica e Genetica molecolare Responsabile: Dott. Guglielmo Sabbadini Specialista in Genetica Medica Informazioni per la diagnosi molecolare di sordita
PRODA Istituto di Diagnostica Clinica Sezione di Citogenetica e Genetica molecolare Responsabile: Dott. Guglielmo Sabbadini Specialista in Genetica Medica Informazioni per la diagnosi molecolare di sordita
La mutazione è una modificazione della sequenza delle basi del DNA
 La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
Il metabolismo dell RNA. Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
 Il metabolismo dell RNA I vari tipi di RNA Il filamento di DNA che dirige la sintesi dello mrna è chiamato filamento stampo o filamento antisenso. L altro filamento che ha sequenza identica a quella dello
Il metabolismo dell RNA I vari tipi di RNA Il filamento di DNA che dirige la sintesi dello mrna è chiamato filamento stampo o filamento antisenso. L altro filamento che ha sequenza identica a quella dello
LA GENETICA: DNA e RNA LA GENETICA. DNA e RNA. Prof. Daniele Verri
 LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
Figura 1. Rappresentazione della doppia elica di DNA e struttura delle differenti basi.
 Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
Strategie di purificazione di proteine
 Laurea Magistrale in Scienze e Biotecnologie degli Alimenti Strategie di purificazione di proteine Lezione n.xx-2-23 PRINCIPIO - ALIMENTI DA MATRICI SOLIDE E LIQUIDE MATRICI SOLIDE - ROMPERE LA STRUTTURA
Laurea Magistrale in Scienze e Biotecnologie degli Alimenti Strategie di purificazione di proteine Lezione n.xx-2-23 PRINCIPIO - ALIMENTI DA MATRICI SOLIDE E LIQUIDE MATRICI SOLIDE - ROMPERE LA STRUTTURA
Macromolecole Biologiche. I domini (III)
 I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
ANALISI GENETICHE. Biologia molecolare
 ANALISI GENETICHE Biologia molecolare Via F. Ferrer 25/27-21052 Busto Arsizio (VA) - Italy Tel +39 0331 652911 - Fax +39 0331 652919 - www.tomalab.com TOMA_biologia_molecolare_scheda_CORR03.indd 1 06/04/2012
ANALISI GENETICHE Biologia molecolare Via F. Ferrer 25/27-21052 Busto Arsizio (VA) - Italy Tel +39 0331 652911 - Fax +39 0331 652919 - www.tomalab.com TOMA_biologia_molecolare_scheda_CORR03.indd 1 06/04/2012
Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica. Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA.
 Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA. In genere si ottengono trattando il DNA con agenti chimici (es.
Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA. In genere si ottengono trattando il DNA con agenti chimici (es.
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #6 Dr. Marco Galardini AA 2012/2013 Gene regulation analysis Lezione #6 Dr. Marco Galardini AA 2012/2013 Regolazione genica Elementi molecolari e
Applicazioni biotecnologiche in systems biology Lezione #6 Dr. Marco Galardini AA 2012/2013 Gene regulation analysis Lezione #6 Dr. Marco Galardini AA 2012/2013 Regolazione genica Elementi molecolari e
