Analisi di sequenziamento degli acidi nucleici
|
|
|
- Dino Negro
- 8 anni fa
- Visualizzazioni
Transcript
1 Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati con apparecchiature automatiche. Il sequenziamento del DNA viene sempre più usato nella diagnostica medica perché molti laboratori, anche di piccole dimensioni, cominciano ad essere dotati delle apparecchiature necessarie. Sequenziare un frammento di DNA, per esempio per la identificazione di mutazioni del genoma, è diventato relativamente facile e veloce, senza dubbio più veloce che sequenziare una proteina, cioè determinare la sequenza amminoacidica di una proteina. A tal punto che, come sapete, la determinazione della sequenza dell intero genoma umano è già stata portata a compimento. Sequenziare un frammento di DNA significa stabilire la successione dei nucleotidi che ne formano la molecola: ACGGGGGGTTTAGCGCGATTCCAT Il sequenziamento del DNA viene attualmente effettuato con un metodo descritto già alla metà degli anni settanta da un certo Sanger. Sono stati abbandonati i metodi chimici di Maxam e Gilbert. Oggi in molte applicazioni si effettua il sequenziamento diretto: che cosa vuol dire? Fino a non molto tempo fa una sequenza di DNA prevedeva il clonaggio del frammento da sequenziare, una procedura che richiedeva e richiede l inserimento del frammento in un vettore, ad esempio un plasmide, che viene poi inserito in un batterio per poterlo replicare. Oggi è possibile evitare il clonaggio del frammento da sequenziale grazie alla possibilità di amplificare il DNA bersaglio con l uso della PCR (clonaggio a-cellulare). Il metodo di Sanger si basa sulla metodica di terminazione della catena coi di-deossi nucleotidi. E attualmente il metodo più usato per definire mutazioni specifiche. I vantaggi principali di questo metodo risiedono nella facilità d uso, e questa è stata aumentata negli anni recenti con l introduzione di marcature fluorescenti e sistemi automatizzati che hanno ovviato alla necessità di sostanze radioattive. Anche se il prezzo iniziale delle apparecchiature è elevato esso è compensato da una chimica single tube (in una sola provetta) e da una rapida analisi e definizione delle basi (base calling). Il vantaggio principale di sequenziare direttamente il DNA usando la PCR anziché sequenziare col clonaggio è che solo una sequenza deve essere determinata. I dati ottenuti rappresentano una media della sequenza dei DNA bersaglio in soluzione. Alternativamente, quando vengono sequenziati cloni di PCR un minimo di 4 o 5 differenti set di dati devono essere determinati.
2 Il principio su cui si basa il sequenziamento del DNA consiste nel blocco della reazione di estensione della catena DNA mediante i cosiddetti analoghi delle basi. Queste sono molecole molto simili a deossiribonucleotidi ma mancano dell ossidrile nel carbonio 3 (di-deossiribonucleotidi) per cui possono essere incorporati in una catena di DNA in fase di sintesi ma ad essi non possono essere legati altri dntp. Quindi se un di-deossi viene per caso incorporato nella catena nascente di DNA l ulteriore allungamento viene bloccato e la reazione termina. Quindi i di-deossi sono chiamati anche terminatori di catena. In pratica vengono allestite 4 reazioni in parallelo, ciascuna contenente una delle 4 basi sotto forma di di-deossi in concentrazione molto debole. Il frammento di DNA da sequenziare (stampo) viene incubato con una DNA polimerasi termostabile in presenza di desossiribonucleotide fosfati (dntps) e di un primer oligonucleotidico, complementare alla parte iniziale del DNA da sequenziare. tgcacacgcgcgctatatag... ACGTGTGCGCGCGATATATCGCTATATGACGGATCGGCGCTATAGC La polimerasi a partire dal primer genera un filamento complementare allo stampo, che si estende per una lunghezza indefinita a valle del primer. Ciò dà luogo a una amplificazione lineare dei prodotti di estensione.
3 Siccome la sintesi del DNA viene effettuata per esterificazione dell ossidrile 3 di un nucleotide, già incorporato mediante l estremità 5 fosfato, sul carbonio in alfa di un deossi nel momento in cui viene incorporato un di-deossi la sintesi si arresta (poiché non possiede un 3 ossidrile). Dal momento che questo nucleotide è presente in una concentrazione molto bassa esso verrà incorporato molto raramente e in modo casuale. Statisticamente si otterranno così tanti frammenti abortivi quante sono le volte in cui le basi corrispondenti sono rappresentate nel pezzo di DNA in questione. La dimensione dei frammenti sintetizzati corrisponde alla distanza tra l inizio del primer e la base a livello della quale si è fermata la replicazione. Il principio statistico è lo stesso utilizzato anche nei metodi chimici. La dimensione dei frammenti viene stimata attraverso un autoradiografia, che consiste in un elettroforesi del DNA marcato con radioisotopi o con molecole fluorescenti. In generale il metodo di sequenziamento più usato è il cosiddetto cicle sequencing cioè il metodo di sequenziamento ciclico basato sulla PCR. Si può utilizzare solo quando la zona di cui si vuole determinare la sequenza è conosciuta. La sua principale applicazione è la evidenziazione rapida della natura di una mutazione puntiforme. Questo lavoro, che normalmente necessita di parecchi mesi, può ora essere realizzato in qualche giorno. Si basa sull uso di una DNA polimerasi termostabile come quella descritta. La sequenza da analizzare è dapprima amplificata direttamente a a partire da un po di DNA gnomico mediante la tecnica del sequenziamento a doppia elica, utilizzando dei primer interni al segmento da amplificare. Uno degli oligonucleotidi è utilizzato in un primo tempo per determinare la sequenza della prima elica. Questa sequenza viene poi confermata mediante la determinazione dell elica complementare, fatta utilizzando un secondo oligonucleotide. Più gli oligo sono scelti vicino alla mutazione da cercare più la realizzazione sarà facile ed il risultato preciso. In una provetta si introducono il DNA campione, il cosiddetto templato, la DNA polimerasi i dntp e un primer di sequenza che funge da innesco per la polimerasi. Sono presenti i cosiddetti terminatori che essendo strutturalmente analoghi ai dntp vengono incorporati dalla polimerasi ma non possono a loro volta essere allungati. Il tutto viene introdotto in un termal cycler e sottoposto a oscillazioni termiche a cicli ripetuti. Il risultato è un amplificazione lineare dei prodotti di estensione. E un metodo che ha molti vantaggi tra cui l uso di piccole quantità di DNA campione (meno di 1 microgrammo). La natura ciclica dà origine ad un segnale forte e riduce la possibilità di inneschi parassiti (random priming) che producono rumore di fondo. L uso di una DNA polimerasi termostabile consente di sequenziale a 60 C e di ridurre le strutture secondarie nel templato dando più efficienti letture nelle regioni ricche in GC. Uno degli svantaggi della Taq è che l efficienza di incorporazione dei terminatori dipende strettamente dalla sequenza e
4 questo dà origine a profili di segnale molto irregolari. Se questo è un problema si può prendere in considerazione l uso di una polimerasi non termostabile come la Sequenase. Questo enzima dà incorporazioni molto più regolari e profili di segnale più uniformi ma non può essere usata a 60 C e quindi problemi di struttura secondaria danno origine a una porzione di sequenza illeggibile in gran parte delle sequenze. Disegno dei primer di sequenza Regole generali Esistono dei software che possono essere scaricati da siti web. Sono importanti la composizione in basi, l eventuale formazione di strutture secondarie, la stabilità. 1. La lunghezza del primer deve essere sufficiente da avere una elevata probabilità di formare un ibrido unico col templato. La temperatura di fusione del primer deve consentire la formazione di una quota significativa di ibridi stabili nelle condizioni di amplificazione prescelte. 2. Lunghezza ideale (minimo 17) % GC 4. Primer troppo ricchi in AT in genere formano ibridi scarsamente stabili 5. La sequenza del primer deve assomigliare il più possibile ad una successione random delle 4 basi evitare regioni omopolimeriche, specialmente al 3 6. Confrontare il primer candidato con sequenze ripetitive note (p.e. Alu) per evitare alineamenti multipli. 7. Evitare sequenze simmetriche che possono dare autocomplementarietà (problema nei primer ricchi in GC che esaltano ogni imperfezione) 8. Fare attenzione al nucleotide al 3. L esperienza insegna di scegliere come base terminale G e come penultima base se possibile una pirimidina C o T 9. Il primer deve essere disegnato da regioni conservate della sequenza, non deve contenere polimorfismi, inserzioni, delezioni Per decenni la marcatura utilizzata è stata quella con radioisotopi, fosforo 32 o fosforo 33, oppure per le sequenze zolfo 35 che ha una vita media più lunga e dà delle bande più definite all autoradiografia. I sequenziatori automatici sono basati invece sulla marcatura con sostanze fluorescenti. I radioisotopi sono stati il metodo di elezione per la marcatura delle macromolecole. Tuttavia restrizioni crescenti all uso dei radioisotopi insieme alla richiesta sempre maggiore di
5 velocità e sensibilità hanno shiftato verso tecnologie alternative di marcatura come la fluorescenza o la chemiluminescenza. Fluorocromi: sono molecole che possono assorbire energia, vengono eccitate e successivamente decadono ad un livello di energia più basso emettendo una parte dell energia assorbita nel processo, di solito sotto forma di luce visibile o a lunghezza d onda ancora maggiore. Per esempio se una molecola fluorescente viene illuminata da luce blu può emettere una luce verde. Questa tendenza recente ad esplorare e sfruttare le possibilità della fluorescenza sono dovute ad una combinazione di recenti sviluppi: sintesi di nuove molecole fluorescenti (fluorocromi) che possono più facilmente essere legate in modo covalente a macromolecole (DNA!), strumentazione migliore, uso di laser che consentono livelli di energia luminosa più alti e metodi migliori per la rivelazione e l analisi del segnale fluorescente. Le applicazioni della fluorescenza vanno dal sequenziamento del DNA all analisi dei cromosomi allo studio delle membrane cellulari, del trasporto ionico, dell apoptosi e della cinetica enzimatica, della struttura cellulare (microscopia confocale in fluorescenza). Negli ultimi 10 anni la fluorescenza ha sostituito la marcatura radioattiva soprattutto in quei campi dove è richiesto il processamento di un gran numero di dati. Per esempio nei laboratori del Progetto Genoma Umano cha ha determinati i circa geni che compongono il genoma umano non avrebbe senza il sequenziamento in cui il vantaggio principale è l automazione che risulta in un processamento più veloce ed efficiente. Il sequenziamento diretto di prodotti di PCR usando il metodo della terminazione della catena con i dideossi sviluppato da Sanger è attualmente il metodo più usato per definire mutazioni specifiche. Benefici principali: facilità di uso ed è stata aumentata recentemente con l introduzione di marcatura fluorescente e sistemi automatizzati che ovviano alla radioattività. Invece di frammenti di DNA marcati con radioisotopi su pellicola autoradiografica il DNA marcato in fluorescenza usando singole molecole può essere separato con elettroforesi. I dati sono raccolti in linea mediante strumentazione automatizzata. Un progresso recente in questo campo è lo sviluppo di marcature basate sul principio della fluorescenza a trasferimento di energia di fluorescenza tra due molecole. Le molecole (oligo) sono state attaccate due molecole fluorescenti. La prima detta molecola donatrice viene scelta per la capacità di trasferire in maniera efficiente energia elettromagnetica alla seconda molecola (accettrice). Questa molecola accettrice emette la luce assorbita come fluorescenza alla lunghezza d onda che le è propria e adatta agli strumenti di rivelazione. In questo modo l intensità effettiva è da 6 a 12 volte maggiore della sola prima molecola.
6 Un altra strategia è quella di marcare i terminatori. Questa variazione presenta tre vantaggi. Basta una sola incubazione invece di 4, perché il colore dipende solo dal dideossi incorporato. Per la stessa ragione gli arresti casuali delle polimerasi (falsi stop) che rendono spesso illeggibili le sequenze con tecniche classiche, sono senza effetto sulla lettura. Infine gli oligo che servono da primer non devono essere marcati, cosa che permette un economia importante. Un miglioramento radicale è stata la sostituzione del gel di poliacrilammide verticali per la separazione elettroforetica con capillari. Altro miglioramento è stato l introduzione di sistemi multicapillari che consentono la separazione elettroforetica di più prodotti di reazione in parallelo. Con i capillari si eliminano le lastre di gel e quindi il caricamento manuale, uno degli aspetti più noiosi della procedura. Invece di pipettare manualmente, il caricamento del campione viene effettuato con una iniezione elettrocinetica direttamente da una piastra da microtitolazione, il gel viene sostituito automaticamente e non ci sono vetri da lavare. L oligo viene marcato all estremità 5 con un fluorocromo di colore differente. A ciascuna di queste frazioni viene quindi aggiunto uno dei 4 dideossi trifosfati e viene condotta un incubazione di determinazione della sequenza. Tutti i frammenti la cui replicazione viene fermata dall incorporazione di un dato dideossi saranno dunque marcati con lo stesso colore, per esempio blu per le C, rosso per le T, arancio per le G e verde per le A. Le quattro miscele di reazione vengono quindi mescolate. Ciascuna banda osservata avrà il colore dell oligo che è servito da primer per la sintesi del frammento corrispondente. Il sequenziatore non è altro che una camera per l elettroforesi verticale di un gel di poliacrilammide. Ciò consente una risoluzione di una base dei prodotti di sequenza. I frammenti marcati con fluorocromi passano attraverso una finestra di lettura, 24 cm dai pozzetti di caricamento, che è scannerizzata da un laser e sono rivelati da filtri girevoli e fotomoltiplicatori. I segnali in fluorescenza sono inviati ad un computer che analizza la loro posizione e intensità produce un cromatogramma consistente di picchi colorati. L area sottesa al picco rappresenta l intensità del segnale e il colore del picco è specifico per la base che si trova in quella posizione. Il software attribuisce una base A, T, C o G a ciascuna posizione o assegna N se la posizione è ambigua. Sistemi di marcatura Marcatura dei primer In questo caso il fluorocromo deve essere attaccato al primer di sequenza. La marcatura viene introdotta al momento della sintesi dell oligo. Esistono dei kit commerciali che consentono di
7 farsi in casa la marcatura. Funzionano abbastanza bene però bisogna purificare il primer per togliere l eccesso di marcatura non legata. Occorrono 4 reazioni di sequenza separate, ognuna contenente un diverso terminatore. I prodotti sono poi mescolati tra loro ed estratti per eliminare l eccesso di primer marcati prima dell elettroforesi. Vantaggi: l intensità del segnale è indipendente dalla sequenza e quindi il profilo del segnale è più uniforme e non si vedono anamlie delle A delle T o problemi di rumore di fondo Maggiore lunghezza delle letture. Svantaggi: maggiore costo (4 marcature) Marcatura dei terminatori Vantaggi: si può usare qualsiasi tipo di primer 4 reazioni di sequenza in una sola provetta: meno pipettate Ideale quando si devono fare sequenze ripetute nella stessa regione Meno falsi stop dovuti al distacco prematuro della DNA polimerasi (non c è marcatura) Svantaggi: profilo del segnale poco uniforme Minore lunghezza delle letture I fluorocromi sono attaccati alle molecole dei dideossi terminatori e così il sequencing può essere ottenuto in una singola provetta Preparazione del campione Il campione di DNA da sequenziale deve essere perfetto. La resa e la purezza dell amplificato sono di importanza critica per ottenere sequenze di buona qualità. Cinque microlitri di amplificato vanno fat correre in agaros al 2% per stimare la concentrazione e la purezza. Se si vedono prodotti aspecifici la reazione di amplificazione deve essere ripetuta in condizioni più stringenti, in presenza di DMSO, etc. Amplificare il DNA mediante PCR e farlo correre in agarosio al 2% contenete bromuro di etidio Visualizzare il gel sul transilluminatore e ritagliare la banda corretta con un bisturi o un ago sterile, eluire overnigth la banda in 50 microlitri di acqua sterile deionizzata in una provetta da centrifuga. Usare 5 microlitri della soluzione quale temprato di una seconda PCR con gli stessi primer. Chekkare una aliquota del secondo prodotto di PCR per concentrazione e purezza oppure purificare il prodotto di PCR con colonnine da ultrafiltrazione tipo centricon che sono molto efficaci.
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Immagine di una sequenza ben riuscita. Sequences troubleshooting
 Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR. (Reazione a catena della polimerasi) ESTRAZIONE del DNA da gel di AGAROSIO. Corso di INGEGNERIA GENETICA, Prof. Renato Fani,
 PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Protocollo Crime Scene Investigation
 Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
8-06-2010. BLOTTING degli acidi nucleici: RT-PCR SOUTHERN BLOTTING NORTHERN BLOTTING. BLOTTING delle proteine: NORTHERN BLOTTING SOUTHERN BLOTTING
 8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16
 Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16 L'immunoistochimica e' una tecnica ampiamente utilizzata per l'identificazione e la localizzazione di costituenti cellulari
Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16 L'immunoistochimica e' una tecnica ampiamente utilizzata per l'identificazione e la localizzazione di costituenti cellulari
Il flusso dell informazione genetica. DNA -->RNA-->Proteine
 Il flusso dell informazione genetica DNA -->RNA-->Proteine Abbiamo visto i principali esperimenti che hanno dimostrato che il DNA è la molecola depositaria dell informazione genetica nella maggior parte
Il flusso dell informazione genetica DNA -->RNA-->Proteine Abbiamo visto i principali esperimenti che hanno dimostrato che il DNA è la molecola depositaria dell informazione genetica nella maggior parte
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Maria Antonietta Lepore. Principali tecniche di biologia molecolare clinica
 Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Elementi di Psicometria con Laboratorio di SPSS 1
 Elementi di Psicometria con Laboratorio di SPSS 1 29-Analisi della potenza statistica vers. 1.0 (12 dicembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
Elementi di Psicometria con Laboratorio di SPSS 1 29-Analisi della potenza statistica vers. 1.0 (12 dicembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
EMISSIONE E ASSORBIMENTO DI LUCE DA PARTE DELLA MATERIA
 EMISSIONE E ASSORBIMENTO DI LUCE DA PARTE DELLA MATERIA Poiché la luce è energia trasportata da oscillazioni del campo elettrico (fotoni) e la materia è fatta di particelle elettricamente cariche (atomi
EMISSIONE E ASSORBIMENTO DI LUCE DA PARTE DELLA MATERIA Poiché la luce è energia trasportata da oscillazioni del campo elettrico (fotoni) e la materia è fatta di particelle elettricamente cariche (atomi
DNA - RNA. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi.
 DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di. DNA umano. 22-26 giugno 2009
 Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Progetto della classe II C
 Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Capitolo 2 Caratteristiche delle sorgenti luminose In questo capitolo sono descritte alcune grandezze utili per caratterizzare le sorgenti luminose.
 Capitolo 2 Caratteristiche delle sorgenti luminose In questo capitolo sono descritte alcune grandezze utili per caratterizzare le sorgenti luminose. 2.1 Spettro di emissione Lo spettro di emissione di
Capitolo 2 Caratteristiche delle sorgenti luminose In questo capitolo sono descritte alcune grandezze utili per caratterizzare le sorgenti luminose. 2.1 Spettro di emissione Lo spettro di emissione di
PROGETTO DNA CHIAVI IN MANO
 PROGETTO DNA CHIAVI IN MANO La collaborazione con il Virgilio e il progetto dell IFOM Il progetto DNA chiavi in mano è un percorso pensato dal Centro di Ricerca internazionale IFOM per avvicinare i ragazzi
PROGETTO DNA CHIAVI IN MANO La collaborazione con il Virgilio e il progetto dell IFOM Il progetto DNA chiavi in mano è un percorso pensato dal Centro di Ricerca internazionale IFOM per avvicinare i ragazzi
Figura 1. Rappresentazione della doppia elica di DNA e struttura delle differenti basi.
 Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
METODI DI MARCATURA DEGLI ACIDI NUCLEICI
 METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
Introduzione all analisi dei segnali digitali.
 Introduzione all analisi dei segnali digitali. Lezioni per il corso di Laboratorio di Fisica IV Isidoro Ferrante A.A. 2001/2002 1 Segnali analogici Si dice segnale la variazione di una qualsiasi grandezza
Introduzione all analisi dei segnali digitali. Lezioni per il corso di Laboratorio di Fisica IV Isidoro Ferrante A.A. 2001/2002 1 Segnali analogici Si dice segnale la variazione di una qualsiasi grandezza
Metodi di analisi mutazionale
 Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
I COLORI DEL CIELO: COME SI FORMANO LE IMMAGINI ASTRONOMICHE
 I COLORI DEL CIELO: COME SI FORMANO LE IMMAGINI ASTRONOMICHE Nell ultima notte di osservazione abbiamo visto bellissime immagini della Galassia, delle sue stelle e delle nubi di gas che la compongono.
I COLORI DEL CIELO: COME SI FORMANO LE IMMAGINI ASTRONOMICHE Nell ultima notte di osservazione abbiamo visto bellissime immagini della Galassia, delle sue stelle e delle nubi di gas che la compongono.
Strutturazione logica dei dati: i file
 Strutturazione logica dei dati: i file Informazioni più complesse possono essere composte a partire da informazioni elementari Esempio di una banca: supponiamo di voler mantenere all'interno di un computer
Strutturazione logica dei dati: i file Informazioni più complesse possono essere composte a partire da informazioni elementari Esempio di una banca: supponiamo di voler mantenere all'interno di un computer
Prof.ssa Gamba Sabrina. Lezione 7: IL DNA. Duplicazione e sintesi delle proteine
 Prof.ssa Gamba Sabrina Lezione 7: IL DNA Duplicazione e sintesi delle proteine concetti chiave della lezione Costituzione fisico-chimica del DNA Basi azotate Duplicazione Concetto di geni Rna Trascrizione
Prof.ssa Gamba Sabrina Lezione 7: IL DNA Duplicazione e sintesi delle proteine concetti chiave della lezione Costituzione fisico-chimica del DNA Basi azotate Duplicazione Concetto di geni Rna Trascrizione
I TUTORI. I tutori vanno creati la prima volta seguendo esclusivamente le procedure sotto descritte.
 I TUTORI Indice Del Manuale 1 - Introduzione al Manuale Operativo 2 - Area Tutore o Area Studente? 3 - Come creare tutti insieme i Tutori per ogni alunno? 3.1 - Come creare il secondo tutore per ogni alunno?
I TUTORI Indice Del Manuale 1 - Introduzione al Manuale Operativo 2 - Area Tutore o Area Studente? 3 - Come creare tutti insieme i Tutori per ogni alunno? 3.1 - Come creare il secondo tutore per ogni alunno?
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
2.0 Gli archivi. 2.1 Inserire gli archivi. 2.2 Archivio Clienti, Fornitori, Materiali, Noleggi ed Altri Costi. Impresa Edile Guida all uso
 2.0 Gli archivi All interno della sezione archivi sono inserite le anagrafiche. In pratica si stratta di tutti quei dati che ricorreranno costantemente all interno dei documenti. 2.1 Inserire gli archivi
2.0 Gli archivi All interno della sezione archivi sono inserite le anagrafiche. In pratica si stratta di tutti quei dati che ricorreranno costantemente all interno dei documenti. 2.1 Inserire gli archivi
Tecniche di microscopia
 Tecniche di microscopia I microscopi permettono di vedere l estremamente piccolo I microscopi ottici utilizzano lenti di vetro in grado di deflettere e focalizzare i raggi luminosi per riprodurre le immagini
Tecniche di microscopia I microscopi permettono di vedere l estremamente piccolo I microscopi ottici utilizzano lenti di vetro in grado di deflettere e focalizzare i raggi luminosi per riprodurre le immagini
Esperienza 3: clonaggio di un gene in un plasmide
 Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Tecniche di sequenziamento del DNA
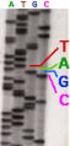 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3. Criteri di identificazione
 Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
LE MOLECOLE INFORMAZIONALI. Lezioni d'autore Treccani
 LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
In genere si ritiene che lo spreco di energia si manifesti solo nell uso dei carburanti, dei combustibili e dell energia elettrica.
 1 2 3 L energia incide sul costo di tutti i beni ed i servizi che utilizziamo ma, in questo caso, prendiamo come riferimento il costo che una famiglia di Aprilia con consumo medio sostiene ogni anno per
1 2 3 L energia incide sul costo di tutti i beni ed i servizi che utilizziamo ma, in questo caso, prendiamo come riferimento il costo che una famiglia di Aprilia con consumo medio sostiene ogni anno per
Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas
 1 Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas Capitolo 7 Enzimi, le macchine della vita Piccole macchine regolano la funzione del corpo umano in un orchestrazione perfetta e a velocità
1 Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas Capitolo 7 Enzimi, le macchine della vita Piccole macchine regolano la funzione del corpo umano in un orchestrazione perfetta e a velocità
Energia nelle reazioni chimiche. Lezioni d'autore di Giorgio Benedetti
 Energia nelle reazioni chimiche Lezioni d'autore di Giorgio Benedetti VIDEO Introduzione (I) L energia chimica è dovuta al particolare arrangiamento degli atomi nei composti chimici e le varie forme di
Energia nelle reazioni chimiche Lezioni d'autore di Giorgio Benedetti VIDEO Introduzione (I) L energia chimica è dovuta al particolare arrangiamento degli atomi nei composti chimici e le varie forme di
Ingegneria Genetica e Microbiologia Applicata
 Corso di Laurea in Biotecnologie Anno Accademico 2009-2010 Ingegneria Genetica e Microbiologia Applicata Percorso n 3: Clonaggio di segmenti di DNA Settima esercitazione - 13 maggio 2010 F 1 1 1: taglio
Corso di Laurea in Biotecnologie Anno Accademico 2009-2010 Ingegneria Genetica e Microbiologia Applicata Percorso n 3: Clonaggio di segmenti di DNA Settima esercitazione - 13 maggio 2010 F 1 1 1: taglio
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
RIPETITORE DI SEGNALE WIRELESS PER SISTEMA VIA RADIO ART. 45RPT000
 RIPETITORE DI SEGNALE WIRELESS PER SISTEMA VIA RADIO ART. 45RPT000 Leggere questo manuale prima dell uso e conservarlo per consultazioni future 1 DESCRIZIONE GENERALE L espansore senza fili è un modulo
RIPETITORE DI SEGNALE WIRELESS PER SISTEMA VIA RADIO ART. 45RPT000 Leggere questo manuale prima dell uso e conservarlo per consultazioni future 1 DESCRIZIONE GENERALE L espansore senza fili è un modulo
MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013]
![MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013] MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013]](/thumbs/25/5308947.jpg) MOCA Modulo Candidatura http://www.federscacchi.it/moca moca@federscacchi.it [Manuale versione 1.0 marzo 2013] 1/12 MOCA in breve MOCA è una funzionalità del sito web della FSI che permette di inserire
MOCA Modulo Candidatura http://www.federscacchi.it/moca moca@federscacchi.it [Manuale versione 1.0 marzo 2013] 1/12 MOCA in breve MOCA è una funzionalità del sito web della FSI che permette di inserire
lo 2 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000
 Capittol lo 2 Visualizzazione 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000 Nel primo capitolo sono state analizzate le diverse componenti della finestra di Word 2000: barra del titolo, barra dei menu,
Capittol lo 2 Visualizzazione 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000 Nel primo capitolo sono state analizzate le diverse componenti della finestra di Word 2000: barra del titolo, barra dei menu,
Analisi e diagramma di Pareto
 Analisi e diagramma di Pareto L'analisi di Pareto è una metodologia statistica utilizzata per individuare i problemi più rilevanti nella situazione in esame e quindi le priorità di intervento. L'obiettivo
Analisi e diagramma di Pareto L'analisi di Pareto è una metodologia statistica utilizzata per individuare i problemi più rilevanti nella situazione in esame e quindi le priorità di intervento. L'obiettivo
Il campionamento. La digitalizzazione. Teoria e pratica. La rappresentazione digitale delle immagini. La rappresentazione digitale delle immagini
 ACQUISIZIONE ED ELABORAZIONE DELLE IMMAGINI Teoria e pratica La digitalizzazione La digitalizzazione di oggetti legati a fenomeni di tipo analogico, avviene attraverso due parametri fondamentali: Il numero
ACQUISIZIONE ED ELABORAZIONE DELLE IMMAGINI Teoria e pratica La digitalizzazione La digitalizzazione di oggetti legati a fenomeni di tipo analogico, avviene attraverso due parametri fondamentali: Il numero
VALORE DELLE MERCI SEQUESTRATE
 La contraffazione in cifre: NUOVA METODOLOGIA PER LA STIMA DEL VALORE DELLE MERCI SEQUESTRATE Roma, Giugno 2013 Giugno 2013-1 Il valore economico dei sequestri In questo Focus si approfondiscono alcune
La contraffazione in cifre: NUOVA METODOLOGIA PER LA STIMA DEL VALORE DELLE MERCI SEQUESTRATE Roma, Giugno 2013 Giugno 2013-1 Il valore economico dei sequestri In questo Focus si approfondiscono alcune
FIRESHOP.NET. Gestione completa delle fidelity card & raccolta punti. Rev. 2014.3.1 www.firesoft.it
 FIRESHOP.NET Gestione completa delle fidelity card & raccolta punti Rev. 2014.3.1 www.firesoft.it Sommario SOMMARIO Introduzione... 3 La gestione delle fidelity card nel POS... 4 Codificare una nuova fidelity
FIRESHOP.NET Gestione completa delle fidelity card & raccolta punti Rev. 2014.3.1 www.firesoft.it Sommario SOMMARIO Introduzione... 3 La gestione delle fidelity card nel POS... 4 Codificare una nuova fidelity
La preparazione per le gare brevi
 I documenti di www.mistermanager.it La preparazione per le gare brevi La maggior parte delle competizioni su cui si cimentano i podisti, sono le gare su strada, le cui lunghezze solitamente sono intorno
I documenti di www.mistermanager.it La preparazione per le gare brevi La maggior parte delle competizioni su cui si cimentano i podisti, sono le gare su strada, le cui lunghezze solitamente sono intorno
www.andreatorinesi.it
 La lunghezza focale Lunghezza focale Si definisce lunghezza focale la distanza tra il centro ottico dell'obiettivo (a infinito ) e il piano su cui si forma l'immagine (nel caso del digitale, il sensore).
La lunghezza focale Lunghezza focale Si definisce lunghezza focale la distanza tra il centro ottico dell'obiettivo (a infinito ) e il piano su cui si forma l'immagine (nel caso del digitale, il sensore).
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
MONOPOLIO, MONOPOLISTA
 Barbara Martini OBIETTIVI IL SIGNIFICATO DI MONOPOLIO, IN CUI UN SINGOLO MONOPOLISTA È L UNICO PRODUTTORE DI UN BENE COME UN MONOPOLISTA DETERMINA L OUTPUT ED IL PREZZO CHE MASSIMIZZANO IL PROFITTO LA
Barbara Martini OBIETTIVI IL SIGNIFICATO DI MONOPOLIO, IN CUI UN SINGOLO MONOPOLISTA È L UNICO PRODUTTORE DI UN BENE COME UN MONOPOLISTA DETERMINA L OUTPUT ED IL PREZZO CHE MASSIMIZZANO IL PROFITTO LA
( x) ( x) 0. Equazioni irrazionali
 Equazioni irrazionali Definizione: si definisce equazione irrazionale un equazione in cui compaiono uno o più radicali contenenti l incognita. Esempio 7 Ricordiamo quanto visto sulle condizioni di esistenza
Equazioni irrazionali Definizione: si definisce equazione irrazionale un equazione in cui compaiono uno o più radicali contenenti l incognita. Esempio 7 Ricordiamo quanto visto sulle condizioni di esistenza
Le Biomolecole I parte. Lezioni d'autore di Giorgio Benedetti
 Le Biomolecole I parte Lezioni d'autore di Giorgio Benedetti LE BIOMOLECOLE Le biomolecole, presenti in tutti gli esseri viventi, sono molecole composte principalmente da carbonio, idrogeno, azoto e ossigeno.
Le Biomolecole I parte Lezioni d'autore di Giorgio Benedetti LE BIOMOLECOLE Le biomolecole, presenti in tutti gli esseri viventi, sono molecole composte principalmente da carbonio, idrogeno, azoto e ossigeno.
IL RISPARMIO ENERGETICO E GLI AZIONAMENTI A VELOCITA VARIABILE L utilizzo dell inverter negli impianti frigoriferi.
 IL RISPARMIO ENERGETICO E GLI AZIONAMENTI A VELOCITA VARIABILE L utilizzo dell inverter negli impianti frigoriferi. Negli ultimi anni, il concetto di risparmio energetico sta diventando di fondamentale
IL RISPARMIO ENERGETICO E GLI AZIONAMENTI A VELOCITA VARIABILE L utilizzo dell inverter negli impianti frigoriferi. Negli ultimi anni, il concetto di risparmio energetico sta diventando di fondamentale
Gestione Turni. Introduzione
 Gestione Turni Introduzione La gestione dei turni di lavoro si rende necessaria quando, per garantire la continuità del servizio di una determinata struttura, è necessario che tutto il personale afferente
Gestione Turni Introduzione La gestione dei turni di lavoro si rende necessaria quando, per garantire la continuità del servizio di una determinata struttura, è necessario che tutto il personale afferente
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
INDICE. Accesso al Portale Pag. 2. Nuovo preventivo - Ricerca articoli. Pag. 4. Nuovo preventivo Ordine. Pag. 6. Modificare il preventivo. Pag.
 Gentile Cliente, benvenuto nel Portale on-line dell Elettrica. Attraverso il nostro Portale potrà: consultare la disponibilità dei prodotti nei nostri magazzini, fare ordini, consultare i suoi prezzi personalizzati,
Gentile Cliente, benvenuto nel Portale on-line dell Elettrica. Attraverso il nostro Portale potrà: consultare la disponibilità dei prodotti nei nostri magazzini, fare ordini, consultare i suoi prezzi personalizzati,
13. Campi vettoriali
 13. Campi vettoriali 1 Il campo di velocità di un fluido Il concetto di campo in fisica non è limitato ai fenomeni elettrici. In generale il valore di una grandezza fisica assegnato per ogni punto dello
13. Campi vettoriali 1 Il campo di velocità di un fluido Il concetto di campo in fisica non è limitato ai fenomeni elettrici. In generale il valore di una grandezza fisica assegnato per ogni punto dello
I documenti di www.mistermanager.it. Gli ingredienti per l allenamento per la corsa LE RIPETUTE
 I documenti di www.mistermanager.it Gli ingredienti per l allenamento per la corsa LE RIPETUTE Le Ripetute sono una delle forme di allenamento che caratterizzano i corridori più evoluti, in quanto partono
I documenti di www.mistermanager.it Gli ingredienti per l allenamento per la corsa LE RIPETUTE Le Ripetute sono una delle forme di allenamento che caratterizzano i corridori più evoluti, in quanto partono
PROGRAMMA GESTIONE TURNI MANUALE UTENTE. Programma Gestione Turni Manuale Utente versione 1.1
 PROGRAMMA GESTIONE TURNI MANUALE UTENTE INDICE 1 PREMESSA 3 2 COMANDI COMUNI 3 3 SEDI 3 4 FESTIVITÀ 4 5 PERIODI TURNI 4 6 COD. TURNI 6 7 TURNI SPORTIVI 9 8 COD. EQUIPAGGI 9 9 DISPONIBILITÀ 10 10 INDISPONIBILITÀ
PROGRAMMA GESTIONE TURNI MANUALE UTENTE INDICE 1 PREMESSA 3 2 COMANDI COMUNI 3 3 SEDI 3 4 FESTIVITÀ 4 5 PERIODI TURNI 4 6 COD. TURNI 6 7 TURNI SPORTIVI 9 8 COD. EQUIPAGGI 9 9 DISPONIBILITÀ 10 10 INDISPONIBILITÀ
MARCATURA DEGLI ACIDI NUCLEICI
 MARCATURA DEGLI ACIDI NUCLEICI MARCATURA DEGLI ACIDI NUCLEICI La marcatura degli acidi nucleici è una delle tecniche di base nello studio della Biologia Molecolare, rappresenta infatti una tappa preliminare
MARCATURA DEGLI ACIDI NUCLEICI MARCATURA DEGLI ACIDI NUCLEICI La marcatura degli acidi nucleici è una delle tecniche di base nello studio della Biologia Molecolare, rappresenta infatti una tappa preliminare
INTEGRATORE E DERIVATORE REALI
 INTEGRATORE E DERIVATORE REALI -Schemi elettrici: Integratore reale : C1 R2 vi (t) R1 vu (t) Derivatore reale : R2 vi (t) R1 C1 vu (t) Elenco componenti utilizzati : - 1 resistenza da 3,3kΩ - 1 resistenza
INTEGRATORE E DERIVATORE REALI -Schemi elettrici: Integratore reale : C1 R2 vi (t) R1 vu (t) Derivatore reale : R2 vi (t) R1 C1 vu (t) Elenco componenti utilizzati : - 1 resistenza da 3,3kΩ - 1 resistenza
UTILIZZATORI A VALLE: COME RENDERE NOTI GLI USI AI FORNITORI
 UTILIZZATORI A VALLE: COME RENDERE NOTI GLI USI AI FORNITORI Un utilizzatore a valle di sostanze chimiche dovrebbe informare i propri fornitori riguardo al suo utilizzo delle sostanze (come tali o all
UTILIZZATORI A VALLE: COME RENDERE NOTI GLI USI AI FORNITORI Un utilizzatore a valle di sostanze chimiche dovrebbe informare i propri fornitori riguardo al suo utilizzo delle sostanze (come tali o all
LA GENETICA: DNA e RNA LA GENETICA. DNA e RNA. Prof. Daniele Verri
 LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Introduzione. Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD... 6
 Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
Transitori del primo ordine
 Università di Ferrara Corso di Elettrotecnica Transitori del primo ordine Si consideri il circuito in figura, composto da un generatore ideale di tensione, una resistenza ed una capacità. I tre bipoli
Università di Ferrara Corso di Elettrotecnica Transitori del primo ordine Si consideri il circuito in figura, composto da un generatore ideale di tensione, una resistenza ed una capacità. I tre bipoli
Sez. J.1 Sistemi e tecnologie ad aria compressa, di ausilio alla produzione SISTEMI DI RAFFREDDAMENTO TUBI VORTEX FRIGID-X TM VORTEX TUBE
 Sez. J.1 Sistemi e tecnologie ad aria compressa, di ausilio alla produzione SISTEMI DI RAFFREDDAMENTO DC COOLING TUBI VORTEX FRIGID-X TM VORTEX TUBE Documentazione non registrata, soggetta a modifiche
Sez. J.1 Sistemi e tecnologie ad aria compressa, di ausilio alla produzione SISTEMI DI RAFFREDDAMENTO DC COOLING TUBI VORTEX FRIGID-X TM VORTEX TUBE Documentazione non registrata, soggetta a modifiche
Mon Ami 3000 Centri di costo Contabilità analitica per centri di costo/ricavo e sub-attività
 Prerequisiti Mon Ami 000 Centri di costo Contabilità analitica per centri di costo/ricavo e sub-attività L opzione Centri di costo è disponibile per le versioni Contabilità o Azienda Pro. Introduzione
Prerequisiti Mon Ami 000 Centri di costo Contabilità analitica per centri di costo/ricavo e sub-attività L opzione Centri di costo è disponibile per le versioni Contabilità o Azienda Pro. Introduzione
Come utilizzare il sistema per ricavare e scaricare un elenco iscritti per la propria gara
 Come utilizzare il sistema per ricavare e scaricare un elenco iscritti per la propria gara Le società organizzatrici attraverso le utilità fornite dal sistema di gestione gare hanno la possibilità di ricavare
Come utilizzare il sistema per ricavare e scaricare un elenco iscritti per la propria gara Le società organizzatrici attraverso le utilità fornite dal sistema di gestione gare hanno la possibilità di ricavare
Genova 15 01 14 TIPOLOGIE DI LAMPADE
 Genova 15 01 14 TIPOLOGIE DI LAMPADE Le lampade a vapori di mercurio sono sicuramente le sorgenti di radiazione UV più utilizzate nella disinfezione delle acque destinate al consumo umano in quanto offrono
Genova 15 01 14 TIPOLOGIE DI LAMPADE Le lampade a vapori di mercurio sono sicuramente le sorgenti di radiazione UV più utilizzate nella disinfezione delle acque destinate al consumo umano in quanto offrono
Abbiamo costruito il grafico delle sst in funzione del tempo (dal 1880 al 1995).
 ANALISI DI UNA SERIE TEMPORALE Analisi statistica elementare Abbiamo costruito il grafico delle sst in funzione del tempo (dal 1880 al 1995). Si puo' osservare una media di circa 26 C e una deviazione
ANALISI DI UNA SERIE TEMPORALE Analisi statistica elementare Abbiamo costruito il grafico delle sst in funzione del tempo (dal 1880 al 1995). Si puo' osservare una media di circa 26 C e una deviazione
LE CARATTERISTICHE DEI PRODOTTI MULTIVARIANTE
 LE CARATTERISTICHE DEI PRODOTTI MULTIVARIANTE Che cosa sono e a cosa servono le caratteristiche? Oltre a descrivere le qualità di un prodotto con un testo generico (descrizione) è possibile dettagliare
LE CARATTERISTICHE DEI PRODOTTI MULTIVARIANTE Che cosa sono e a cosa servono le caratteristiche? Oltre a descrivere le qualità di un prodotto con un testo generico (descrizione) è possibile dettagliare
Lezioni di biotecnologie
 Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Guida Compilazione Piani di Studio on-line
 Guida Compilazione Piani di Studio on-line SIA (Sistemi Informativi d Ateneo) Visualizzazione e presentazione piani di studio ordinamento 509 e 270 Università della Calabria (Unità organizzativa complessa-
Guida Compilazione Piani di Studio on-line SIA (Sistemi Informativi d Ateneo) Visualizzazione e presentazione piani di studio ordinamento 509 e 270 Università della Calabria (Unità organizzativa complessa-
ITIS Mattei Sondrio. Appunti veloci su moodle versione 2.7
 ITIS Mattei Sondrio Appunti veloci su moodle versione 2.7 Generalità Moodle è una piattaforma web per pubblicare corsi, creare test, depositare dispense ecc. Ci sono vari tipi di utenti che usano moodle:
ITIS Mattei Sondrio Appunti veloci su moodle versione 2.7 Generalità Moodle è una piattaforma web per pubblicare corsi, creare test, depositare dispense ecc. Ci sono vari tipi di utenti che usano moodle:
Mon Ami 3000 Varianti articolo Gestione di varianti articoli
 Prerequisiti Mon Ami 3000 Varianti articolo Gestione di varianti articoli L opzione Varianti articolo è disponibile per le versioni Azienda Light e Azienda Pro e include tre funzionalità distinte: 1. Gestione
Prerequisiti Mon Ami 3000 Varianti articolo Gestione di varianti articoli L opzione Varianti articolo è disponibile per le versioni Azienda Light e Azienda Pro e include tre funzionalità distinte: 1. Gestione
Il Problem-Based Learning dalla pratica alla teoria
 Il Problem-Based Learning dalla pratica alla teoria Il Problem-based learning (apprendimento basato su un problema) è un metodo di insegnamento in cui un problema costituisce il punto di inizio del processo
Il Problem-Based Learning dalla pratica alla teoria Il Problem-based learning (apprendimento basato su un problema) è un metodo di insegnamento in cui un problema costituisce il punto di inizio del processo
Funzioni in C. Violetta Lonati
 Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Biosintesi non ribosomiale di metaboliti peptidici bioattivi
 Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
Soluzioni per il risparmio idrico per i WC
 Soluzioni per il risparmio idrico per i WC Per un utenza standard di tipo residenziale che utilizza cassette di risciacquo di tipo convenzionale (da 9 a 12 litri per risciacquo), il 30% dei consumi di
Soluzioni per il risparmio idrico per i WC Per un utenza standard di tipo residenziale che utilizza cassette di risciacquo di tipo convenzionale (da 9 a 12 litri per risciacquo), il 30% dei consumi di
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
La mutazione è una modificazione della sequenza delle basi del DNA
 La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
LUdeS Informatica 2 EXCEL. Seconda parte AA 2013/2014
 LUdeS Informatica 2 EXCEL Seconda parte AA 2013/2014 STAMPA Quando si esegue il comando FILE STAMPA, Excel manda alla stampante tutte le celle del foglio di lavoro corrente che hanno un contenuto. Il numero
LUdeS Informatica 2 EXCEL Seconda parte AA 2013/2014 STAMPA Quando si esegue il comando FILE STAMPA, Excel manda alla stampante tutte le celle del foglio di lavoro corrente che hanno un contenuto. Il numero
Excel. A cura di Luigi Labonia. e-mail: luigi.lab@libero.it
 Excel A cura di Luigi Labonia e-mail: luigi.lab@libero.it Introduzione Un foglio elettronico è un applicazione comunemente usata per bilanci, previsioni ed altri compiti tipici del campo amministrativo
Excel A cura di Luigi Labonia e-mail: luigi.lab@libero.it Introduzione Un foglio elettronico è un applicazione comunemente usata per bilanci, previsioni ed altri compiti tipici del campo amministrativo
EXCEL PER WINDOWS95. sfruttare le potenzialità di calcolo dei personal computer. Essi si basano su un area di lavoro, detta foglio di lavoro,
 EXCEL PER WINDOWS95 1.Introduzione ai fogli elettronici I fogli elettronici sono delle applicazioni che permettono di sfruttare le potenzialità di calcolo dei personal computer. Essi si basano su un area
EXCEL PER WINDOWS95 1.Introduzione ai fogli elettronici I fogli elettronici sono delle applicazioni che permettono di sfruttare le potenzialità di calcolo dei personal computer. Essi si basano su un area
