Lezione 7. Allineamento di sequenze biologiche
|
|
|
- Bianca Zamboni
- 7 anni fa
- Visualizzazioni
Transcript
1 Lezione 7 Allineamento di sequenze biologiche
2 Allineamento di sequenze Determinare la similarità e dedurre l omologia
3 Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra utilizzando passaggi mutazionali come -sostituzione -inserzione/delezione mismatch 1 LA CASA È NUOVA 2 LA CASSA È VUOTA 1 LA CAS-A È NUO-VA 2 LA CASSA È V-UOT-A 5 indels gap 1 LA CASA È NUOVA 2 LA CASSA È VUOTA 1 LA CAS-A È NUOVA 2 LA CASSA È VUOTA match 1 indels + 2 sostituzioni Tra le due soluzioni mostrate (ce ne sono altre!), quale scegliamo? Esistono criteri e algoritmi che ci possono aiutare
4 Perchè allineare? Per fornire una misura di quanto sequenze nucleotidiche o aminoacidiche siano imparentate, abbiano in comune Questa parentela ci permette di fare inferenze biologiche in termini di relazioni strutturali relazioni funzionali relazioni evolutive Alignment-based database searching
5 Terminologia La misura QUANTITATIVA: Similarità Si esprime in genere come % di identità, quantifica i cambiamenti che sono avvenuti dal momento della divergenza tra due specie (sostituzioni, Indels) Identifica i residui cruciali per mantenere la struttura o la funzione di una proteina Alti livelli di similarità possono indicare una divergenza recente tra le sequenze, una storia evolutiva comune, simile funzione biologica
6 Terminologia Una valutazione di STATO: Omologia Implica l esistenza di relazioni evolutive Geni omologhi: geni che si sono originati per divergenza da un antenato comune I geni SONO o NON SONO omologhi, non esiste una misura quantitativa dell omologia
7 Eyeless ha un ruolo importante nel dirigere lo sviluppo dell occhio in drosofila; Pax6 lo stesso nel topo Eyeless e Pax6 sono decisamente simili in sequenza e funzione probably > 500 MYA
8 Terminologia Ortologhi: Geni che si sono separati in seguito ad un evento di speciazione Le sequenze discendono da un antenato comune Molto probabilmente codificano per proteine con domini simili e simili strutture tridimensionali Spesso mantengono funzioni simili Possono essere usati per predire funzioni geniche in genomi nuovi Paraloghi: Geni che si sono evoluti per duplicazione in una specifica linea evolutiva E meno probabile che mantengano funzioni simili, più comunemente evolvono nuove funzioni
9 Homologous sequences. Orthologs and Paralogs are two types of homologous sequences. Orthology describes genes in different species that derive from a common ancestor. Orthologous genes may or may not have the same function. Paralogy describes homologous genes within a single species that diverged by gene duplication.
10 Globale Allineamenti globali e locali trova l allineamento ottimale sul totale della lunghezza delle sequenze È la soluzione migliore per sequenze di lunghezza simile ed omologhe Al dimiuire del grado di similarità (es. aumento distanza evolutiva, alto tasso di ricombinazione) i metodi di allineamento globale tendono a peggiorare molto in efficienza
11 Cercare in GENE di NCBI PAX6 and mouse > scaricare il cds in format FASTA > incollare nell allineatore di BLAST PAX6 and chimpanzee > scaricare il cds in format FASTA > incollare nell allineatore di BLAST Cosa otteniamo?
12 Locale Allineamenti globali e locali Ha lo scopo di trovare regioni simili (es. domini) in due sequenze ( paired subsequences ) Le regioni fuori dalle aree di allineamento locale vengono escluse Può essere generato più di un allineamento locale per ogni coppia di sequenze confrontate Scelta indicata nel caso di due sequenze a similarità ridotta o di differenti lunghezze
13 Local vs. Global Alignment Global Alignment --T -CC-C-AGT -TATGT-CAGGGGACACG A-GCATGCAGA-GAC AATTGCCGCC-GTCGT-T-TTCAG----CA-GTTATG T-CAGAT--C Local Alignment migliore per trovare regioni conservate tcccagttatgtcaggggacacgagcatgcagagac aattgccgccgtcgttttcagcagttatgtcagatc
14 Allineamenti locali: perchè? Due geni in specie diverse possono essere simili in corte regioni conservate e diversi nel resto della sequenza. Esempio: I geni Homeobox (chiaramente omologhi) hanno corte regioni chiamate omeodomini altamente conservate tra specie. Un allineamento globale non troverebbe gli omeodomini perchè cercherebbe di allineare l INTERA sequenza
15 Allineamento: ipotesi circa l omologia posizionale (discendenza da antenato comune) di due residui in due (o più) sequenze Sequenza ancestrale (prima della speciazione: antenato comune delle due sequenze. Non direttamente osservabile (a meno di avere il DNA antico), ma ricostruibile base nr GCGGTCCATCAGCTGGTTGGTGG passato T > C pos 5 C > G pos 13 G > A pos 15 G > T pos 4 Del AG pos 11 e 12 Ins T pos 23 GCGGCCCATCAGGTAGTTGGTGG GCGTTCCATCCTGGTTGGTGTG Sequenza della specie 1 sequenza della specie 2 presente
16 Un allineamento a coppie consiste di una serie di residui o basi accoppiati, una per sequenza. Ci sono tre tipi di coppie: (a) match = stesso nucleotide (o AA) in entrambe le sequenze (b) mismatch = diverso nucleotide (o AA) in una delle sequenze (c) gap = una base (o AA) in una sequenza e niente nell altra Specie1 GCGGCCCATCAGGTAGTTGGTG-G Specie2 GCGTTCCATC--CTGGTTGGTGTG aaabbaaaaaccbabaaaaaaaca Come si può fare in modo non manuale??
17 Nelle prossime diapositive cercheremo di rispondere alla domanda: su che cosa si basa un allineatore (algoritmo di allineamento) per operare un allineamento? Come si può fare in modo non manuale??
18 1. Matrici di punteggio e 2. Penalità per i gap Sostituzioni o mismatch In/del o gap Il vero allineamento tra due sequenze è quello che riflette in modo accurato le loro relazioni evolutive (vedi i numerini nell esempio precedente: omologia posizionale). Poichè il vero allineamento non è conosciuto in pratica si cerca l allineamento ottimale: minimizza i mismatches e i gaps secondo certi criteri.purtroppo mms gaps gaps mms
19 ( ( ( Matches Mismatches Gaps (1 terminal) Matches Mismatches Gaps Matches Mismatches Gaps (both terminal)
20 Matrici di punteggio e penalità per i gap Lo schema di punteggio include una penalizzazione per le in-del (gap penalty) e una matrice di punteggio (scoring matrix) M(a,b), che specifica ogni tipo di match (a = b) o di mismatch (a b). Le unità nella matrice di punteggio possono essere nucleotidi nelle sequenze di DNA o RNA, i codoni nelle regioni codificanti, o gli aminoacidi nelle sequenze proteiche.
21 Sostituzioni : mismatches Cos è una matrice di punteggio? Matrice che associa un punteggio ad ogni coppia di entità che troviamo in un allineamento Ogni linea e ogni colonna rappresentano un residuo (4 nucleotidi o 20 aminoacidi) La diagonale è l identità Il triangolo inferiore corrisponde alle sostituzioni e il superiore è simmetrico (non necessario) I valori negativi indicano penalità per certe sostituzioni, l algoritmo di allineamento cercherà di evitarle I valori positivi indicano sostituzioni accettate in termini evoolutivi, strutturali o funzionali
22 Sostituzioni : mismatches Perché è importante capire le matrici di punteggio? Compaiono in ogni analisi che implichi un confronto tra sequenze Implicano un determinato percorso evolutivo Possono influenzare fortemente il risultato delle analisi
23 Sostituzioni : mismatches DNA scoring matrices Di solito sono semplici. La più semplice: M(a,b) assegna valori positivi se a = b (match), altrimenti negativi (mismatch) M(a,b) 0 if a b 0 if a b
24 Sostituzioni : mismatches DNA scoring matrices Matrici più complesse possono distinguere ad esempio tra transizioni e trasversioni (le prime avvengono più facilmente trattandosi di molecole più simili, però ci sono 4 possibili trasversioni e solo 2 transizioni)
25 Sostituzioni : mismatches Amino acid/protein scoring matrices Margareth Dayhoff 1965: Atlas of potein sequences contenente le sequenze aminoacidiche di 65 proteine Inizio delle collezioni di dati da cui avranno origine le banche dati elettroniche Dayhoff et al. nel decennio hanno proposto una procedura per il calcolo di matrici di punteggio per quantificare la propensione di AA a mutare l uno nell altro durante l evoluzione (matrici 20 x 20). Alla base c è l osservazione delle proteine note: MATRICI DI SOSTITUZIONI EMPIRICHE
26 Sostituzioni : mismatches Amino acid/protein scoring matrices Empirical substitution matrices PAM matrix (Percent/Point Accepted Mutation Matrix) BLOSUM (BLOcks SUbstitution Matrix)
27 ogni valore indica la probabilità che l AAx sia sostituito con l AAy attraverso una o più mutazioni accettate in uno specifico intervallo evolutivo, rispetto alla probabilità che i due aminoacidi siano stati allineati per caso Sostituzioni : mismatches P > 0 P = 0 P < 0 lo scambio AAx AAy si osserva più frequentemente di quanto atteso per caso la frequenza della sostituzione è indistinguibile da quella casuale lo scambio AAx AAy è più raro di quanto atteso per caso
28 Sostituzioni : mismatches BLOSUM (BLOcks SUbstitution Matrix) Henikoff and Henikoff (1992): matrice basata su molte più osservazioni della PAM: scambi aminoacidici calcolati su circa 2000 «blocchi» Blocco: regione conservata di una famiglia di proteine senza indels Direttamente calcolate sulla base di allineamenti locali Probabilità di sostituzione (conservazione) Frequenza degli aminoacidi
29 Default in BLAST Sostituzioni : mismatches E: Asp D: Glu Maggiore il punteggio (score) maggiormente imparentate sono le sequenze
30 Sostituzioni : mismatches Maggiore è il valore, più simili sono le proteine utilizzate nel calcolo della matrice
31 In/Dels : gaps Gap penalties Quanto è probabile una Costo delle indels (GAP) certa sostituzione (matrici) Costo dell introduzione di un gap (Gap opening penalty: G) Costo dell estensione di un gap (Gap extension penalty: L*n) Costo complessivo: G+Ln Questi sono valori usati spesso, ma si possono cambiare!
32
33
34 Algoritmi di allineamento Obiettivo: trovare il miglior allineamento, cioè il massimo numero di simboli identici e il minor numero di gap (=minor numero di mutazioni = più breve percorso evolutivo) Per due sequenze di DNA di 200 basi ci sono possibili allineamenti.meglio non farli a mano!
35 Dynamic programming = tecnica computazionale. Si usa per effettuare ricerche complesse dividendole in una successione di piccoli passaggi, inizialmente semplici e poi più complessi. L ultimo passaggio contiene la soluzione complessiva
36 42
37 Algoritmi di allineamento Esausitivi o esatti: esplorano tutte le possibili soluzioni e scelgono la migliore (lenti, computazionalmente intensi, precisi) Euristici: prendono scorciatoie e cercano di arrivare ad una soluzione ottimale basandosi su ipotesi plausibili Algoritmi di allineamento comuni Algoritmo Esaustivo? Loc/Glo Mul align Db searches Needleman -Wunsch Smith- Waterman Si Global Si No Si Local Si Si FASTA No Local Si Si BLAST No Local No Si
38 Needleman-Wunsch Exact global alignment method Non adatto in molti casi (es. db searches, ricerca di piccole regioni di similarità, allinemanti tra sequenze con grosse differenze di lunghezza) Il più rigoroso e completo se lo scopo è di allineare sequenze che non si sono evolute per exon shuffling, inserzione/delezione di domini, etc. Il metodo migliore se le sequenze sono di lunghezze simili e si sono evolute da un antenato comune attraverso mutazioni di punto, piccole ind/dels
39 Smith-Waterman Exact local alignment method Modifica del N-W che permette di allineare in locale (non serve allineare tutta la seq) Allineamento molto buono per db searching, allineamento multiplo e a coppie Esaustivo, quindi può essere molto lento. A differenza del N-W considera qualunque allineamento che parta da qualunque posizione della sequenza, non solo quelli che cominciano all inizio e terminano alla fine
40 L algoritmo deve identificare le sequenze omologhe e non omologhe separate da un valore soglia Ricerche in database Query (sequenza sonda) ricerca Sequenze nelle banche dati FP: falsi positivi Caso 1: buon lavoro dell algoritmo Sequenze non omologhe Sequenze omologhe VP: veri positivi FN: falsi negativi VN: veri negativi Caso 2: c è una zona in cui non è possibile discriminare omologhe e non VN Regione di sovrapposizione FN FP VP Punteggio soglia
41 FASTA: Pearson WR (1996) Effective protein sequence comparison. Academic Press Inc Pearson WR and Lipman DJ (1998) Improved tools for biological sequence comparison. PNAS 85:2444 Euristico locale Prima identifica regioni di identità tra la sequenza sonda ( query ) e le sequenze in db. (KTUP) I geni o proteine con la densità maggiore di segnale vengono riesaminati L allineamento viene esteso ad entrambi i lati delle regioni di match aggiungendo gaps e mismatches sulla base di matrici di punteggio L allineamento ottiene un punteggio NB: leggere l HELP del programma
42 E value: significatività statistica Non si interpretano come p values dove p < 0.05 sono generalmente considerati significativi Regola generale E values < 10-6 sono molto probabilmente significativi < E values < 10-3 meritano una seconda occhiata. E values < 10-3 andrebbero scartati (ci aspettiamo di trovare sequenze non correlate alla nostra-falsi positivi- che ottengono un punteggio superiore a quell S).
Lezione 7. Allineamento di sequenze biologiche
 Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
Allineamento e similarità di sequenze
 Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Omologia di sequenze: allineamento e ricerca
 Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
FASTA: Lipman & Pearson (1985) BLAST: Altshul (1990) Algoritmi EURISTICI di allineamento
 Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Organizzazione del genoma umano
 Organizzazione del genoma umano Famiglie di geni o geniche Copie multiple di geni, tutte con sequenza identica o simile. La famiglia multigenica corrisponde a un insieme di geni correlati che si sono evoluti
Organizzazione del genoma umano Famiglie di geni o geniche Copie multiple di geni, tutte con sequenza identica o simile. La famiglia multigenica corrisponde a un insieme di geni correlati che si sono evoluti
Lezione 2: Allineamento di sequenze. BLAST e CLUSTALW
 Lezione 2: Allineamento di sequenze BLAST e CLUSTALW Allineamento di sequenze Allineamenti L avvento della genomica moderna permette di analizzare le similitudini e le differenze tra organismi a livello
Lezione 2: Allineamento di sequenze BLAST e CLUSTALW Allineamento di sequenze Allineamenti L avvento della genomica moderna permette di analizzare le similitudini e le differenze tra organismi a livello
Ricerca di omologia di sequenza
 Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Z-score. lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random
 Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Informatica e biotecnologie II parte
 Informatica e biotecnologie II parte Analisi di sequenze: allineamenti CGCTTCGGACGAAATCGCATCAGCATACGATCGCATGCCGGGCGGGATAAC CGAAATCGCATCAGCATACGATCGCATGC Bioinformatica La Bioinformatica è una disciplina
Informatica e biotecnologie II parte Analisi di sequenze: allineamenti CGCTTCGGACGAAATCGCATCAGCATACGATCGCATGCCGGGCGGGATAAC CGAAATCGCATCAGCATACGATCGCATGC Bioinformatica La Bioinformatica è una disciplina
Bioinformatica e Biologia Computazionale per la Medicina Molecolare
 Facoltà di Ingegneria dell Informazione Laurea Specialistica e Magistrale in Ingegneria Informatica Facoltà di Ingegneria dei Sistemi Laurea Magistrale in Ingegneria Biomedica Dipartimento di Elettronica
Facoltà di Ingegneria dell Informazione Laurea Specialistica e Magistrale in Ingegneria Informatica Facoltà di Ingegneria dei Sistemi Laurea Magistrale in Ingegneria Biomedica Dipartimento di Elettronica
q xi Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p i >0;
 Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
La ricerca di similarità: i metodi
 La ricerca di similarità: i metodi Pairwise alignment allineamenti a coppie 1. Analisi della matrice a punti (dot matrix) 2. Programmazione dinamica (dynamic programming) allineamenti locale e globale.
La ricerca di similarità: i metodi Pairwise alignment allineamenti a coppie 1. Analisi della matrice a punti (dot matrix) 2. Programmazione dinamica (dynamic programming) allineamenti locale e globale.
Lezione 1. Le molecole di base che costituiscono la vita
 Lezione 1 Le molecole di base che costituiscono la vita Le molecole dell ereditarietà 5 3 L informazione ereditaria di tutti gli organismi viventi, con l eccezione di alcuni virus, è a carico della molecola
Lezione 1 Le molecole di base che costituiscono la vita Le molecole dell ereditarietà 5 3 L informazione ereditaria di tutti gli organismi viventi, con l eccezione di alcuni virus, è a carico della molecola
Come si sceglie l algoritmo di allineamento? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione?
 Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
A W T V A S A V R T S I A Y T V A A A V R T S I A Y T V A A A V L T S I
 COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
Corso di Bioinformatica
 Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
Relazione sequenza-struttura e funzione
 Biotecnologie applicate alla progettazione e sviluppo di molecole biologicamente attive A.A. 2010-2011 Modulo di Biologia Strutturale Relazione sequenza-struttura e funzione Marco Nardini Dipartimento
Biotecnologie applicate alla progettazione e sviluppo di molecole biologicamente attive A.A. 2010-2011 Modulo di Biologia Strutturale Relazione sequenza-struttura e funzione Marco Nardini Dipartimento
Biologia Molecolare Computazionale
 Biologia Molecolare Computazionale Paolo Provero - paolo.provero@unito.it 2008-2009 Argomenti Allineamento di sequenze Ricostruzione di alberi filogenetici Gene prediction Allineamento Allineamento di
Biologia Molecolare Computazionale Paolo Provero - paolo.provero@unito.it 2008-2009 Argomenti Allineamento di sequenze Ricostruzione di alberi filogenetici Gene prediction Allineamento Allineamento di
Ricerche con BLAST (Laboratorio)
 Laboratorio di Bioinformatica I Ricerche con BLAST (Laboratorio) Dott. Sergio Marin Vargas (2014 / 2015) NCBI BLAST BLAST: Basic Local Alignment Search Tool http://blast.ncbi.nlm.nih.gov/blast.cgi NCBI
Laboratorio di Bioinformatica I Ricerche con BLAST (Laboratorio) Dott. Sergio Marin Vargas (2014 / 2015) NCBI BLAST BLAST: Basic Local Alignment Search Tool http://blast.ncbi.nlm.nih.gov/blast.cgi NCBI
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Corso di Bioinformatica. Docente: Dr. Antinisca DI MARCO
 Corso di Bioinformatica Docente: Dr. Antinisca DI MARCO Email: antinisca.dimarco@univaq.it Analisi Filogenetica Gene Ancestrale duplicazione genica La filogenesi è lo studio delle relazioni evolutive tra
Corso di Bioinformatica Docente: Dr. Antinisca DI MARCO Email: antinisca.dimarco@univaq.it Analisi Filogenetica Gene Ancestrale duplicazione genica La filogenesi è lo studio delle relazioni evolutive tra
COME CALCOLARE IL PUNTEGGIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi
 COME CALCOLARE IL PUNTEGGIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
COME CALCOLARE IL PUNTEGGIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
Metodi di Distanza. G.Allegrucci riproduzione vietata
 Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
Ricevimento Studenti: Lunedì previa prenotazione. Cenci lab
 Cenci lab Giovanni Cenci Dip.to Biologia e Biotecnologie C. Darwin Sezione Genetica Piano 2 -Citofono 3/4 0649912-655 (office) 0649912-843 (lab) giovanni.cenci@uniroma1.it Ricevimento Studenti: Lunedì
Cenci lab Giovanni Cenci Dip.to Biologia e Biotecnologie C. Darwin Sezione Genetica Piano 2 -Citofono 3/4 0649912-655 (office) 0649912-843 (lab) giovanni.cenci@uniroma1.it Ricevimento Studenti: Lunedì
Lezione 12. Origine degli introni
 Lezione 12 Origine degli introni Nature Reviews Genetics 2006 Introni di gruppo I batteri, organelli identificati circa 1500 Introni di gruppo II identificati circa 200 Introni con spliceosomi genoma nucleare
Lezione 12 Origine degli introni Nature Reviews Genetics 2006 Introni di gruppo I batteri, organelli identificati circa 1500 Introni di gruppo II identificati circa 200 Introni con spliceosomi genoma nucleare
BLAST: Basic Local Alignment Search Tool
 BLAST: Basic Local Alignment Search Tool 1 Outline della lezione di oggi BLAST Uso pratico Algoritmo Strategie Trovare proteine lontanamente legate: PSI-BLAST 2 Problema con gli algoritmi dinamici Gli
BLAST: Basic Local Alignment Search Tool 1 Outline della lezione di oggi BLAST Uso pratico Algoritmo Strategie Trovare proteine lontanamente legate: PSI-BLAST 2 Problema con gli algoritmi dinamici Gli
Riconoscimento e recupero dell informazione per bioinformatica
 Riconoscimento e recupero dell informazione per bioinformatica Filogenesi Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Introduzione alla
Riconoscimento e recupero dell informazione per bioinformatica Filogenesi Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Introduzione alla
FORMAZIONE DEL LEGAME PEPTIDICO
 AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
RELAZIONE di BIOLOGIA MOLECOLARE
 NOME: Marini Selena MATRICOLA: 592330 RELAZIONE di BIOLOGIA MOLECOLARE CHE ORGANISMO MODELLO È DICTYOSTELIUM? CHE RISORSE BIOINFORMATICHE AGEVOLANO I RICERCATORI CHE LO STUDIANO? Dictyostelium è un genere
NOME: Marini Selena MATRICOLA: 592330 RELAZIONE di BIOLOGIA MOLECOLARE CHE ORGANISMO MODELLO È DICTYOSTELIUM? CHE RISORSE BIOINFORMATICHE AGEVOLANO I RICERCATORI CHE LO STUDIANO? Dictyostelium è un genere
Perché considerare la struttura 3D di una proteina
 Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
ESERCITAZIONE 3. OBIETTIVO: Ricerca di omologhe mediante i programmi FASTA e BLAST
 ESERCITAZIONE 3 OBIETTIVO: Ricerca di omologhe mediante i programmi FASTA e BLAST L'esercitazione prevede l'utilizzo di risorse web per effettuare ricerche di similarità con la proteina GRB2 (growth factor
ESERCITAZIONE 3 OBIETTIVO: Ricerca di omologhe mediante i programmi FASTA e BLAST L'esercitazione prevede l'utilizzo di risorse web per effettuare ricerche di similarità con la proteina GRB2 (growth factor
LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI
 CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
Strategie di annotazione di geni e genomi
 Strategie di annotazione di geni e genomi Dr. Giovanni Emiliani giovanni.emiliani@unifi.it Bioinformatica A.A. 2011-1012 Concetti generali Le nuove tecnologie consentono l ottenimento di una grande mole
Strategie di annotazione di geni e genomi Dr. Giovanni Emiliani giovanni.emiliani@unifi.it Bioinformatica A.A. 2011-1012 Concetti generali Le nuove tecnologie consentono l ottenimento di una grande mole
Le L z e io i ne n 6 Co C n o f n ro r n o t n i i fra r a se s q e u q e u n e z n e z : e di d s i t s a t nz n e z, e allineamenti
 Lezione 6 Confronti fra sequenze: distanze, Confronti fra sequenze: distanze, allineamenti Distanze fra sequenze Per N siti ed n differenze: grado di divergenza = n/n AATGAAAGAA 10 siti; 3 differenze ACTGGAGGAA
Lezione 6 Confronti fra sequenze: distanze, Confronti fra sequenze: distanze, allineamenti Distanze fra sequenze Per N siti ed n differenze: grado di divergenza = n/n AATGAAAGAA 10 siti; 3 differenze ACTGGAGGAA
UNIVERSITÀ DEGLI STUDI DI MILANO. Bioinformatica. A.A semestre II. 4 Evoluzione e filogenesi
 Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre II 4 Evoluzione e filogenesi FILOGENETICA CS Definzione Studio delle relazioni evolutive tra
Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre II 4 Evoluzione e filogenesi FILOGENETICA CS Definzione Studio delle relazioni evolutive tra
ALLINEAMENTO DI SEQUENZE
 ALLINEAMENTO DI SEQUENZE 1 DATABASE DI SEQUENZE RICERCA TESTUALE Ricerca dei record i cui campi soddisfano determinati criteri (hanno certi valori) Abbiamo già visto nelle lezioni precedenti SIMILARITA
ALLINEAMENTO DI SEQUENZE 1 DATABASE DI SEQUENZE RICERCA TESTUALE Ricerca dei record i cui campi soddisfano determinati criteri (hanno certi valori) Abbiamo già visto nelle lezioni precedenti SIMILARITA
IL CODICE GENETICO E I CARATTERI EREDITARI
 IL CODICE GENETICO E I CARATTERI EREDITARI Il DNA porta le informazioni genetiche scritte nella sequenza di basi. Qualunque sequenza è possibile. Il DNA virus più semplici: 5000 basi appaiate; 46 cromosomi
IL CODICE GENETICO E I CARATTERI EREDITARI Il DNA porta le informazioni genetiche scritte nella sequenza di basi. Qualunque sequenza è possibile. Il DNA virus più semplici: 5000 basi appaiate; 46 cromosomi
Vai al sito: Incolla nel box vuoto la sequenza nucleotidica
 Identificare il gene a cui appartiene la sequenza (sonda) e la sua posizione sul cromosoma. Per raggiungere l obiettivo della prima parte dell attività devi usare il software BLAT (BLAST- Like Alignment
Identificare il gene a cui appartiene la sequenza (sonda) e la sua posizione sul cromosoma. Per raggiungere l obiettivo della prima parte dell attività devi usare il software BLAT (BLAST- Like Alignment
La mappatura dei geni umani. SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione
 La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
Bioinformatica (1) Introduzione. Dott. Alessandro Laganà
 Bioinformatica (1) Introduzione Dott. Alessandro Laganà Dott. Alessandro Laganà Martedi 15.30 16.30 Studio Assegnisti - 1 Piano (Davanti biblioteca) Dipartimento di Matematica e Informatica (Città Universitaria)
Bioinformatica (1) Introduzione Dott. Alessandro Laganà Dott. Alessandro Laganà Martedi 15.30 16.30 Studio Assegnisti - 1 Piano (Davanti biblioteca) Dipartimento di Matematica e Informatica (Città Universitaria)
Introduzione al Calcolo Scientifico A.A Lab. 11
 Introduzione al Calcolo Scientifico A.A. 2009-2010 - Lab. 11 Si consideri il problema dell allineamento di sequenze di proteine in biologia, legato per esempio all annotamento di genomi Si realizzi con
Introduzione al Calcolo Scientifico A.A. 2009-2010 - Lab. 11 Si consideri il problema dell allineamento di sequenze di proteine in biologia, legato per esempio all annotamento di genomi Si realizzi con
Misure di diversità tra unità statistiche. Loredana Cerbara
 Misure di diversità tra unità statistiche Loredana Cerbara LA DISTANZA IN STATISTICA In statistica la distanza ha un significato diverso da quello che si può intuire in altre discipline, dove, peraltro,
Misure di diversità tra unità statistiche Loredana Cerbara LA DISTANZA IN STATISTICA In statistica la distanza ha un significato diverso da quello che si può intuire in altre discipline, dove, peraltro,
Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016. Docente: Silvia Fuselli fss@unife.it
 Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016 Docente: Silvia Fuselli fss@unife.it Possibili testi di riferimento Introduction to Genomics, A.M. Lesk, Oxford Capitoli 1, 3,
Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016 Docente: Silvia Fuselli fss@unife.it Possibili testi di riferimento Introduction to Genomics, A.M. Lesk, Oxford Capitoli 1, 3,
Sistemi di numerazione
 Sistemi di numerazione Introduzione Un sistema di numerazione è un sistema utilizzato per esprimere i numeri e possibilmente alcune operazioni che si possono effettuare su di essi. Storicamente i sistemi
Sistemi di numerazione Introduzione Un sistema di numerazione è un sistema utilizzato per esprimere i numeri e possibilmente alcune operazioni che si possono effettuare su di essi. Storicamente i sistemi
19/09/14. Il codice ASCII. Altri codici importanti. Extended ASCII. Tabella del codice ASCII a 7 bit. Prof. Daniele Gorla
 Il codice ASCII ASCII è un acronimo per American Standard Code for Information Interchange Nato nell IBM nel 1961, diventa standard ISO (International Organization for Standardization) nel 1968. Codifica
Il codice ASCII ASCII è un acronimo per American Standard Code for Information Interchange Nato nell IBM nel 1961, diventa standard ISO (International Organization for Standardization) nel 1968. Codifica
3. Confronto tra due sequenze
 3. Confronto tra due sequenze Esercizio 1: uso di DotLet Il programma DotLet è accessibile dal sito http://myhits.isb-sib.ch/cgi-bin/dotlet, dove può essere utilizzato attraverso un interfaccia utente
3. Confronto tra due sequenze Esercizio 1: uso di DotLet Il programma DotLet è accessibile dal sito http://myhits.isb-sib.ch/cgi-bin/dotlet, dove può essere utilizzato attraverso un interfaccia utente
Laboratorio di Bioinformatica I. Parte 2. Dott. Sergio Marin Vargas (2014 / 2015)
 Laboratorio di Bioinformatica I Banche dati Parte 2 Dott. Sergio Marin Vargas (2014 / 2015) Google Scholar https://scholar.google.it/ E un motore di ricerca di Google, specializzato nella ricerca di articoli
Laboratorio di Bioinformatica I Banche dati Parte 2 Dott. Sergio Marin Vargas (2014 / 2015) Google Scholar https://scholar.google.it/ E un motore di ricerca di Google, specializzato nella ricerca di articoli
Legami chimici. Covalente. Legami deboli
 Legami chimici Covalente Legami deboli Legame fosfodiesterico Legami deboli Legami idrogeno Interazioni idrofobiche Attrazioni di Van der Waals Legami ionici STRUTTURA TERZIARIA La struttura tridimensionale
Legami chimici Covalente Legami deboli Legame fosfodiesterico Legami deboli Legami idrogeno Interazioni idrofobiche Attrazioni di Van der Waals Legami ionici STRUTTURA TERZIARIA La struttura tridimensionale
Ricerca di omologhi. La sequenza di cui vogliamo trovare gli omologhi viene de6a query.
 Ricerca di omologhi La sequenza di cui vogliamo trovare gli omologhi viene de6a query. Dobbiamo cercare i suoi omologhi in una banca da= di sequenze (qualche decina di milioni) Allineamento con ciascuna
Ricerca di omologhi La sequenza di cui vogliamo trovare gli omologhi viene de6a query. Dobbiamo cercare i suoi omologhi in una banca da= di sequenze (qualche decina di milioni) Allineamento con ciascuna
Programmazione dinamica
 Programmazione dinamica Violetta Lonati Università degli studi di Milano Dipartimento di Informatica Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Violetta Lonati Programmazione
Programmazione dinamica Violetta Lonati Università degli studi di Milano Dipartimento di Informatica Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Violetta Lonati Programmazione
Il processo inferenziale consente di generalizzare, con un certo grado di sicurezza, i risultati ottenuti osservando uno o più campioni
 La statistica inferenziale Il processo inferenziale consente di generalizzare, con un certo grado di sicurezza, i risultati ottenuti osservando uno o più campioni E necessario però anche aggiungere con
La statistica inferenziale Il processo inferenziale consente di generalizzare, con un certo grado di sicurezza, i risultati ottenuti osservando uno o più campioni E necessario però anche aggiungere con
Edit distance. v intner RIMDMDMMI wri t ers
 L'allineamento Edit distance Le operazioni permesse sono: I: insert (inserimento, inserzione) D: delete (cancellazione, delezione, rimozione) R: replacement (substition, sostituzione) M: match (corrispondenza,
L'allineamento Edit distance Le operazioni permesse sono: I: insert (inserimento, inserzione) D: delete (cancellazione, delezione, rimozione) R: replacement (substition, sostituzione) M: match (corrispondenza,
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
I MOTORI DELL EVOLUZIONE PT6. POMERIGGIO DI AGGIORNAMENTO PROF. M.A. ZORDAN, Ph.D UNIVERSITÀ DEGLI STUDI DI PADOVA
 I MOTORI DELL EVOLUZIONE PT6 POMERIGGIO DI AGGIORNAMENTO 23.03.2011 PROF. M.A. ZORDAN, Ph.D UNIVERSITÀ DEGLI STUDI DI PADOVA 1 EFFETTO MATERNO 2 Effetto materno Si definisce effetto materno la modalità
I MOTORI DELL EVOLUZIONE PT6 POMERIGGIO DI AGGIORNAMENTO 23.03.2011 PROF. M.A. ZORDAN, Ph.D UNIVERSITÀ DEGLI STUDI DI PADOVA 1 EFFETTO MATERNO 2 Effetto materno Si definisce effetto materno la modalità
I.4 Rappresentazione dell informazione
 I.4 Rappresentazione dell informazione Università di Ferrara Dipartimento di Economia e Management Insegnamento di Informatica Ottobre 13, 2015 Argomenti Introduzione 1 Introduzione 2 3 L elaboratore Introduzione
I.4 Rappresentazione dell informazione Università di Ferrara Dipartimento di Economia e Management Insegnamento di Informatica Ottobre 13, 2015 Argomenti Introduzione 1 Introduzione 2 3 L elaboratore Introduzione
LA SINTESI PROTEICA LE MOLECOLE CHE INTERVENGONO IN TALE PROCESSO SONO:
 LA SINTESI PROTEICA La sintesi proteica è il processo che porta alla formazione delle proteine utilizzando le informazioni contenute nel DNA. Nelle sue linee fondamentali questo processo è identico in
LA SINTESI PROTEICA La sintesi proteica è il processo che porta alla formazione delle proteine utilizzando le informazioni contenute nel DNA. Nelle sue linee fondamentali questo processo è identico in
Laboratorio di Elementi di Bioinformatica
 Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2016/2017 Formato GTF per annotare un gene Docente del laboratorio: Raffaella Rizzi 1 GTF (Gene Transfer
Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2016/2017 Formato GTF per annotare un gene Docente del laboratorio: Raffaella Rizzi 1 GTF (Gene Transfer
Riarrangiamento genico
 Riarrangiamento genico 1 3 quesiti per la comprensione Ø l esistenza nello stesso anticorpo di una parte variabile ed una costante; Ø l esistenza della enorme variabilità (diversità) del sito combinatorio;
Riarrangiamento genico 1 3 quesiti per la comprensione Ø l esistenza nello stesso anticorpo di una parte variabile ed una costante; Ø l esistenza della enorme variabilità (diversità) del sito combinatorio;
Ingegneria della Conoscenza e Sistemi Esperti Lezione 9: Evolutionary Computation
 Ingegneria della Conoscenza e Sistemi Esperti Lezione 9: Evolutionary Computation Dipartimento di Elettronica e Informazione Politecnico di Milano Evolutionary Computation Raggruppa modelli di calcolo
Ingegneria della Conoscenza e Sistemi Esperti Lezione 9: Evolutionary Computation Dipartimento di Elettronica e Informazione Politecnico di Milano Evolutionary Computation Raggruppa modelli di calcolo
Il Codice Gene,co. Il dogma centrale, il flusso dell informazione genica e la decifrazione della informazione del DNA
 Corso di Laurea in Chimica e Tecnologie Farmaceu,che a.a. 2014-2015 Università di Catania Il Codice Gene,co Il dogma centrale, il flusso dell informazione genica e la decifrazione della informazione del
Corso di Laurea in Chimica e Tecnologie Farmaceu,che a.a. 2014-2015 Università di Catania Il Codice Gene,co Il dogma centrale, il flusso dell informazione genica e la decifrazione della informazione del
Matrici di Raven (PM47)
 Matrici di Raven (PM47) Matrici di Raven (PM47) Matrici di Raven (PM47) Matrici di Raven (PM38) TRA I TEST DI TIPICA PERFORMANCE Test proiettivi Test self-report di personalità Questionari psichiatrici
Matrici di Raven (PM47) Matrici di Raven (PM47) Matrici di Raven (PM47) Matrici di Raven (PM38) TRA I TEST DI TIPICA PERFORMANCE Test proiettivi Test self-report di personalità Questionari psichiatrici
Nucleotide / aminoacido? 4 aa. 2 Nucleotide / aminoacido? 4 2 = 16 aa. 3 Nucleotide / aminoacido? 4 3 = 64 aa. UNIVERSALE e DEGENERATO
 La#scoperta#della#stru.ura#a#doppia#elica# nel#1953#ha#fa.o#immediatamente# sorgere#una#domanda:## come#l informazione#gene;ca#può#essere# codificata#dal#dna?# Nucleotide / aminoacido? 4 aa 2 Nucleotide
La#scoperta#della#stru.ura#a#doppia#elica# nel#1953#ha#fa.o#immediatamente# sorgere#una#domanda:## come#l informazione#gene;ca#può#essere# codificata#dal#dna?# Nucleotide / aminoacido? 4 aa 2 Nucleotide
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
Rapida Nota sulla Rappresentazione dei Caratteri
 TECNOLOGIA DIGITALE TECNOLOGIA DIGITALE (segue) CPU, memoria centrale e dispositivi sono realizzati con tecnologia elettronica digitale Dati ed operazioni vengono codificati tramite sequenze di bit 8 bit
TECNOLOGIA DIGITALE TECNOLOGIA DIGITALE (segue) CPU, memoria centrale e dispositivi sono realizzati con tecnologia elettronica digitale Dati ed operazioni vengono codificati tramite sequenze di bit 8 bit
Valutazione dei test diagnostici
 Valutazione dei test diagnostici Maria Miceli M. Miceli 2011 1 Diagnosi individuale (test di laboratorio) Esame collaterale nell ambito dell iter diagnostico condotto generalmente su animali sintomatici
Valutazione dei test diagnostici Maria Miceli M. Miceli 2011 1 Diagnosi individuale (test di laboratorio) Esame collaterale nell ambito dell iter diagnostico condotto generalmente su animali sintomatici
Corso di Elementi di Bionformatica
 Corso di Elementi di Bionformatica Laurea Triennale in Informatica Il formato FASTQ per la qualità delle sequenze Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 La qualità delle sequenze
Corso di Elementi di Bionformatica Laurea Triennale in Informatica Il formato FASTQ per la qualità delle sequenze Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 La qualità delle sequenze
Probabilità congiunta
 Una vera matrice di sostituzione F K M N P Q 4 5 5 4 3 3 5 6 5 4 3 5 4 4 3 6 4 3 4 5 3 F 8 5 5 4 5 5 4 5 3 4 3 3 6 3 5 K 5 3 3 6 4 3 3 3 M 6 N P 6 Q 4 6 V Y 3 3 3 3 V 4 4 6 8 6 6 6 3 5 3 4 4 6 5 5 6 7
Una vera matrice di sostituzione F K M N P Q 4 5 5 4 3 3 5 6 5 4 3 5 4 4 3 6 4 3 4 5 3 F 8 5 5 4 5 5 4 5 3 4 3 3 6 3 5 K 5 3 3 6 4 3 3 3 M 6 N P 6 Q 4 6 V Y 3 3 3 3 V 4 4 6 8 6 6 6 3 5 3 4 4 6 5 5 6 7
FISICA. Elaborazione dei dati sperimentali. Autore: prof. Pappalardo Vincenzo docente di Matematica e Fisica
 FISICA Elaborazione dei dati sperimentali Autore: prof. Pappalardo Vincenzo docente di Matematica e Fisica LA MISURA GLI STRUMENTI DI MISURA Gli strumenti di misura possono essere analogici o digitali.
FISICA Elaborazione dei dati sperimentali Autore: prof. Pappalardo Vincenzo docente di Matematica e Fisica LA MISURA GLI STRUMENTI DI MISURA Gli strumenti di misura possono essere analogici o digitali.
Relatrice: dott.ssa Ilaria Pegoretti
 Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
Rappresentazione dell informazione
 Rappresentazione dell informazione Problema che coinvolge aspetti filosofici Interessa soprattutto distinguere informazioni diverse Con un solo simbolo è impossibile Pertanto l insieme minimo è costituito
Rappresentazione dell informazione Problema che coinvolge aspetti filosofici Interessa soprattutto distinguere informazioni diverse Con un solo simbolo è impossibile Pertanto l insieme minimo è costituito
Distanza di Edit. Speaker: Antinisca Di Marco Data:
 Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
CHEMIOMETRIA. CONFRONTO CON VALORE ATTESO (test d ipotesi) CONFRONTO DI VALORI MISURATI (test d ipotesi) CONFRONTO DI RIPRODUCIBILITA (test d ipotesi)
 CHEMIOMETRIA Applicazione di metodi matematici e statistici per estrarre (massima) informazione chimica (affidabile) da dati chimici INCERTEZZA DI MISURA (intervallo di confidenza/fiducia) CONFRONTO CON
CHEMIOMETRIA Applicazione di metodi matematici e statistici per estrarre (massima) informazione chimica (affidabile) da dati chimici INCERTEZZA DI MISURA (intervallo di confidenza/fiducia) CONFRONTO CON
Traguardi per lo sviluppo delle competenze. Obiettivi operativi. essenziali
 Traguardi per lo sviluppo delle competenze Nuclei tematici Obiettivi d apprendimento essenziali Obiettivi operativi Sviluppa un atteggiamento positivo rispetto alla matematica, attraverso esperienze significative,
Traguardi per lo sviluppo delle competenze Nuclei tematici Obiettivi d apprendimento essenziali Obiettivi operativi Sviluppa un atteggiamento positivo rispetto alla matematica, attraverso esperienze significative,
In alternativa all intervallo di riferimento. VALORI o LIVELLI DECISIONALI
 In alternativa all intervallo di riferimento VALORI o LIVELLI DECISIONALI Valori sopra o sotto i quali è raccomandabile seguire un determinato comportamento clinico: - Instaurare o modificare un regime
In alternativa all intervallo di riferimento VALORI o LIVELLI DECISIONALI Valori sopra o sotto i quali è raccomandabile seguire un determinato comportamento clinico: - Instaurare o modificare un regime
Allineamento di sequenze di DNA e proteine: possibilità, limiti ed interpretazione
 ATTENZIONE: lo sfondo giallo NON riguarda parti più importanti ma evidenzia solo parti di testo (frasi, paragrafi) aggiornate (correzioni, miglioramento dello stile, o aggiunta di note o esempi per chiarire
ATTENZIONE: lo sfondo giallo NON riguarda parti più importanti ma evidenzia solo parti di testo (frasi, paragrafi) aggiornate (correzioni, miglioramento dello stile, o aggiunta di note o esempi per chiarire
Introduzione all analisi di arrays: clustering.
 Statistica per la Ricerca Sperimentale Introduzione all analisi di arrays: clustering. Lezione 2-14 Marzo 2006 Stefano Moretti Dipartimento di Matematica, Università di Genova e Unità di Epidemiologia
Statistica per la Ricerca Sperimentale Introduzione all analisi di arrays: clustering. Lezione 2-14 Marzo 2006 Stefano Moretti Dipartimento di Matematica, Università di Genova e Unità di Epidemiologia
Il nucleo compartimento nucleare involucro nucleare la cromatina (DNA + proteine), uno
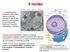 Il nucleo Il compartimento nucleare, tipico delle cellule eucariote, segrega le attività del genoma (replicazione e trascrizione del DNA) dal rimanente metabolismo cellulare Il confine del compartimento
Il nucleo Il compartimento nucleare, tipico delle cellule eucariote, segrega le attività del genoma (replicazione e trascrizione del DNA) dal rimanente metabolismo cellulare Il confine del compartimento
FILE E INDICI Architettura DBMS
 FILE E INDICI Architettura DBMS Giorgio Giacinto 2010 Database 2 Dati su dispositivi di memorizzazione esterni! Dischi! si può leggere qualunque pagina a costo medio fisso! Nastri! si possono leggere le
FILE E INDICI Architettura DBMS Giorgio Giacinto 2010 Database 2 Dati su dispositivi di memorizzazione esterni! Dischi! si può leggere qualunque pagina a costo medio fisso! Nastri! si possono leggere le
Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata Via Belmeloro, 8 Bologna
 GENETICA GENERALE - 1 CFU Modulo Biologia Applicata e Genetica generale CORSO INTEGRATO: SCIENZE BIOLOGICHE - 7 CFU Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata
GENETICA GENERALE - 1 CFU Modulo Biologia Applicata e Genetica generale CORSO INTEGRATO: SCIENZE BIOLOGICHE - 7 CFU Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata
VALORI o LIVELLI DECISIONALI
 In alternativa all intervallo di riferimento VALORI o LIVELLI DECISIONALI Valori sopra o sotto i quali è raccomandabile seguire un determinato comportamento clinico: - Instaurare o modificare un regime
In alternativa all intervallo di riferimento VALORI o LIVELLI DECISIONALI Valori sopra o sotto i quali è raccomandabile seguire un determinato comportamento clinico: - Instaurare o modificare un regime
18/05/2014. Università di Ferrara Corso di Ingegneria del Software AA 2013/2014
 Università di Ferrara Corso di Ingegneria del Software AA 2013/2014 La misura come strumento scientifico Misure, metriche e indicatori Metriche del software Metriche per software orientato agli oggetti
Università di Ferrara Corso di Ingegneria del Software AA 2013/2014 La misura come strumento scientifico Misure, metriche e indicatori Metriche del software Metriche per software orientato agli oggetti
LEZIONE 4. { x + y + z = 1 x y + 2z = 3
 LEZIONE 4 4.. Operazioni elementari di riga. Abbiamo visto, nella precedente lezione, quanto sia semplice risolvere sistemi di equazioni lineari aventi matrice incompleta fortemente ridotta per righe.
LEZIONE 4 4.. Operazioni elementari di riga. Abbiamo visto, nella precedente lezione, quanto sia semplice risolvere sistemi di equazioni lineari aventi matrice incompleta fortemente ridotta per righe.
Immunologia e Immunologia Diagnostica MATURAZIONE DEI LINFOCITI
 Immunologia e Immunologia Diagnostica MATURAZIONE DEI LINFOCITI Il percorso di maturazione dei linfociti Sviluppo della specicifità immunologica I linfociti B e T avviano le risposte immunitarie dopo il
Immunologia e Immunologia Diagnostica MATURAZIONE DEI LINFOCITI Il percorso di maturazione dei linfociti Sviluppo della specicifità immunologica I linfociti B e T avviano le risposte immunitarie dopo il
Lezioni di economia monetaria e creditizia
 Lezioni di economia monetaria e creditizia La curva di Phillips e il modello di Lucas Fabrizio Mattesini Università di Roma "Tor Vergata" May 12, 2014 abrizio Mattesini (Università di Roma "Tor Vergata")
Lezioni di economia monetaria e creditizia La curva di Phillips e il modello di Lucas Fabrizio Mattesini Università di Roma "Tor Vergata" May 12, 2014 abrizio Mattesini (Università di Roma "Tor Vergata")
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
Esempio 1 Si consideri il seguente gioco in forma estesa:
 Best reply: strategie pure e miste c Fioravante Patrone Esempio Si consideri il seguente gioco in forma estesa: 5 T L R L R 4 4 B T B a) scriverne la forma strategica; b) determinarne gli equilibri di
Best reply: strategie pure e miste c Fioravante Patrone Esempio Si consideri il seguente gioco in forma estesa: 5 T L R L R 4 4 B T B a) scriverne la forma strategica; b) determinarne gli equilibri di
Modelli e Metodi per la Simulazione (MMS)
 Modelli e Metodi per la Simulazione (MMS) adacher@dia.uniroma3.it Programma La simulazione ad eventi discreti, è una metodologia fondamentale per la valutazione delle prestazioni di sistemi complessi (di
Modelli e Metodi per la Simulazione (MMS) adacher@dia.uniroma3.it Programma La simulazione ad eventi discreti, è una metodologia fondamentale per la valutazione delle prestazioni di sistemi complessi (di
Genomica, proteomica, genomica strutturale, banche dati.
 Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Mutazioni genetiche 2
 Mutazioni genetiche 2 Cosa sono le mutazioni? Le proteine sono in grado di svolgere la loro funzione solo se la loro sequenza amminoacidica è quella corretta. In caso contrario si possono generare delle
Mutazioni genetiche 2 Cosa sono le mutazioni? Le proteine sono in grado di svolgere la loro funzione solo se la loro sequenza amminoacidica è quella corretta. In caso contrario si possono generare delle
La simulazione è l'imitazione di un processo o di un sistema reale per un
 1 2 La simulazione è l'imitazione di un processo o di un sistema reale per un determinato periodo di tempo. La simulazione è l'imitazione di un processo o di un sistema reale per un determinato periodo
1 2 La simulazione è l'imitazione di un processo o di un sistema reale per un determinato periodo di tempo. La simulazione è l'imitazione di un processo o di un sistema reale per un determinato periodo
Cercare il percorso minimo Ant Colony Optimization
 Cercare il percorso minimo Ant Colony Optimization Author: Luca Albergante 1 Dipartimento di Matematica, Università degli Studi di Milano 4 Aprile 2011 L. Albergante (Univ. of Milan) PSO 4 Aprile 2011
Cercare il percorso minimo Ant Colony Optimization Author: Luca Albergante 1 Dipartimento di Matematica, Università degli Studi di Milano 4 Aprile 2011 L. Albergante (Univ. of Milan) PSO 4 Aprile 2011
Appunti su Indipendenza Lineare di Vettori
 Appunti su Indipendenza Lineare di Vettori Claudia Fassino a.a. Queste dispense, relative a una parte del corso di Matematica Computazionale (Laurea in Informatica), rappresentano solo un aiuto per lo
Appunti su Indipendenza Lineare di Vettori Claudia Fassino a.a. Queste dispense, relative a una parte del corso di Matematica Computazionale (Laurea in Informatica), rappresentano solo un aiuto per lo
Markov Chains and Markov Chain Monte Carlo (MCMC)
 Markov Chains and Markov Chain Monte Carlo (MCMC) Alberto Garfagnini Università degli studi di Padova December 11, 2013 Catene di Markov Discrete dato un valore x t del sistema ad un istante di tempo fissato,
Markov Chains and Markov Chain Monte Carlo (MCMC) Alberto Garfagnini Università degli studi di Padova December 11, 2013 Catene di Markov Discrete dato un valore x t del sistema ad un istante di tempo fissato,
La struttura elettronica degli atomi
 1 In unità atomiche: a 0 me 0,59A unità di lunghezza e H 7, ev a H=Hartree unità di energia L energia dell atomo di idrogeno nello stato fondamentale espresso in unità atomiche è: 4 0 me 1 e 1 E H 13,
1 In unità atomiche: a 0 me 0,59A unità di lunghezza e H 7, ev a H=Hartree unità di energia L energia dell atomo di idrogeno nello stato fondamentale espresso in unità atomiche è: 4 0 me 1 e 1 E H 13,
Ingegneria della Conoscenza e Sistemi Esperti Lezione 5: Regole di Decisione
 Ingegneria della Conoscenza e Sistemi Esperti Lezione 5: Regole di Decisione Dipartimento di Elettronica e Informazione Politecnico di Milano Perchè le Regole? Le regole (if-then) sono espressive e leggibili
Ingegneria della Conoscenza e Sistemi Esperti Lezione 5: Regole di Decisione Dipartimento di Elettronica e Informazione Politecnico di Milano Perchè le Regole? Le regole (if-then) sono espressive e leggibili
Teoria dell informazione
 Corso di Laurea a Distanza in Ingegneria Elettrica Corso di Comunicazioni Elettriche Teoria dell informazione A.A. 2008-09 Alberto Perotti DELEN-DAUIN Modello di sistema di comunicazione Il modello di
Corso di Laurea a Distanza in Ingegneria Elettrica Corso di Comunicazioni Elettriche Teoria dell informazione A.A. 2008-09 Alberto Perotti DELEN-DAUIN Modello di sistema di comunicazione Il modello di
La funzione Hash. Garanzia dell integrità dei dati e autenticazione dei messaggi
 La funzione Hash Garanzia dell integrità dei dati e autenticazione dei messaggi Come funziona l Hash function Associa stringhe di bit di lunghezza arbitraria e finita stringhe di bit di lunghezza inferiore.
La funzione Hash Garanzia dell integrità dei dati e autenticazione dei messaggi Come funziona l Hash function Associa stringhe di bit di lunghezza arbitraria e finita stringhe di bit di lunghezza inferiore.
Fogli Elettronici: MS Excel
 Fogli Elettronici: MS Excel Foglio Elettronico Un foglio elettronico (o spreadsheet) è un software applicativo nato dall esigenza di: organizzare insiemi di dati tramite tabelle, schemi, grafici, etc.
Fogli Elettronici: MS Excel Foglio Elettronico Un foglio elettronico (o spreadsheet) è un software applicativo nato dall esigenza di: organizzare insiemi di dati tramite tabelle, schemi, grafici, etc.
λ è detto intensità e rappresenta il numero di eventi che si
 ESERCITAZIONE N 1 STUDIO DI UN SISTEMA DI CODA M/M/1 1. Introduzione Per poter studiare un sistema di coda occorre necessariamente simulare gli arrivi, le partenze e i tempi di ingresso nel sistema e di
ESERCITAZIONE N 1 STUDIO DI UN SISTEMA DI CODA M/M/1 1. Introduzione Per poter studiare un sistema di coda occorre necessariamente simulare gli arrivi, le partenze e i tempi di ingresso nel sistema e di
