PCR (Polymerase chain reaction)
|
|
|
- Nicoletta Massari
- 7 anni fa
- Visualizzazioni
Transcript
1 PCR (Polymerase chain reaction) La PCR è un metodo di biologia molecolare che perette di amplificare una regione specifica di DNA mediante cicli ripetuti di replicazione a temperature differenti. E una tecnica molto specifica e sensibile. I principali componenti della PCR sono: - Double strand DNA, che contiene il frammento da amplificare; - Due primers (Forward e Reverse), che determinano l inizio e la fine della regione da amplificare. I primer di solito hanno una lunghezza tra 18 e 25 bp, dovrebbero avere un contenuto di G+C non inferiore al 50-60% e non devono avere una sequenza complementare per evitare che si appaino (dimerizzino) tra loro. Il Forward ha una sequenza identica alla sequenza 5-3 del frammento da amplificare. Il reverse invece, ha una sequenza complementare, cioè è costituito da una sequenza di basi complementari a quelle della sequenza target e con una direzione opposta DNA Polimerasi, che copia la regione da amplificare (si usano le DNA polimerasi di batteri termofili, che sono stabili ad elevate temperature; es. Taq Polimerasi isolata dal batterio Thermofilus aquaticus); - Nucleotidi (dntps deossiribonucleotidi trifosfati), forniscono le basi nucleotidiche per la sintesi di nuovo DNA; - Buffer composto da Tris-HCL, MgCL 2. che fornisce l ambiente chimico ideale per la DNA Polimerasi. Il Tris-HCl tampona il ph della reazione, mentre gli ioni Mg 2+ sono cofattori fondamentali per l attività della polimerasi. Il processo della PCR consta di tre steps principali che vengono ripetuti dalle 30 alle 40 volte, nella stessa provetta attraverso l utilizzo di un termociclatore. 1). Denaturazione del frammento che avviene a C, temperatura in grado di rompere i legami idrogeno che uniscono le due eliche di DNA 2). Ibridazione o Annealing a C: Ciascun primer si appaia all estremità 3 del frammento del DNA stampo, definendo il tratto da amplificare. 3). Estensione a C (Elongation): La temperatura di elongation dipende dalla DNA Polimerasi. Durante l estensione, la Polimerasi sintetizza un nuovo filamento di DNA in direzione 5-3 a partire dall estremità libera del primer appaiato alla sequenza bersaglio. Il risultato sono 2 copie di DNA a doppio filamento. Poiché il DNA sintetizzato ad ogni ciclo funge da stampo per la sintesi del ciclo successivo, la
2 successione dei cicli comporta un aumento esponenziale del numero delle copie di DNA. Il DNA amplificato viene sottoposto a corsa elettroforetica Volt in gel di agarosio. L amplificazione di geni target è definita da tre fasi: Fase esponenziale (tutti i reagenti sono presenti) Fase lineare (alcuni reagenti iniziano a terminare) Plateau (tutti reagenti terminano) Il processo di duplicazione non procede all infinito, esso è limitato da: - Quantità dei primers primers, dntp, MgCl2, Buffer - Attività della Taq polimerasi polimerasi - Reannealing dei filamenti dei filamenti Raggiunto il plateau non si osserva più un incremento nei prodotti VANTAGGI: - Sensibilità e rapidità - Analisi simultanea di molti campioni - Analisi simultanea di diverse sequenze sullo stesso campione - Analisi di DNA degradato o incluso in mezzi strani, o fissato. SVANTAGGI: - Sensibilità (rischio di contaminazioni-falsi positivi) - Efficienza variabile di amplificazione a seconda della sequenza - Richiede conoscenza di base delle sequenze da amplificare e messa a punto delle coppie di oligonucleotidi di innesco (primers) Può sintetizzare frammenti relativamente corti (< 5000 bp). - la sintesi è imprecisa e può introdurre errori nella sequenza (la Taq pol non possiede attività 3 ->5 esonucleasica) La PCR può essere utilizzata in un ampia varietà di analisi tra cui: - Genetic fingerprinting (usata in medicina legale) - Test di paternità; - Studi di evoluzione e filogenesi;
3 - Identificazione di malattie ereditarie; - Identificazione di malattie virali; - Clonaggio di un gene; - Analisi di DNA antico; - Genotipizzazione di specifiche mutazioni e polimorfismi. Il campione si sottopone al numero di cicli stabiliti, alla fine della reazione un aliquota si sottopone ad elettroforesi su gel agorosio per verificare: 1. la lunghezza dell amplificato (controllo del peso la lunghezza dell amplificato (controllo del peso molecolare in bp mediante ladder) 2. L assenza di frammenti aspecifici 3. La quantità di amplificato ottenuto (proporzionale all intensità della banda) ELETTROFORESI in gel di Agarosio: L elettroforesi in gel di agarosio consente di separare molecole cariche (DNA, RNA, proteine) grazie all applicazione di un campo elettrico, in base alle dimensioni (500 bp a 2Kb). Il gel funziona da «setaccio molecolare»: le molecole piccole, infatti migrano attraverso l agarosio più velocemente di quelle grandi. A ph neutro, il DNA è carico negativamente grazie alla presenza dei gruppi fosfato; poiché le cariche opposte si attraggono, i frammenti di DNA migrano verso il polo positivo del campo elettrico, quindi il DNA, carico ( ) a ph neutro, migra verso il polo positivo (anodo). Dopo un certo intervallo di tempo si interrompe la corrente elettrica e si esamina la distanza percorsa dai frammenti; per visualizzare il DNA si usa un colorante che diventa fluorescente quando viene esposto alla luce ultravioletta L agarosio è un polisaccaride estratto da un alga marina, costituito da residui alternati di D-galattosio e 3,6- anidro L-galattosio. L' agarosio è sciolto in opportuno tampone (TBE o TAE) e fatto gelificare su un supporto di vetro o di plastica. Si crea così una matrice gelatinosa, che dipende dalla concentrazione dell agarosio, le cui maglie permettono la separazione di macromolecole a diverso PM. In linea di massima il range di concentrazione varia tra lo 0,3% - 2%.. Le quantità di agarosio e di tampone determinano la porosità del gel, quindi si stabiliscono in base al peso molecolare della banda di DNA attesa. Parametri che influenzano la velocità di migrazione DIMENSIONE DEL DNA Le molecole lineari di dsdna migrano a velocità inversamente proporzionali al logaritmo in base 10 del numero di paia di basi. Le molecole più grandi sono sottoposte a maggiori forze di attrito, e inoltre incontrano maggiore ostacolo nel trovare una via attraverso i pori del gel rispetto alle molecole più piccole. CONCENTRAZIONE DI AGAROSIO NEL GEL la sua concentrazione determina il potere risolutivo del gel. CONFORMAZIONE DEL DNA: un DNA superavvolto, lineare e circolare ha velocità di migrazione diversa anche se di dimensione uguale:
4 - La forma SUPERAVVOLTA corre più veloce perché è più compatta; - La forma CIRCOLARE corre più lenta perché è la più ingombrante e fa più fatica a muoversi all interno dei pori del gel; - La forma LINEARE si colloca a metà (la forma lineare è quella che si ritrova come prodotto nella PCR). VOLTAGGIO APPLICATO_ PRESENZA DI BROMURO DI ETIDIO Colorante fluorescente (intercalante) che si intercala tra le basi planari della doppia elica di DNA. Se esposto a luce ultravioletta diventa fluorescente e permette di visualizzare le bande di DNA che hanno migrato sul gel. La fluorescenza emessa è proporzionale alla quantità del campione. Assorbimento a 254 nm (U.V.) ed emissione nel visibile (590 nm) COMPOSIZIONE (FORZA IONICA) DEL BUFFER Il tampone è una soluzione salina che serve: a condurre la corrente elettrica e a controllare il ph durante l elettroforesi (Tris, che consente il mantenimento di un valore costante di ph della soluzione. A o B sta per acido Borico o Acetico che forniscono l appropriata forza ionica al tampone. EDTA, che chela i cationi divalenti, come il magnesio) Come preparare un gel d agarosio Scioglere l agarosio in polvere in una soluzione tampone a 100 C Aggiungere l etidio di bromuro alla soluzione di agarosio sciolto Versare la soluzione di agarosio, evitando di formare bolle, nella vaschetta per elettroforesi, inserire il pettine per i pozzetti. I pozzetti si formano quando, una volta solidificato il gel, viene tolto il pettine. Lasciare solidificare il gel a temperatura ambiente per circa 15min. Quando è solidificato i gel diventa opaco. Una volta solidificato il gel inserire il supporto con il gel nella vaschetta per elettroforesi, versare il Tampone TBE 1x nella camera di corsa Togliere lentamente il pettine, tenendosi perpendicolare rispetto al gel, senza rompere i pozzetti Caricare i campioni, in un pozzetto laterale caricare il marker di DNA a peso molecolare noto. Chiudere la cameretta Avviare la corsa, applicando il voltaggio.
5 Reverse trascriptase-pcr RT-PCR La reazione di retro-trascrizione (trascrittasi inversa) consente la produzione di cdna ( DNA complementare) a partire da una molecola di mrna (RNA messaggero). L enzima che catalizza questa reazione è la trascrittasi inversa, una DNA polimerasi RNA-dipendente con attività 5-3, identificato inizialmente nei retrovirus eucariotico, AMV avian myeloblastosis virus, M-MuLV Moloney leukemia virus murino ed anche in altri. Il prodotto di retro trascrizione, cdna, può essere in seguito amplificato mediante PCR classica oppure utilizzato per valutare l espressione genica di un gene specifico mediante qrt-pcr (quantitative real time PCR). Prima di retro-trascrivere il cdna, si tratta mrna con DNAse per eliminare ogni tracce di DNA genomico che potrebbe dare falsi positivi. I principali componenti per effettuare una RT-PCR sono: - mrna precedentemente estratto - enzima DNA polimerasi RNA-dipendente, detto comunemente trascrittasi inversa o retrotrascrittasi. - Deossiribonucleotidi trifosfato (dntps). - Soluzione tampone - ioni bivalenti Mg 2+, che fungono da cofattori per la polimerasi. - Oligo dt, sequenze oligonucleotidiche di Timina in grado di legarsi alla coda di polya dell RNA e fungere da innesco per l inizio della retro trascrizione. Real Time PCR La real time-pcr, definita anche quantitative real time-pcr (qrt-pcr), è una tecnica che consente l amplificazione e la quantizzazione in tempo reale del cdna target man mano che esso si forma, e non alla fine della reazione come nella PCR classica. Questa tecnica associa amplificazione e quantizzazione in un unica reazione e si basa sugli stessi steps di una classica PCR. Utilizza marcatori fluorescenti, i quali emettendo un segnale fluorescente permettono Il calcolo della quantità di DNA dei campioni determinando il ciclo della PCR, ciclo soglia (threshold cycle Ct) in cui si osserva un significativo incremento di fluorescenza al di sopra del basale. Il Ct è strettamente correlato al numero di copie iniziali del target quindi un numero iniziale di copie pari al doppio anticipa il Ct di un ciclo; un numero iniziale di copie pari alla metà posticipa il Ct di un ciclo.
6 In una reazione di real-time PCR, la fluorescenza aumenta in proporzione all accumulo dei prodotti di PCR. Il numero dei cicli necessari affinché un campione raggiunga il suo Ct è inversamente proporzionale al numero di copie target presente inizialmente. Per valutare la quantità fluorescenza emessa ad ogni ciclo, è possibile monitorare la reazione di PCR durante la fase esponenziale, dove il primo aumento significativo dei prodotti di PCR correlano con l aumento iniziale del cdna target. Il segnale fluorescente può essere dato da diversi fluorocromi, come il SYBR Green, oppure da sonde, quali le TaqMan. Il SYBR Green è un colorante fluorescente che lega il DNA a doppio filamento. L'eccitazione massima è a 494 nm, mentre l'emissione massima è a 521 nm. L'intensità del segnale emesso aumenta durante il procedere della PCR a causa dell'accumularsi dei prodotti di amplificazione. L'utilizzo delle sonde costituisce un metodo di rivelazione altamente specifico e sensibile. Le sonde ad ibridazione, specifiche per il frammento di interesse, sono marcate con molecole fluorescenti e devono essere disegnate in una regione del DNA compresa tra i due primers, pertanto i prodotti aspecifici della PCR non verranno rivelati. La sonda TaqMan è un oligonucleotide che, come i primers della PCR, viene disegnato per essere complementare alla sequenza bersaglio da amplificare. La sonda è disegnata in modo da ibridarsi all interno del frammento amplificato nella reazione di PCR e presenta all estremità 5 un fluoroforo Reporter ed all estremità 3 una molecola Quencher. 5 REPORTER (R): fluorocromo ad alta energia che emette fluorescenza 3 QUENCHER (Q): fluorocromo a bassa energia che spegne la fluorescenza del reporter Se R e Q si trovano vicini, Q spegne l'effetto di R perchè i fotoni di R vengono assorbiti da Q. L aumento di fluorescenza del Reporter è direttamente proporzionale al numero di ampliconi generati. Nel corso dell amplificazione la sonda viene distrutta mediante idrolisi dall attività esonucleasica 5-3 specifica per il doppio filamento associata alla Taq polimerasi. Questo risulta in un aumento del segnale fluorescente dovuto all abolizione dell effetto di "quenching" che dipende dalla vicinanza. In altre parole una volta legata alla sequenza, la sonda resta lì immobile senza emettere nessun segnale. Quando la polimerasi comincia la fase di estensione del primer, l'attività 5' nucleasica dell'enzima degrada la sonda, questo porta al rilascio del fluorocromo, il quale non essendo più vicino alla molecola "Quencer" emette una fluorescenza, per cui i prodotti di amplificazione sono direttamente proporzionali al segnale di amplificazione. I campioni possono essere quantizzati in modo:
7 - Assoluto: necessita di standard di cui si conosce la concentrazione nota (virus, DNA plasmidico, RNA) Amplificando in parallelo ai campioni a concentrazione non nota gli standard a concentrazione nota a diverse diluizioni, è possibile ottenere una retta di regressione (curva di taratura o curva standard). Conoscendo il valore del Ct, la concentrazione dei campioni non noti si può ricavare interpolazione ponendo il Ct in ordinata e la concentrazione iniziale in ascissa. Il sistema consente di valutare l efficienza di amplificazione mediante tre punti: Slope indica il numero dei cicli che intercorrono tra due diluizioni dello standard che differiscono per 1 log. Coeff. Correlazione (R): esprime le correlazione esistenti tra i diversi segmenti che compongono la retta di regressione. Intercetta nell asse X: definisce il numero di cicli necessari per rilevare una copia di DNA target. - Relativa: la quantificazione viene effettuata paragonando i Ct per determinare il cambiamento di espressione di un gene target in un campione rispetto ad un altro e necessita di controlli endogeni (gene housekeeping) non di una curva standard. Per ogni esperimento di quantificazione relativa è necessario un : target: sequenza di DNA da analizzare calibratore: il campione da usare come riferimento per l analisi comparativa controllo endogeno: un gene espresso costitutivamente in tutti i campioni analizzati, necessario per normalizzare i dati rispetto alla quantità di DNA caricato e a variazioni di efficienza della reazione. COMPONENTI DELLA REAZIONE: 1) DNA target 2) DNA polimerasi 3) Due oligonucleotidi 4) dntps 5) Probe fluorescente Real-Time PCR: applicazioni: - Quantificazione virale - Quantificazione dell espressione genica - Efficacia della terapia farmacologica - Misura dei danni al DNA - Controllo di qualità e validazione dei saggi - Genotyping
8 - Malattia minima residua (MRD) IMMUNOFLUORESCENZA L immunofluorescenza, come l immunoistochimica, è un metodo altamente specifico per la rilevazione di determinati antigeni presenti nel tessuto o nelle cellule da esaminare, mediante l uso di anticorpi specifici. Gli anticorpi monoclonali sono in grado di legarsi solo alla proteina di interesse con elevata specificità. L'interazione anticorpo-antigene è detectabile utilizzando un fluorocromo coniugato all'anticorpo e può essere visualizzata mediante microscopia a fluorescenza. Il fluorocromo consente di visualizzare la distribuzione dell antigene nel campione tramite un microscopio a fluorescenza (per es. microscopi a fluorescenza e confocali). Si distinguono due metodi di IF a seconda che il fluoroforo sia coniugato all'anticorpo primario o secondario: IF diretta: utilizza un singolo anticorpo diretto contro l antigene selezionato (noto come anticorpo primario). L'anticorpo è direttamente coniugato a un fluoroforo. (fluorocromi, ad es. FITC, l'isotiocianato di fluoresceina, TRITC l isotiocianato di tetrametilrodamina) IF indiretta: utilizza due anticorpi. L'anticorpo primario non è coniugato e per la rilevazione si utilizza un anticorpo secondario coniugato al fluoroforo diretto contro l'anticorpo primario. Vantaggi: Rapidità Semplicità Economicità Versatilità Svantaggi : Operatore-dipendenza Lettura interpretativa Tessuti di difficile reperimento Microscopio a fluorescenza
9 Dosaggio immunoenzimatico (ELISA) Enzyme-linked immunosorbent assay (ELISA), conosciuto anche come dosaggio immunoenzimatico, è una tecnica biochimica utilizzata principalmente in immunologia per rilevare la presenza di un anticorpo o un antigene in un campione. E una tecnica utile anche per quanto riguarda la produzione di anticorpi monoconali nello screening di ibridomi. Sfrutta la capacità delle superfici di plastica di adsorbire quantità piccole ma rivelabili di proteine. Si tratta di un saggio in fase solida effettuato su piastre da micro dosaggio, ogni piastra contiene 96 pozzetti e permette di saggiare contemporaneamente un elevato numero di campioni. I tests ELISA rappresentano un mezzo fondamentale per l industria farmaceutica, con applicazioni nella scoperta di farmaci, negli studi con modelli animali, nei trials clinici. Drug discovery: nelle industrie farmaceutiche più di molecole sono testate di routine con kit ELISA nelle fasi iniziali dello screen per identificare molecole promettenti. Food science industry: i kit ELISA vengono utilizzati per analizzare le tossine nel cibo. Negli ultimi anni l utilizzo della tecnica ELISA si è sempre più diffuso anche nei laboratori di ricerca. In particolare è utilizzata per: - Determinare presenza e concentrazione di un antigene nel campione da testare; - Determinare presenza e concentrazione di un anticorpo nel campione da testare; - Determinare presenza e concentrazione di citochine secrete, sia in vivo nel plasma che in vitro in medium La sensibilità di questa tecnica dipende da 4 fattori: 1. numero di molecole di anticorpo legato alla fase solida; 2. avidità dell anticorpo per l antigene; 3. avidità dell anticorpo detection per l antigene; 4. attività specifica dell anticorpo detection. Limite: informazioni solo sulla presenza/concentrazione di una molecola, ma non sulle sue proprietà biochimiche (es peso molecolare, localizzazione cellulare, ecc). Si possono utilizzare diversi metodi: Metodo Diretto: consente di dosare un antigene. L anticorpo primario che deve essere marcato con un enzima indicatore. Si fa reagire una soluzione di un anticorpo specifico marcato con un antigene ancorato ad una fase solida. Dopo aver lavato, si aggiunge il substrato dell'enzima. In questa tecnica, l'attività enzimatica misurata sarà direttamente proporzionale alla quantità di antigene presente. Metodo Indiretto: Serve per dosare un anticorpo, anziché un antigene. E l anticorpo secondario ad essere coniugato all enzima. Il materiale da dosare (ad es. un siero umano contenente IgG) viene fatto reagire con l apposito antigene legato ad una fase solida. Dopo reazione del siero con l antigene immobilizzato, il materiale che non si è legato viene rimosso mediante lavaggio. Si aggiunge poi un anticorpo con un enzima. Si lava di nuovo e si aggiunge substrato dell enzima: l attività misurata sarà direttamente proporzionale alla quantità di anticorpo specifico presente nel campione analizzato.
10 Caratteristiche: - è veloce ed accurato; - permette di determinare: - la presenza di anticorpi contro l antigene immobilizzato; - la quantità di immunoglobuline presenti, grazie all utilizzo di controlli positivi presenti in concentrazioni note. Metodo competitivo : si fa reagire una quantità nota di antigene marcato con un enzima e una quantità ignota dello stesso antigene libero, con uno specifico anticorpo legato ad una fase solida. Sul sito di legame dell'anticorpo si crea competizione tra l'antigene marcato e quello "freddo", cioè non marcato. Dopo aver lavato il complesso con tampone, si aggiunge il substrato dell'enzima e si misura l'attività enzimatica. In condizioni standard, l'attività enzimatica misurata è proporzionale alla frazione di antigene marcato presente nella miscela. La differenza tra questo valore e quello di un campione in cui si è omesso l'antigene libero rappresenta la misura della concentrazione di antigene nel campione ignoto. Metodo del doppio anticorpo (sandwich): si fa reagire una soluzione ignota di antigene con un anticorpo specifico legato ad una fase solida, si lava e si aggiunge un secondo anticorpo; questo anticorpo dovrà essere policlonale opp monoclonale in grado di legarsi ad un diverso epitopo dell'antigene. Dopo un secondo lavaggio si aggiunge il substrato dell'enzima. L'attività enzimatica dosata in condizioni standard, sarà direttamente proporzionale alla quantità di antigene presente. Caratteristiche: - E una tecnica veloce ed accurata; - utilizzando l antigene purificato (se disponibile) come standard, permette di determinare la quantità assoluta dell antigene nel campione analizzato. - richiede due anticorpi che riconoscono epitopi diversi sull antigene - Questo risultato può essere raggiunto sia utilizzando due anticorpi monoclonali con diversa specificità, o anticorpi policlonali Vantaggi: Elevato grado di specificità Svantaggi: non tutti gli anticorpi possono essere utilizzati Il metodo più utilizzato per la rivelazione dell avvenuto legame antigene anticorpo consiste nella coniugazione dell anticorpo secondario con una molecola che può essere direttamente rivelata. Anticorpi coniugati ad enzimi: vengono utilizzati enzimi in grado di convertire un substrato incolore in un prodotto colorato. - Alkaline phosphatase: per anticorpi coniugati alla fosfatasi alcalina si usa in genere il substrato p- nitrophenylphosphate (pnpp), che sviluppa un intenso colore giallo misurabile a nm. - Peroxidase: per gli anticorpi coniugati alla perossidasi si possono scegliere diversi substrati, tra i più utilizzati: 2,2-azinodiethyl-benzthiazoline sulfonate (ABTS), che sviluppa un colore blu-verde misurabile a nm. o-phenylene diamine (OPD), sviluppa un colore arancio scuro misurabile a 492 nm.
11 Caratteristiche dei substrati: - stabile - sicuro, non tossico - poco costoso Un comune obiettivo che si vuole raggiungere con il test ELISA è quello di stimare la concentrazione di una molecola in una preparazione test. Questo è possibile confrontando la risposta con quella ottenuta con una preparazione standard che contiene concentrazioni note della molecola. Il test viene eseguito diluendo progressivamente sia la preparazione standard (concentrazione nota) sia la preparazione test (concentrazione sconosciuta). Si ottiene una curva standard utilizzata per determinare la concentrazione della molecola nella preparazione test. La quantità di segnale generato dal legame del secondo anticorpo è proporzionale alla quantità di antigene presente nel campione testato. La quantificazione può essere ottenuta generando una curva standard e comparando l attività ottenuta con il campione a quella ottenuta sulla curva standard. Dosaggio immunometrico (CLIA) Chemiluminescent immuno Assay (CLIA) è una tecnica immunoenzimatica di ultima generazione comparsa nei primi anni novanta e permette di determinare la concentrazione di un campione in base alla quantità di luce emessa inseguito ad una reazione chimica, in cui l'antigene o l'anticorpo sono etichettati con una molecola in grado di emettere luce durante la reazione chimica. Questa luce viene utilizzato per misurare la formazione del complesso antigene anticorpo e quindi la quantità all interno del campione in analisi. A differenza dell ELISA, la tecnologia CLIA consente una riduzione dei tempi di analisi (30-45 minuti). CLIA è stato applicato in diversi campi, tra cui il monitoraggio ambientale, diagnosi clinica, sicurezza alimentare e analisi farmaceutica. E una tecnica: - che permette un'analisi selettiva, sensibile, rapida e semplice. - analizzare un numero di campioni maggiore rispetto ad un ELISA - completamente automatizzata, riducendo gli errori operatori dipendente L utilizzo di enzimi che marcano specifici substrati, con elevata attività è una delle caratteristiche di questa tecnologia. I enzimi più utilizzati sono HRP e ALP. Viene utilizzata nella routine di laboratorio per dosaggio di ormoni, marcatori tumorali, diagnosi di malattie autoimmuni etc.
12
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
5- Estrazione ed analisi di DNA
 5- Estrazione ed analisi di DNA Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU) 16 ore) Marco Scocchi ( BIO/10-11) La struttura del DNA La traduzione dell informazione
5- Estrazione ed analisi di DNA Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU) 16 ore) Marco Scocchi ( BIO/10-11) La struttura del DNA La traduzione dell informazione
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
TECNICHE IMMUNOCHIMICHE
 TECNICHE IMMUNOCHIMICHE Tutte le tecniche immunochimiche si basano sull impiego di anticorpi Struttura tipica di un immunoglobulina (Ig) Le immunoglobuline si dividono in diverse classi, ciascuna dotata
TECNICHE IMMUNOCHIMICHE Tutte le tecniche immunochimiche si basano sull impiego di anticorpi Struttura tipica di un immunoglobulina (Ig) Le immunoglobuline si dividono in diverse classi, ciascuna dotata
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Analisi degli alimenti tramite PCR Real-Time. Introduzione ad una tecnologia all'avanguardia
 Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
METODICHE DI IMMUNOCHIMICA
 METODICHE DI IMMUNOCHIMICA IMMUNOCHIMICA permette la determinazione quantitativa di proteine in tessuti e omogenati INTERAZIONI ANTIGENE-ANTICORPO L immunochimica si basa sulle interazioni antigene-anticorpo
METODICHE DI IMMUNOCHIMICA IMMUNOCHIMICA permette la determinazione quantitativa di proteine in tessuti e omogenati INTERAZIONI ANTIGENE-ANTICORPO L immunochimica si basa sulle interazioni antigene-anticorpo
Immunologia e Immunologia Diagnostica TECNICHE IMMUNOLOGICHE
 Immunologia e Immunologia Diagnostica TECNICHE IMMUNOLOGICHE Tecniche di laboratorio La maggior parte delle tecniche di laboratorio biomedico si basano sull utilizzo degli anticorpi. Gli anticorpi sono
Immunologia e Immunologia Diagnostica TECNICHE IMMUNOLOGICHE Tecniche di laboratorio La maggior parte delle tecniche di laboratorio biomedico si basano sull utilizzo degli anticorpi. Gli anticorpi sono
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli. I nostri inquilini invisibili
 Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
Elettroforesi di Tamara Benincasa
 Elettroforesi di Tamara Benincasa L elettroforesi è una metodologia utilizzata in laboratorio attraverso la quale molecole biologiche (ammino acidi, peptidi, proteine, nucleotidi ed acidi nucleici), dotate
Elettroforesi di Tamara Benincasa L elettroforesi è una metodologia utilizzata in laboratorio attraverso la quale molecole biologiche (ammino acidi, peptidi, proteine, nucleotidi ed acidi nucleici), dotate
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola)
 Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
Reazione a catena della polimerasi (PCR) ed elettroforesi
 Gioele Casagranda, Camilla Larentis, Emanuele Pallaoro, Filippo Quattrocchi Povo, 1 marzo 2016 P r o g e t t o C L I L - E s p e r i m e n t o f i n a l e Reazione a catena della polimerasi (PCR) ed elettroforesi
Gioele Casagranda, Camilla Larentis, Emanuele Pallaoro, Filippo Quattrocchi Povo, 1 marzo 2016 P r o g e t t o C L I L - E s p e r i m e n t o f i n a l e Reazione a catena della polimerasi (PCR) ed elettroforesi
Polymerase Chain Reaction
 Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Quantizzazione del DNA con spettrofotometro
 Quantizzazione del DNA ed elettroforesi 1 Quantizzazione del DNA con spettrofotometro Al termine della metodica di estrazione, occorre quantificare il DNA isolato. Un metodo per quantificare in modo preciso
Quantizzazione del DNA ed elettroforesi 1 Quantizzazione del DNA con spettrofotometro Al termine della metodica di estrazione, occorre quantificare il DNA isolato. Un metodo per quantificare in modo preciso
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI
 DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3. Criteri di identificazione
 Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Perché sintetizzare per via chimica gli oligonucleotidi?
 Perché sintetizzare per via chimica gli oligonucleotidi? Molte sono le utilizzazioni di oligonucleotidi sintetizzati chimicamente.. Oligonucleotidi sintetici servono: in molti esperimenti in biologia molecolare
Perché sintetizzare per via chimica gli oligonucleotidi? Molte sono le utilizzazioni di oligonucleotidi sintetizzati chimicamente.. Oligonucleotidi sintetici servono: in molti esperimenti in biologia molecolare
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
ELETTROFORESI. Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico.
 ELETTROFORESI ELETTROFORESI Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico. Anioni ANODO (+) Cationi CATODO (-) v = velocità di migrazione μ =
ELETTROFORESI ELETTROFORESI Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico. Anioni ANODO (+) Cationi CATODO (-) v = velocità di migrazione μ =
Metodi di Quantificazione dell Espressione Genica
 Metodi di Quantificazione dell Espressione Genica Cosa significa quantificare l espressione genica? Alcune tecniche per quantificare l espressione genica Parte I: RT PCR qrt PCR (Real Time) Parte II: Northern
Metodi di Quantificazione dell Espressione Genica Cosa significa quantificare l espressione genica? Alcune tecniche per quantificare l espressione genica Parte I: RT PCR qrt PCR (Real Time) Parte II: Northern
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
REAL-TIME PCR QUANTIFICAZIONE DELL ESPRESSIONE GENICA
 REAL-TIME PCR QUANTIFICAZIONE DELL ESPRESSIONE GENICA Un metodo per misurare l mrna nei tessuti biologici e in varie condizioni sperimentali Identificazione del meccanismo di azione dei farmaci Farmaco
REAL-TIME PCR QUANTIFICAZIONE DELL ESPRESSIONE GENICA Un metodo per misurare l mrna nei tessuti biologici e in varie condizioni sperimentali Identificazione del meccanismo di azione dei farmaci Farmaco
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
PRINCIPALI APPLICAZIONI DELLA PCR. PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b) PRINCIPALI TIPI DI PCR a)
 EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
PROGETTO DNA chiavi in mano. Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi
 PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
Scientists with lab coats: Corso di laboratorio Chimico-Biologico. Dott.ssa Valeria Berton Università di Verona
 Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
ELETTROFORESI. Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico.
 ELETTROFORESI ELETTROFORESI Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico. Anioni ANODO (+) Cationi CATODO (-) v = velocità di migrazione μ =
ELETTROFORESI ELETTROFORESI Tecnica analitica che consente di separare molecole cariche grazie all applicazione di un campo elettrico. Anioni ANODO (+) Cationi CATODO (-) v = velocità di migrazione μ =
Real Time PCR. La PCR Real Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico.
 eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI
 VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
Ag multivalenti o polivalenti
 ANTIGENE (Ag)= qualsiasi sostanza capace di legarsi in modo specifico ad un anticorpo (Ab) o al TCR Tutti gli antigeni sono riconosciuti dai linfociti o da anticorpi specifici. Peptidi Zuccheri Lipidi
ANTIGENE (Ag)= qualsiasi sostanza capace di legarsi in modo specifico ad un anticorpo (Ab) o al TCR Tutti gli antigeni sono riconosciuti dai linfociti o da anticorpi specifici. Peptidi Zuccheri Lipidi
Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa
 WESTERN BLOTTING Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa Tecnica di trasferimento delle proteine da un gel ad una membrana associata ad utilizzo di anticorpi
WESTERN BLOTTING Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa Tecnica di trasferimento delle proteine da un gel ad una membrana associata ad utilizzo di anticorpi
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Laboratorio di Genetica Molecolare: Prof. Davide Bizzaro, Prof. Anna La Teana
 Laboratorio di Genetica Molecolare: Prof. Davide Bizzaro, Prof. Anna La Teana Determinazione del polimorfismo del gene TAS2R38 coinvolto nella percezione dell amaro. I mammiferi riconoscono soltanto 5
Laboratorio di Genetica Molecolare: Prof. Davide Bizzaro, Prof. Anna La Teana Determinazione del polimorfismo del gene TAS2R38 coinvolto nella percezione dell amaro. I mammiferi riconoscono soltanto 5
PCR. (Reazione a catena della polimerasi) ESTRAZIONE del DNA da gel di AGAROSIO. Corso di INGEGNERIA GENETICA, Prof. Renato Fani,
 PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Retrovirus e DNA ricombinante. Lezioni d'autore di Paola Vinesi
 Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Southern blot. !Per. determinare la presenza o assenza di specifici frammenti genici. !Studio. della struttura e dell organizzazione di un gene
 Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
lezione martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
Lo studio delle comunità microbiche. È stato reso possibile dalle Tecniche molecolari
 Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
Metodiche analitiche. Anna Maria Eleuteri
 Metodiche analitiche per la rilevazione i di mutazioni i Anna Maria Eleuteri Analisi del DNA dopo PCR Analisi dei prodotti di amplificazione - Elettroforesi su gel di agarosio Metodi per lo studio di mutazioni
Metodiche analitiche per la rilevazione i di mutazioni i Anna Maria Eleuteri Analisi del DNA dopo PCR Analisi dei prodotti di amplificazione - Elettroforesi su gel di agarosio Metodi per lo studio di mutazioni
ELETTROFORESI DI PROTEINE
 ELETTROFORESI DI PROTEINE ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni
ELETTROFORESI DI PROTEINE ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni
Analisi quantitativa dell interazione proteina-proteina
 Analisi quantitativa dell interazione proteina-proteina Legame recettore-ligando (proteina-proteina) Il legame per il ligando deve essere: reversibile ad alta affinità saturabile deve portare ad una risposta
Analisi quantitativa dell interazione proteina-proteina Legame recettore-ligando (proteina-proteina) Il legame per il ligando deve essere: reversibile ad alta affinità saturabile deve portare ad una risposta
Protocollo Crime Scene Investigation
 Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Elettroforesi proteine ed acidi nucleici
 Elettroforesi proteine ed acidi nucleici Flavia Frabetti Tecnici di laboratorio 2010/2011 ELETTORFORESI metodo fisico di separazione che sfrutta la mobilità elettroforetica di molecole cariche, sottoposte
Elettroforesi proteine ed acidi nucleici Flavia Frabetti Tecnici di laboratorio 2010/2011 ELETTORFORESI metodo fisico di separazione che sfrutta la mobilità elettroforetica di molecole cariche, sottoposte
Analisi Immunoenzimatiche
 Analisi Immunoenzimatiche Mara Stefanelli INAIL Dipartimento Innovazioni Tecnologiche e Sicurezza degli Impianti, Prodotti e Insediamenti antropici Istituto Superiore di Sanità Dipartimento Ambiente e
Analisi Immunoenzimatiche Mara Stefanelli INAIL Dipartimento Innovazioni Tecnologiche e Sicurezza degli Impianti, Prodotti e Insediamenti antropici Istituto Superiore di Sanità Dipartimento Ambiente e
PROCEDURA NEGOZIATA PER LA FORNITURA DI PRODOTTI PER BIOLOGIA MOLECOLARE OCCORRENTI ALLA U.O. DI EMATOLOGIA
 AZIENDA OSPEDALIERA REGIONALE SAN CARLO DI POTENZA Ospedale S. Carlo di Potenza Ospedale S. Francesco di Paola di Pescopagano Via Potito Petrone 85100 Potenza Tel. 0971-61 11 11 Codice Fiscale e Partita
AZIENDA OSPEDALIERA REGIONALE SAN CARLO DI POTENZA Ospedale S. Carlo di Potenza Ospedale S. Francesco di Paola di Pescopagano Via Potito Petrone 85100 Potenza Tel. 0971-61 11 11 Codice Fiscale e Partita
Buone regole. -nel dubbio, chiedere! LEGGERE BENE IL PROTOCOLLO PRIMA DI INIZIARE
 Precauzioni da adottare in laboratorio: -non mangiare nè bere -indossare il camice -indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette -nel dubbio, chiedere! Buone regole LEGGERE
Precauzioni da adottare in laboratorio: -non mangiare nè bere -indossare il camice -indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette -nel dubbio, chiedere! Buone regole LEGGERE
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
TECNICHE ELETTROFORETICHE
 TECNICHE ELETTROFORETICHE L elettroforesi e un metodo di separazione che si basa sulla diversa mobilita degli ioni (o di sostanze elettricamente cariche), quando posti all interno di un campo elettrico
TECNICHE ELETTROFORETICHE L elettroforesi e un metodo di separazione che si basa sulla diversa mobilita degli ioni (o di sostanze elettricamente cariche), quando posti all interno di un campo elettrico
Southern Blot: Per l analisi del DNA. (sonda = DNA o RNA). Northern Blot: Per l analisi dell RNA. (sonda = DNA o RNA).
 Southern Blot: Per l analisi del DNA. (sonda = DNA o RNA). Northern Blot: Per l analisi dell RNA. (sonda = DNA o RNA). Western Blot: Per l analisi delle proteine. (sonda = anticorpo). Una tecnica in cui
Southern Blot: Per l analisi del DNA. (sonda = DNA o RNA). Northern Blot: Per l analisi dell RNA. (sonda = DNA o RNA). Western Blot: Per l analisi delle proteine. (sonda = anticorpo). Una tecnica in cui
WESTERN BLOT o IMMUNOFISSAZIONE
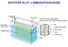 WESTERN BLOT o IMMUNOFISSAZIONE BLOTTING Trasferimento di macromolecole su una membrana immobilizzante. Southern DNA - da Edward Southern 1970 Northern Western RNA Proteine SCOPO DEL WESTERN BLOTTING Consente
WESTERN BLOT o IMMUNOFISSAZIONE BLOTTING Trasferimento di macromolecole su una membrana immobilizzante. Southern DNA - da Edward Southern 1970 Northern Western RNA Proteine SCOPO DEL WESTERN BLOTTING Consente
U.O.C. di Epatologia Clinica e Biomolecolare. Unità di misura. Repertorio. 200 ml 10.000 U. 500 test
 U.O.C. di Epatologia Clinica e Biomolecolare Lotto N. DESCRIZIONE PRODOTTO Quantità annua richiesta Unità di misura Repertorio CND Codice Prodotto, Confezione Offerta e nome commerciale Prezzo unitario
U.O.C. di Epatologia Clinica e Biomolecolare Lotto N. DESCRIZIONE PRODOTTO Quantità annua richiesta Unità di misura Repertorio CND Codice Prodotto, Confezione Offerta e nome commerciale Prezzo unitario
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR. Dott. Paolo Cascio
 LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Biologia Molecolare. CDLM in CTF La trascrizione negli eucarioti
 Biologia Molecolare CDLM in CTF 2010-2011 La trascrizione negli eucarioti I meccanismi della trascrizione Organizzazione generale delle sequenze regolative Il macchinario generale della trascrizione Si
Biologia Molecolare CDLM in CTF 2010-2011 La trascrizione negli eucarioti I meccanismi della trascrizione Organizzazione generale delle sequenze regolative Il macchinario generale della trascrizione Si
Lezioni di biotecnologie
 Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
04_biochimica. MFN0366-A1 (I. Perroteau) - Tecniche biochimiche-molecolari: Western blot
 1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI
 CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
Metodologie di microbiologia e genetica
 Metodologie di microbiologia e genetica Modulo Metodologie di Genetica Prof.ssa Antonella Furini ESERCITAZIONE N 3 PREPARAZIONE DEL FRAMMENTO E DEL taglio con enzimi di restrizione 1. Taglio del plasmide
Metodologie di microbiologia e genetica Modulo Metodologie di Genetica Prof.ssa Antonella Furini ESERCITAZIONE N 3 PREPARAZIONE DEL FRAMMENTO E DEL taglio con enzimi di restrizione 1. Taglio del plasmide
USO DEI MARCATORI MOLECOLARI A DNA PER LA TRACCIABILITÀ DEI VITIGNI NEI VINI
 USO DEI MARCATORI MOLECOLARI A DNA PER LA TRACCIABILITÀ DEI VITIGNI NEI VINI CLAUDIO D ONOFRIO*, FABIOLA MATESE*, ELEONORA DUCCI E GIUSEPPE CELANO *UNIVERSITÀ DI PISA, DIPARTIMENTO DI SCIENZE AGRARIE,
USO DEI MARCATORI MOLECOLARI A DNA PER LA TRACCIABILITÀ DEI VITIGNI NEI VINI CLAUDIO D ONOFRIO*, FABIOLA MATESE*, ELEONORA DUCCI E GIUSEPPE CELANO *UNIVERSITÀ DI PISA, DIPARTIMENTO DI SCIENZE AGRARIE,
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Microscopia confocale
 MFN0366-A1 (I. Perroteau) - microscopia confocale Microscopia confocale 1 MFN0366-A1 (I. Perroteau) - microscopia confocale Esempio di analisi con focale: vedi il filmato corrispondente Vedi filmato 2
MFN0366-A1 (I. Perroteau) - microscopia confocale Microscopia confocale 1 MFN0366-A1 (I. Perroteau) - microscopia confocale Esempio di analisi con focale: vedi il filmato corrispondente Vedi filmato 2
LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara
 LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara Molti sono i virus che possono causare epatiti acute e
LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara Molti sono i virus che possono causare epatiti acute e
Metodi di studio delle proteine :
 Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D determinazione del peso molecolare Spettrofotometro
Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D determinazione del peso molecolare Spettrofotometro
Il DNA come molecola in grado di veicolare informazione ereditabile (genetica)
 Il DNA come molecola in grado di veicolare informazione ereditabile (genetica) Essenz. Alberts: cap 6 La trasmissione dell informazione replicazione trascrizione traduzione DNA RNA Proteina da, dg, dc,
Il DNA come molecola in grado di veicolare informazione ereditabile (genetica) Essenz. Alberts: cap 6 La trasmissione dell informazione replicazione trascrizione traduzione DNA RNA Proteina da, dg, dc,
Schema a blocchi di uno spettrofluorimetro
 MONOCROMATORE EMISSIONE EM Schema a blocchi di uno spettrofluorimetro MONOCROMATORE ECCITAZIONE SORGENTE EXC RIVELATORE (TUBO FOTOMOLTIPLICATORE) Anche il DNA assorbe nell UV Cosa determina l assorbanza
MONOCROMATORE EMISSIONE EM Schema a blocchi di uno spettrofluorimetro MONOCROMATORE ECCITAZIONE SORGENTE EXC RIVELATORE (TUBO FOTOMOLTIPLICATORE) Anche il DNA assorbe nell UV Cosa determina l assorbanza
Reg. (CE) 2073, punto 24 delle considerazioni preliminari
 I risultati delle analisi dipendono dal metodo analitico utilizzato; pertanto occorre associare ad ogni criterio microbiologico un metodo di riferimento specifico. Tuttavia, gli operatori del settore alimentare
I risultati delle analisi dipendono dal metodo analitico utilizzato; pertanto occorre associare ad ogni criterio microbiologico un metodo di riferimento specifico. Tuttavia, gli operatori del settore alimentare
ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe
 ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe Z-2111-200 20 (0.2 ml) Per la rilevazione della translocazione t(9;22)(q34;q11)mediante ibridazione in situ fluorescente (FISH).... Dispositivo per uso
ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe Z-2111-200 20 (0.2 ml) Per la rilevazione della translocazione t(9;22)(q34;q11)mediante ibridazione in situ fluorescente (FISH).... Dispositivo per uso
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Applicazione della biologia molecolare nella valutazione del benessere del cavallo
 UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
lati esterni altamente Idrofilici
 I due filamenti complementari del DNA sono antiparalleli: uno è in direzione 5-3 e l altro in direzione 3-5. parte interna idrofobica lati esterni altamente Idrofilici APPAIAMENTO DELLE BASI AZOTATE: 2
I due filamenti complementari del DNA sono antiparalleli: uno è in direzione 5-3 e l altro in direzione 3-5. parte interna idrofobica lati esterni altamente Idrofilici APPAIAMENTO DELLE BASI AZOTATE: 2
Determinazioni quantitative in farmacologia
 Determinazioni quantitative in farmacologia 1 Dosaggio biologico determinazione quantitativa della concentrazione o della potenza di una sostanza per mezzo della valutazione della risposta biologica che
Determinazioni quantitative in farmacologia 1 Dosaggio biologico determinazione quantitativa della concentrazione o della potenza di una sostanza per mezzo della valutazione della risposta biologica che
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
14/02/2013 VALORIZZAZIONE E CARATTERIZZAZIONE DEL POLLINE SECONDO L APPROCCIO MOLECOLARE. Caratterizzazione molecolare dei pollini (1)
 VALORIZZAZIONE E CARATTERIZZAZIONE DEL POLLINE SECONDO L APPROCCIO MOLECOLARE Antonella Cersini*, Marcella Milito**, Ugo Marchesi*, Silvia Puccica*, Valeria Antognetti* *U.S. Biotecnologie e **Apicoltura.
VALORIZZAZIONE E CARATTERIZZAZIONE DEL POLLINE SECONDO L APPROCCIO MOLECOLARE Antonella Cersini*, Marcella Milito**, Ugo Marchesi*, Silvia Puccica*, Valeria Antognetti* *U.S. Biotecnologie e **Apicoltura.
ENZIMI. Un enzima è un catalizzatore (acceleratore) di reazioni biologiche.
 ENZIMI ENZIMI Un enzima è un catalizzatore (acceleratore) di reazioni biologiche. Catalizzatore = sostanza in grado di accelerare lo svolgimento di una reazione chimica e quindi di aumentarne la sua velocità,
ENZIMI ENZIMI Un enzima è un catalizzatore (acceleratore) di reazioni biologiche. Catalizzatore = sostanza in grado di accelerare lo svolgimento di una reazione chimica e quindi di aumentarne la sua velocità,
Esempi di tecniche analitiche che sono dirette applicazioni di elettromagnetismo
 Esempi di tecniche analitiche che sono dirette applicazioni di elettromagnetismo spettrometria di massa elettroforesi Spettrometria di massa Tecnica per indagine che si basa sulla ionizzazione di una molecola
Esempi di tecniche analitiche che sono dirette applicazioni di elettromagnetismo spettrometria di massa elettroforesi Spettrometria di massa Tecnica per indagine che si basa sulla ionizzazione di una molecola
Un anticorpo è una proteina prodotta dai linfociti B in risposta all ingresso nell organismo di un antigene.
 Un antigene è una molecola (proteica o polisaccaridica), o una sua parte, che è in grado di stimolare una risposta specifica del sistema immunitario. Affinché una molecola funga da antigene è necessario
Un antigene è una molecola (proteica o polisaccaridica), o una sua parte, che è in grado di stimolare una risposta specifica del sistema immunitario. Affinché una molecola funga da antigene è necessario
