Sintesi chimica del DNA
|
|
|
- Gaspare Venturi
- 5 anni fa
- Visualizzazioni
Transcript
1 Sintesi chimica del DNA
2 Sintesi chimica di oligonucleotidi Per la sintesi vengono utilizzati fosforamidi dei vari nucleotidi. La fosforamide contiene un buon gruppo uscente che promuove la reazione di formazione di un legame fosfoestere (fosfito). Il monomero attivato aggiunto contiene dimetossitritile (DMT) che protegge il 5 OH e un gruppo β-cianoetilico di protezione del gruppo ossidrilico 3.
3 Sintesi in fase solida di oligonucleotidi Nella sintesi su fase solida l addizione di nucleotidi avviene in direzione 3-5, contraria alla direzione di sintesi enzimatica. Il nucleotide 3 terminale è legato covalentemente con il gruppo 3 alla fase solida e poi il suo 5 viene deprotetto dal gruppo DMT. Ad ogni ciclo un nucleotide viene aggiunto, reagisce con il suo 3 attivato con la fosforamidite e si lega al 5 del nucleotide associato alla fase solida. Successivamente il 5 del nucleotide appena aggiunto viene deprotetto e la reazione può essere di nuovo effettuate con un altro nucleotide. Dopo la sintesi i legami fosfoestere vengono ossidati da fosfiti a fosfati
4 Sintesi in fase solida di oligonucleotidi La fase di capping serve ad inattivare le estremità 5 OH che non hanno reagito ed impedire la sintesi di oligonucleotidi con sequenze diverse da quella selezionata (resteranno di lunghezza inferiore)
5 Efficienza nella sintesi su fase solida
6 Sequenziamento
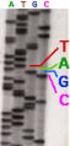
7 Sequenziamento con idrolisi chimica (Maxam and Gilbert 1977) (formic acid) Uso di reagenti chimici che alterano una o al massimo due basi. La base alterata viene essere rimossa (attraverso riscaldamento) e una seconda reazione con piperidina porta alla rottura della catena polinucleotidica nel sito abasico I vari frammenti ottenuti dopo l idrolisi nelle diverse condizioni sono separati su gel Le molecole di DNA, sottoposte alie differenti reazioni chimiche e specifiche per le varie basi, sono marcate ad una estremità. Questo permette di visualizzare solo i frammenti che iniziano con l estremità marcata e terminano con il sito di taglio e la misura loro lunghezza identifica la posizione della base che è stata alterata nella specifica reazione
8 Lettura della sequenza di DNA Esempio dei frammenti, contenenti l estremità marcata, ottenibili con l idrolisi chimica specifica per la guanina di una molecola di DNA
9
10 Sequenziamento chain-terminator o metodo con dideoxinucleotidi (Sanger, 1977) Metodo enzimatico che richiede l uso di una DNA-polimerasi e specifici primer La reazione è condotta in presenza di paricolari nucleotidi,ididesossinucleotidi, che se incorporati portano alla prematura terminazione della sintesi del DNA perchè privi del 3 OH Principio della tecnica 3 T T T T 5 5 primer 3 dda dda dda dda Nella reazione oltre al datp è presente anche il ddatp (in genere in rapporto 1:10). Quando occasionalmente viene incorporato il ddatp la reazione di polimerizzazione si interrompe e la lunghezza del frammento mi fornisce la posizione della base usata come ddntp
11 Le molecole di DNA da sequenziare vengono sottoposte a quattro differenti reazioni, in ognuna delle quali è presente un ddntp. Le reazioni di sintesi del DNA vengono innescate da uno stesso prime opportunamente marcato per permettere la migliore visualizzazione dei frammenti prodotti. I frammenti prodotti durante la sintesi vengono successivamente separati su gel e, anche in questo caso, la misura loro lunghezza identifica la posizione della base che è stata utilizzata come ddntp nella reazione di polimerizzazione.
12 Lettura della sequenza di DNA
13 Sequenziamento automatico Nelle quattro differenti reazioni di polimerizzazione, oltre che essere utilizzati differenti ddntp, la sintesi del DNA viene innescata dallo stesso primer ma marcato con quattro fluorofori differenti. Questo permette di distingure i frammenti che sono terminati dal ddatp o da un altro ddntp perché hanno una fluorescenza differente. I prodotti delle quattro reazioni vengono riuniti e analizzati sullo stesso gel. La separazione elettroforetica permetterà di distinguere i vari frammenti in base alle dimensioni e lo specifico colore di fluorescenza indicherà la base che si trovava in quella posizione.
14 Sequenziamento automatico I prodotti delle quattro reazioni vengono riuniti e separati in continua su gel o elettroforesi capillare. Un rivelatore è posto alla fine del ge,l o del capillare, e analizza l intensità ed il colore del frammento che viaggia nel gel, permettendo cosi di avere informazioni sul tipo di base presente al termine di quel frammento. Il risultato è una serie di picchi consecutivi, che rappresentano i vari frammenti eluiti a dimensioni crescenti (ognuno è più lungo di una base) ed il colore identifica la base terminale (permettendo di leggere direttamente la sequenza del frammento).
15 NGS Next Generation Sequencing
16
17 Illumina
18 Ion torrent e Pyrosequencing
19 La PCR
20 3 5 La Polymerase Chain Reaction o PCR Come funziona? Miscela di reazione Replicazione in vitro di determinati tratti di DNA, selezionati tramite specifici primers 5 3 Primers d. NTPs Thermal Stable DNA Polymerase Denaturazione 95 C 3 Taq Ripetizione del ciclo Taq 5 3 Estensione 3 Taq 5 3 Annealing C Taq 3 Estensione 72 C
21 La PCR (il primo ciclo)
22 La PCR (secondo ciclo)
23
24 CICLO QUANTITA' DI DNA
25 Applicazioni della PCR Amplificazione selettiva di specifiche regioni di DNA (la sequenza di DNA amplificato è delimitata dai primers e la loro specificità determina la qualità dell amplificazione Indicazioni sulla presenza di specifiche sequenze di DNA in un campione. Se presenti verranno amplificate e quindi rivelabili tramite elettroforesi altrimenti possono essere considerate assenti. In campo diagnostico permette di determinare infezioni virali (esempio retrovirus o virus difficili da diagnosticare), altri patogeni, presenza di DNA estraneo, ecc. Clonaggio di sequenze informazionali (geni o altre regioni di DNA), a patto di conoscere le sequenze delle regioni limitrofe (inizio e fine) per costruire specifici primers. Genotipizzazione (DNA fingerprinting)
26 Genotipizzazione tramite PCR
27 ANALISI DELLE STR Le STR (Short Tandem Repeats) sono sequenze di 2-6 bp ripetute da 3 a 50 volte che si trovano all interno di regioni introniche Sono note anche come DNA microsatellite o simple sequence repeats (SSRs) Le STR possono essere amplificate mediante PCR La regione ripetuta è di lunghezza variabile mentre le regioni adiacenti, dove si legano i primers, sono costanti Gli individui possono essere omozigoti (i due alleli hanno la stessa lunghezza) o eterozigoti (i due alleli hanno lunghezze diverse)
28 Amplificazione di una STR 195 bp 170 bp TCAT repeat unit
29 Frequency Frequenza allelica nelle STR Number of repeats TH01 Marker Caucasians (N=427) Blacks (N=414) Hispanics (N=414) *Proc. Int. Sym. Hum. ID (Promega) 1997, p. 34
30 FBI s CODIS DNA Database
31 Loci STR del CODIS TPOX D3S1358 D5S818 D8S1179 TH01 VWA FGA CSF1PO D7S820 AMEL D13S317 D16S539 D18S51 D21S11 AMEL 13 CODIS Core STR Loci with Chromosomal Positions
32 Analisi comparativa delle STR Amplificazione contemporanea di più STR mediante Multiplex PCR Ridotta probabilità di errori introdotti con la PCR (Informazione nella lunghezza e non nella sequenza) Risultati uniformi Possibilità di analizzare campioni in tracce Analisi mediante DNA-PAGE o elettroforesi capillare Rapidità nella separazione Comparazione e/o identificazione L analisi comparativa di 13 STR permette di ridurre la probabilità di un random match fino a 1 su Disponibilità di kit commerciali e apparecchiature dedicate
33 Human identity testing Casi forensici analisi comparative tra sospettati e prove Test di paternità/maternità identificazione padre/madre Studi storici Studi su persone scomparse Disastri di massa identificazione Military DNA dog tag (alternativa alla piastrina) Creazione di DNA databases relativi a criminali condannati
34 Sangue Liquido seminale Saliva Urine Capelli Denti Ossa Tessuti Campioni analizzabili
35 Procedura di analisi
36 Color Separation Esempio di un Forensic STR Multiplex Kit AmpFlSTR Profiler Plus Kit available from PE Biosystems (Foster City, CA) 100 bp 200 bp 300 bp 400 bp Size Separation D3 vwa FGA 5-FAM (blue) A D8 D21 D18 JOE (green) D5 D13 D7 NED (yellow) GS500-internal lane standard ROX (red) 9 STRs amplified along with sex-typing marker amelogenin in a single PCR reaction
37 Two different individuals Human Identity Testing con Multiplex STRs AmpFlSTR SGM Plus kit D3 amelogenin D19 D8 TH01 VWA D21 FGA DNA Size (base pairs) D16 D18 D2 probability of a random match: ~1 in 3 trillion amelogenin D3 Results obtained in less than 5 hours with a spot of blood the size of a pinhead D19 D8 TH01 VWA D21 FGA D16 D18 D2 Simultaneous Analysis of 10 STRs and Gender ID
38 PCR quantitativa
39 Amplificazione e quantificazione di specifici frammenti di DNA Amplificazione attraverso l uso della Polymerase Chain Reaction o PCR (reazione a catena della polimerasi) Quantificazione in tempo reale del DNA amplificato mediante l uso di sonde fluorescenti (molecole in grado di assorbire la luce e riemetterla ad un determinato colore) L insieme di queste due tecniche prende il nome di Real-time PCR
40 DNA amplificato (scala log) Limite della PCR In teoria la quantità di DNA amplificato raddoppia ad ogni ciclo e il suo quantitativo può essere espresso dalla formula Teorico Prodotto (T) = (DNA iniziale) x 2 n (n = numero di cicli) In realtà dopo un certo numero di cicli la quantità di DNA non aumenta più esponenzialmente (consumo dei substrati, ovvero I precursori per costruire le catene di DNA, e inattivazione dell enzima) Reale Numero di cicli
41 Campioni con diverso contenuto iniziale dello stesso DNA e sottoposti ad amplificazione mediante PCR seguiranno delle cinetiche di amplificazione differenti 4 unità 1 unità 1/8 di unità
42 Determinazione del quantitativo di DNA amplificato SYBR Green I
43 Il SYBR Green I agente fluorescente intercalante Estensione 3 ID ID Taq 5 5 ID ID ID Taq 3 Lettura della fluorescenza 3 l l l ID ID ID ID ID Taq 5 Taq ID ID ID ID 3 l l
44 DNA amplificato (scala log) Come sono quantificati i dati raccolti dallo strumento? Teorico Reale Detector threshold Numero di ciclo Una volta determinata la linea di base (fluorescenza nei primi cicli di amplificazione), la linea di threshold viene scelta come il valore di fluorescenza che statisticamente supera la linea di base (10 volte la deviazione standard della linea di base).
45 Ct : threshold cycle E il ciclo al quale la fluorescenza relativa al DNA amplificato supera il valore della linea di threshold. Il Ct è proporzionale al numero di sequenze di DNA iniziali amplificate con la PCR.
46 Il Ct è proporzionale al numero di sequenze di DNA iniziali amplificate con la PCR. 4 unità Ct =23 1 unità Ct = 25 1/8 di unità Ct = 28 Valori di Ct 4 units Ct=
47 Ct : threshold cycle L uso di curve di taratura permette anche di determinare il valore assoluto di specifiche sequenze di DNA nel campione analizzato.
48 Che cosa è la Real-time PCR? Si basa sulla determinazione quantitativa e in tempo reale dei prodotti di amplificazione della PCR mediante l uso di molecole fluorescenti che si legano al DNA La Real-time PCR permette di quantificare i livelli di una specifica molecola di DNA misurando il numero di cicli (C t ) necessari per ottenere un quantitativo di prodotto amplificato superiore ad un determinato livello (threshold)
49 Applicazioni della Real-time PCR Quantificazione precisa di DNA o RNA in campioni Stima del numero di copie di un gene (OGM) Studi di espressione genica RNA (quantificazione del mrna)
50 Quantificazione dell espressione genica Tutte le cellule di un organismo contengono lo stesso DNA (e quindi le stesse informazioni o geni). Tuttavia non tutti i geni vengono egualmente espressi (diversità tra cellule) Ogni gene espresso viene convertito in mrna per poi essere tradotto in proteina Le differenti proteine prodotte in una cellula ne determinano le specifiche funzioni
51 E possibile quantificare i livelli di mrna di specifici geni mediante la Real-time PCR L mrna viene prima convertito in DNA (cdna) e poi sequenze specifiche (corrispondenti a determinati geni) vengono quantificate mediante la tecnica E così possibile identificare e quantificare geni che vengono diversamente espressi in determinate cellule o in seguito a modifiche (trasformazione tumorale)
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
BIOCHIMICA MOLECOLARE CLINICA
 Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato
 Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato Lo strumento è composto da:! Un sistema ottico per eccitare il marcatore fluorescente
Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato Lo strumento è composto da:! Un sistema ottico per eccitare il marcatore fluorescente
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
PCR. (Reazione a Catena della Polimerasi) Kary Mullis
 POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL ELETTROFORESI SU GEL Metodo per separare le molecole (DNA, RNA, proteine, etc.) sulla base di proprieta fisiche o chimiche quali: (1)dimensioni (2)forma (3)carica elettrica L'AGAROSIO
ELETTROFORESI SU GEL ELETTROFORESI SU GEL Metodo per separare le molecole (DNA, RNA, proteine, etc.) sulla base di proprieta fisiche o chimiche quali: (1)dimensioni (2)forma (3)carica elettrica L'AGAROSIO
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay)
 Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Analisi degli alimenti tramite PCR Real-Time. Introduzione ad una tecnologia all'avanguardia
 Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti
 Seminario per il corso misure con biomarcatori A.A. 2005-2006 Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti Perché utilizzare
Seminario per il corso misure con biomarcatori A.A. 2005-2006 Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti Perché utilizzare
Studio pilota per la creazione e l utilizzo di un database contenente i profili genetici di una popolazione controllata di cani
 MSRCTE 01/11 Studio pilota per la creazione e l utilizzo di un database contenente i profili genetici di una popolazione controllata di cani C. Cammà Reparto Ricerca e Sviluppo Biotecnologie Obiettivo
MSRCTE 01/11 Studio pilota per la creazione e l utilizzo di un database contenente i profili genetici di una popolazione controllata di cani C. Cammà Reparto Ricerca e Sviluppo Biotecnologie Obiettivo
DNA Profiling e Genetica Forense. Silvia Fuselli Lezione 1
 DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Patologie da analizzare
 Fasi cruciali Scelta della patologia da analizzare Scelta del campione da analizzare Scelta dell approccio da utilizzare Scelta della tecnica da utilizzare Analisi statistica del dati Conferme con approcci
Fasi cruciali Scelta della patologia da analizzare Scelta del campione da analizzare Scelta dell approccio da utilizzare Scelta della tecnica da utilizzare Analisi statistica del dati Conferme con approcci
Utilizzo di marcatori molecolari in evoluzione e conservazione
 Utilizzo di marcatori molecolari in evoluzione e conservazione Un marcatore genetico è qualsiasi elemento con una base genetica, in genere identificabile con facilità, che permette di caratterizzare un
Utilizzo di marcatori molecolari in evoluzione e conservazione Un marcatore genetico è qualsiasi elemento con una base genetica, in genere identificabile con facilità, che permette di caratterizzare un
Genome variability part II
 Genome variability part II I MARCATORI POLIMORFICI 1. RFLPs Restriction Fragment Lenght Polymorphism (bi-allelic).tipizzabili mediante PCR or Southern based screening. Studi di associazione. 2. SNPs Single
Genome variability part II I MARCATORI POLIMORFICI 1. RFLPs Restriction Fragment Lenght Polymorphism (bi-allelic).tipizzabili mediante PCR or Southern based screening. Studi di associazione. 2. SNPs Single
Polymerase Chain Reaction
 Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Regione del Veneto - POR FESR BANDO PER IL SOSTEGNO A PROGETTI DI RICERCA CHE PREVEDONO L IMPIEGO DI RICERCATORI
 MESSA A PUNTO DI UNA PROCEDURA DIAGNOSTICO MOLECOLARE PER L INDIVIDUAZIONE DELLA PREDISPOSIZIONE GENETICA ALL ALLERGIA ALLE ARACHIDI ED ALLA FRUTTA SECCA IN GENERALE Regione del Veneto - POR FESR 2014-2020
MESSA A PUNTO DI UNA PROCEDURA DIAGNOSTICO MOLECOLARE PER L INDIVIDUAZIONE DELLA PREDISPOSIZIONE GENETICA ALL ALLERGIA ALLE ARACHIDI ED ALLA FRUTTA SECCA IN GENERALE Regione del Veneto - POR FESR 2014-2020
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
Tecnologia del DNA e la genomica. Dr ssa Elisa Mazzoni, Ph.D Biologia Applicata a.a Università di Ferrara
 Tecnologia del DNA e la genomica Dr ssa Elisa Mazzoni, Ph.D Biologia Applicata a.a 2018-2019 Università di Ferrara Alcune Tecnologie del DNA Estrazione del DNA Taglio del DNA con enzimi di restrizione
Tecnologia del DNA e la genomica Dr ssa Elisa Mazzoni, Ph.D Biologia Applicata a.a 2018-2019 Università di Ferrara Alcune Tecnologie del DNA Estrazione del DNA Taglio del DNA con enzimi di restrizione
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia. Numero di gara
 Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
NEXT- GENERATION DNA SEQUENCING
 NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin»
 Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin» E-mail: beniamino.trombetta@uniroma1.it Individuare in modo UNIVOCO un individuo (parentela, reati, ecc) da una data traccia
Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin» E-mail: beniamino.trombetta@uniroma1.it Individuare in modo UNIVOCO un individuo (parentela, reati, ecc) da una data traccia
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
TRASCRIZIONE DEL DNA. Formazione mrna
 TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
Scientists with lab coats: Corso di laboratorio Chimico-Biologico. Dott.ssa Valeria Berton Università di Verona
 Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Polymerase chain reaction (PCR) qualche ora
 Lezione 3 Metodologie di sequenziamemento di DNA ed RNA. Da Sanger sequencing ad High Throughput (HTS) o di Next Generation (NGS) o di seconda generation Polymerase chain reaction (PCR) qualche ora 25
Lezione 3 Metodologie di sequenziamemento di DNA ed RNA. Da Sanger sequencing ad High Throughput (HTS) o di Next Generation (NGS) o di seconda generation Polymerase chain reaction (PCR) qualche ora 25
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
La GENETICA DELLE POPOLAZIONI. studia con modelli matematici, a livello di gruppi di individui, variabilità genetica
 La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna Istituto Superiore di Sanità
 Piani per la Sicurezza delle Acque Nuove metodologie di analisi ed inquinanti emergenti Bologna, 12 ottobre 2017 Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna
Piani per la Sicurezza delle Acque Nuove metodologie di analisi ed inquinanti emergenti Bologna, 12 ottobre 2017 Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna
E POSSIBILE FAR EVOLVERE LE PROTEINE IN LABORATORIO
 E POSSIBILE FAR EVOLVERE LE PROTEINE IN LABORATORIO EVOLUZIONE GUIDATA PROTEINE CON FUNZIONI OTTIMIZZATE O NUOVE L EVOLUZIONE NATURALE RICHIEDE TEMPO VARIABILITA GENETICA Ricombinazione Mutazione Selezione
E POSSIBILE FAR EVOLVERE LE PROTEINE IN LABORATORIO EVOLUZIONE GUIDATA PROTEINE CON FUNZIONI OTTIMIZZATE O NUOVE L EVOLUZIONE NATURALE RICHIEDE TEMPO VARIABILITA GENETICA Ricombinazione Mutazione Selezione
Progetto Tandem Biologia saperi minimi Anno accademico Marzo 2012 COGNOME...
 Progetto Tandem Biologia saperi minimi Anno accademico 2011-2012 2 Marzo 2012 COGNOME... NOME 1) Quali delle seguenti affermazioni sulla struttura primaria delle proteine è falsa? a) può essere ramificata
Progetto Tandem Biologia saperi minimi Anno accademico 2011-2012 2 Marzo 2012 COGNOME... NOME 1) Quali delle seguenti affermazioni sulla struttura primaria delle proteine è falsa? a) può essere ramificata
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
a) DENATURAZIONE b) APPAIAMENTO c) SINTESI
 La PCR è una tecnica biomolecolare messa a punto da Mullis et al. (1986) che perme6e l amplificazione esponenziale in vitro di sequenze di acidi nucleici. La reazione di PCR illustrata in figura prevede
La PCR è una tecnica biomolecolare messa a punto da Mullis et al. (1986) che perme6e l amplificazione esponenziale in vitro di sequenze di acidi nucleici. La reazione di PCR illustrata in figura prevede
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PRINCIPALI APPLICAZIONI DELLA PCR. PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b) PRINCIPALI TIPI DI PCR a)
 EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
1. Quantificazione del genomico e determinazione della purezza
 ESERCITAZIONE 2 1. Quantificazione del genomico e determinazione della purezza Una volta estratto il DNA si procede con la quantificazione. Tale quantificazione viene fatta normalmente con l utilizzo di
ESERCITAZIONE 2 1. Quantificazione del genomico e determinazione della purezza Una volta estratto il DNA si procede con la quantificazione. Tale quantificazione viene fatta normalmente con l utilizzo di
0,8% riesce a separare un range di dimensioni da circa 500 bp a circa bp.
 L elettroforesi è una tecnica che utilizza un campo elettrico per separare e visualizzare frammenti di DNA in base al loro peso molecolare ed alla loro carica. Gli acidi nucleici, migrano sempre verso
L elettroforesi è una tecnica che utilizza un campo elettrico per separare e visualizzare frammenti di DNA in base al loro peso molecolare ed alla loro carica. Gli acidi nucleici, migrano sempre verso
Metodi di Quantificazione dell Espressione Genica
 Metodi di Quantificazione dell Espressione Genica Cosa significa quantificare l espressione genica? Alcune tecniche per quantificare l espressione genica Parte I: RT PCR qrt PCR (Real Time) Parte II: Northern
Metodi di Quantificazione dell Espressione Genica Cosa significa quantificare l espressione genica? Alcune tecniche per quantificare l espressione genica Parte I: RT PCR qrt PCR (Real Time) Parte II: Northern
LA REAZIONE A CATENA DELLA POLIMERASI
 Polymerase Chain Reaction LA REAZIONE A CATENA DELLA POLIMERASI e LE SUE APPLICAZIONI Prima diagnosi molecolare sul DNA 1978 *Primo ormone umano ricombinante (insulina) *Premio Nobel agli scopritori degli
Polymerase Chain Reaction LA REAZIONE A CATENA DELLA POLIMERASI e LE SUE APPLICAZIONI Prima diagnosi molecolare sul DNA 1978 *Primo ormone umano ricombinante (insulina) *Premio Nobel agli scopritori degli
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Elettroforesi = migrazione in campo elettrico
 Elettroforesi = migrazione in campo elettrico Molte molecole di interesse biologico possiedono gruppi ionizzabili. In opportune condizioni di ph presentano una carica elettrica + o e quindi sono in grado
Elettroforesi = migrazione in campo elettrico Molte molecole di interesse biologico possiedono gruppi ionizzabili. In opportune condizioni di ph presentano una carica elettrica + o e quindi sono in grado
ANALISI MOLECOLARE DEL GENOMA
 ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
Lezione 3. Genoma umano come esempio di genoma eucariote
 Lezione 3 Genoma umano come esempio di genoma eucariote Schema della lezione Sommario degli elementi contenuti in un genoma eucariote Variabilità: dove si trova e come si definisce I grandi progetti internazionali
Lezione 3 Genoma umano come esempio di genoma eucariote Schema della lezione Sommario degli elementi contenuti in un genoma eucariote Variabilità: dove si trova e come si definisce I grandi progetti internazionali
DNA: Struttura e caratteristiche
 DNA: Struttura e caratteristiche Il DNA è un acido nucleico formato da monomeri detti nucleotidi. Ogni nucleotide è formato da: Zucchero pentoso (desossiribosio) Gruppo fosfato Base azotata Basi azotate:
DNA: Struttura e caratteristiche Il DNA è un acido nucleico formato da monomeri detti nucleotidi. Ogni nucleotide è formato da: Zucchero pentoso (desossiribosio) Gruppo fosfato Base azotata Basi azotate:
BIOLOGIA MOLECOLARE Ha fondamentalmente due scopi
 BIOLOGIA MOLECOLARE Ha fondamentalmente due scopi DIMOSTRAZIONE DELLA CLONALITA : LINFOMI DIMOSTRAZIONE DI ALTERAZIONI GENETICHE SCOPO DIAGNOSTICO (e di ricerca) soprattutto SCOPO TERAPEUTICO CONCETTO
BIOLOGIA MOLECOLARE Ha fondamentalmente due scopi DIMOSTRAZIONE DELLA CLONALITA : LINFOMI DIMOSTRAZIONE DI ALTERAZIONI GENETICHE SCOPO DIAGNOSTICO (e di ricerca) soprattutto SCOPO TERAPEUTICO CONCETTO
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
RELAZIONE TECNICA. Indagini Biologico-Molecolari per Accertamento di Paternità
 RELAZIONE TECNICA Indagini Biologico-Molecolari per Accertamento di Paternità PROSPETTO RIASSUNTIVO DELL'ANALISI ANALISI INDAGINE DI PATERNITA' PERSONE SOTTOPOSTE AL TEST QUESITO Campione Biologico Tampone
RELAZIONE TECNICA Indagini Biologico-Molecolari per Accertamento di Paternità PROSPETTO RIASSUNTIVO DELL'ANALISI ANALISI INDAGINE DI PATERNITA' PERSONE SOTTOPOSTE AL TEST QUESITO Campione Biologico Tampone
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Tecniche di sequenziamento del DNA
 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
COME È FATTO? Ogni filamento corrisponde ad una catena di nucleotidi
 Il DNA Il DNA è una sostanza che si trova in ogni cellula e contiene tutte le informazioni sulla forma e sulle funzioni di ogni essere vivente: eppure è una molecola incredibilmente semplice. COME È FATTO?
Il DNA Il DNA è una sostanza che si trova in ogni cellula e contiene tutte le informazioni sulla forma e sulle funzioni di ogni essere vivente: eppure è una molecola incredibilmente semplice. COME È FATTO?
Diagnosi Molecolare delle
 Diagnosi Molecolare delle Malattie infettive Diagnosimolecolare Biologiamolecolare Cosa è la Biologia molecolare? E una branca della biologia che studia gli esseri viventi a livello dei meccanismi molecolari
Diagnosi Molecolare delle Malattie infettive Diagnosimolecolare Biologiamolecolare Cosa è la Biologia molecolare? E una branca della biologia che studia gli esseri viventi a livello dei meccanismi molecolari
Capitolo B5 Manipolare il genoma: le biotecnologie
 Capitolo B5 Manipolare il genoma: le biotecnologie Quesiti e problemi 1 Le biotecnologie sono applicazioni tecnologiche che utilizzano organismi viventi o loro derivati per realizzare prodotti e processi
Capitolo B5 Manipolare il genoma: le biotecnologie Quesiti e problemi 1 Le biotecnologie sono applicazioni tecnologiche che utilizzano organismi viventi o loro derivati per realizzare prodotti e processi
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
CORSO INTEGRATO DI GENETICA a.a. 2011-2012. Genetica molecolare in medicina: Analisi di Mutazioni. Cristina Bombieri 7 dicembre 2011
 CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
Progetto Lars-Biotec
 Unità didattiche: prima fase: Progetto Lars-Biotec Laboratorio di Ricerca sperimentale nel settore delle Biotecnologie Bioinformatica: vengono scelti e analizzati geni appartenente al genoma umano conosciuti
Unità didattiche: prima fase: Progetto Lars-Biotec Laboratorio di Ricerca sperimentale nel settore delle Biotecnologie Bioinformatica: vengono scelti e analizzati geni appartenente al genoma umano conosciuti
Ibridazione in situ per mrna: tecnica e interpretazione
 Patologia nodulare della tiroide: focus sugli approcci diagnostici Ibridazione in situ per mrna: tecnica e interpretazione Isabella Giovannoni U.O.C. Anatomia Patologica Ibridazione -> appaiamento di sequenze
Patologia nodulare della tiroide: focus sugli approcci diagnostici Ibridazione in situ per mrna: tecnica e interpretazione Isabella Giovannoni U.O.C. Anatomia Patologica Ibridazione -> appaiamento di sequenze
Marcatori molecolari per l analisi genica, genetica e genomica
 Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
MARCATORI MOLECOLARI
 MARCATORI MOLECOLARI MARCATORI ENZIMATICI A B D E C Marcatori biochimici: (A) fosfo-gluco-mutasi (PGM); (B) lipoossigenasi (LOX) in soia; (C) esterasi (EXT) in poa pratense; (D,E) rappresentazioni schematiche
MARCATORI MOLECOLARI MARCATORI ENZIMATICI A B D E C Marcatori biochimici: (A) fosfo-gluco-mutasi (PGM); (B) lipoossigenasi (LOX) in soia; (C) esterasi (EXT) in poa pratense; (D,E) rappresentazioni schematiche
FONDO DI PEREQUAZIONE 2002
 - 1 - FONDO DI PEREQUAZIONE 2002 Camera di commercio di Torino In collaborazione con CO.AL.VI.(Consorzio di Tutela della Razza Bovina Piemontese) Laboratorio della Camera di Commercio CARATTERIZZAZIONE
- 1 - FONDO DI PEREQUAZIONE 2002 Camera di commercio di Torino In collaborazione con CO.AL.VI.(Consorzio di Tutela della Razza Bovina Piemontese) Laboratorio della Camera di Commercio CARATTERIZZAZIONE
Scientists with lab coats: Corso di laboratorio Chimico-Biologico. Dott.ssa Valeria Berton Università di Verona
 Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Come «vedere» il DNA: la PCR PCR = Polymerase Chain Reaction (reazione a catena della polimerasi)
Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Come «vedere» il DNA: la PCR PCR = Polymerase Chain Reaction (reazione a catena della polimerasi)
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
AZIENDA OSPEDALIERA UNIVERSITARIA INTEGRATA - VERONA (D.Lgs. n. 517/ Art. 3 L.R.Veneto n. 18/2009) D I M T
 1 Allegato 1 D I M T PROCEDURA APERTA PER LA FORNITURA QUINQUENNALE PER IL DIPARTIMENTO INTERAZIENDALE DI MEDICINA TRASFUSIONALE DI PRODOTTI PER LA TIPIZZAZIONE GENOMICA A BASSA E ALTA RISOLUZIONE SUDDIVISA
1 Allegato 1 D I M T PROCEDURA APERTA PER LA FORNITURA QUINQUENNALE PER IL DIPARTIMENTO INTERAZIENDALE DI MEDICINA TRASFUSIONALE DI PRODOTTI PER LA TIPIZZAZIONE GENOMICA A BASSA E ALTA RISOLUZIONE SUDDIVISA
LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE
 LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE Metodi e tecniche per la rilevazione e quantificazione della resistenza Insetticidi ed acaricidi Manicardi
LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE Metodi e tecniche per la rilevazione e quantificazione della resistenza Insetticidi ed acaricidi Manicardi
Applicazione della biologia molecolare nella valutazione del benessere del cavallo
 UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
