Allineamento multiplo
|
|
|
- Norberto Gastone Bernasconi
- 6 anni fa
- Visualizzazioni
Transcript
1 Allineamento multiplo
2 Allineamenti multipli Vs. allineamenti a coppie A B 1: 2: 3: 4: 5: 6: 1: 2: 3: 4: 5: 6:
3 Applicazioni dell allineamento multiplo Assemblaggio dei genomi Primers per PCR Consensi, motivi Profili, modelli markoviani Definizione di famiglie Alberi filgenetici Inferenze strutturali Inferenze funzionali
4 Significato biologico dell allineamento multiplo L allineamento multiplo riassume La storia evolutiva di una famiglia di proteine Quindi, si possono ricavare informazioni su: La conservazione dei residui dipendente dalla funzione La conservazione dei residui dipendente dalla struttura
5 Significato funzionale della conservazione TRYI_DROME : ENTK_PIG/8 : THRB_BOVIN : KLK1_MOUSE : CTRA_BOVIN : CTR1_ANOGA : CTRL_HALRU : IIGGSDQLIRNAPWQVSIQISAR----HECGGVIYSKEIIITAGHCLHER-SVTLMKV-----RVGA---QNHNYGG-TLVPVAAY--KVHEQFDSRFLH--- IVGGNDSREGAWPWVVALYYNG----QLLCGASLVSRDWLVSAAHCVYG----RNLEPSKWKAILG--LHMTSNLTSPQIVTRLIDEIVINPHYNRRRKD--- IVEGQDAEVGLSPWQVMLFRKSPQE--LLCGASLISDRWVLTAAHCLLYPPWDKNFTVDDLLVRIGK-HSRTRYERKVEKISMLDK-IYIHPRYNWKEN---- IVGGFNCEKNSQPWQVAVYRFT----KYQCGGILLNVNWVLTAAHCHND-----KYQV-----WLGK-NNFLEDEPSAQHRLVSK--AIPHPDFNMSLLNEHT IVNGEEAVPGSWPWQVSLQDKTG---FHFCGGSLINENWVVTAAHCGVT----TSDVV-----VAGEFDQGSSSEK-IQKLKIAK--VFKNSKYNSLTIN--- VVGGEVAKNGSAPYQVSLQVPGWG---HNCGGSLLNDRWVLTAAHCLVG-HAPGDLMV-----LVGT---NSLKEGG-ELLKVDK--LLYHSRYNLPRFH--- IVGGSNAAAGEFPWQGSLQVRSGTSWFHICGCVLYTTSKALTAAHCLSN--SASSYRL--G---FGMLR-MNNVDGTEQYSSVTS--YTNHPNYNGNAAG--- : 84 : 90 : 95 : 86 : 85 : 85 : 90 TRYI_DROME : ENTK_PIG/8 : THRB_BOVIN : KLK1_MOUSE : CTRA_BOVIN : CTR1_ANOGA : CTRL_HALRU : YDIAVLRLSTP-LTFGLSTRAINLAS---TSP--SGGTTVTVTGWGH----TDNG---ALSDSLQKAQLQIIDRGECASQKFGYGAD-FVGEETI SDIAMMHLEFK-VNYTDYIQPICLPE---ENQVFPPGRICSIAGWGK---VIYQG---SPADILQEADVPLLSNEKCQQQMP-EYN---ITENMM LDRDIALLKLKRP-IELSDYIHPVCLPDKQTAAKLLHAGFKGRVTGWGNRRETWTTSVAEVQPSVLQVVNLPLVERPVCKAS---TRIR--ITDNMF PQPEDDYSNDLMLLRLKKP-ADITDVVKPIDLPT---EEP--KLGSTCLASGWGS---ITPVKY--EYPDELQCVNLKLLPNEDCAKA---HIEK--VTDDML NDITLLKLSTA-ASFSQTVSAVCLPS---ASDDFAAGTTCVTTGWGL---TRYTNA--NTPDRLQQASLPLLSNTNCKKY---WGTK--IKDAMI NDIGLVRLEQP-VQFSELVQSVEYSE-----KAVPANATVRLTGWGR---TSANG---PSPTLLQSLNVVTLSNEDCNKK---GGDPGYTDVGHL YPNDIAVLRLTSSMDTSSSAVGPSVWLL VERLCRTNMYDQR--MGKTQWRWQHPNNLQKVDMTVLTNSDCSSRWSGISGAT-VNSGHI : 165 : 171 : 186 : 173 : 166 : 165 : 175 TRYI_DROME : ENTK_PIG/8 : THRB_BOVIN : KLK1_MOUSE : CTRA_BOVIN : CTR1_ANOGA : CTRL_HALRU : CAAS----TD-ADACTGDSGGPLVASSQ------LVGIVSWG-YRCADDNYPGVYADVAILRPWI CAGYE--EGG-IDSCQGDSGGPLMCLEN--NRWLLAGVTSFG-YQCALPNRPGVYARVPKFTEWI CAGYKPGEGKRGDACEGDSGGPFVMKSPYNNRWYQMGIVSWG-EGCDRDGKYGFYTHVFRLKKWI CAGDM--DGG-KDTCAGDSGGPLICDGV------LQGITSWGPSPCGKPNVPGIYTRVLNFNTWI CAGA----SG-VSSCMGDSGGPLVCKKN--GAWTLVGIVSWG-SSTCSTSTPGVYARVTALVNWV CTLTK---TG-EGACNGDSGGPLVYEGK------LVGVVNFG-VPCALG-YPDGFARVSYYHDWV CIFE----SG-RSACSGDSGGPLVCGNT------LTGITSWGISSCSGS-YPSVYTRVSSFYNWV : 218 : 230 : 250 : 229 : 223 : 218 : 228 Conservazione degli amino acidi catalitici in alcuni membri della famiglia della tripsina
6 Significato strutturale dell allineamento multiplo PDBSUM of 1tlk a b c C d e f g Sequenze molto divergenti del domino immunoglobulinico allineate manualmente sulla base dell informazione della struttura di uno dei membri della famiglia (PDB 1tlk). Si noti la presenza di inserzioni o delezioni soprattutto in regioni corrispondenti a loop. Due residui di cisteina che formano un ponte disolfuro nel core idrofobico della proteina sono conservati in tutti i componenti della famiglia.
7 Informazioni strutturali ottenibili da un allineamento multiplo Regioni maggiormente conservate corrispondono probabilmente al sito attivo. Regioni ricche di inserzioni e delezioni corrispondono a loops. Posizioni invarianti con glicine o proline invarianti corrispondono spesso a turn Un pattern conservato di residui idrofobici alternati a residui idrofilici suggerisce un filamento beta superficiale. Un pattern conservato di residui idrofobici ogni 4 residui suggerisce l'esistenza di un alfa elica. Coppie di cisteine invarianti formano spesso ponti disolfuro (in particolare nelle proteine secrete).
8 Significato evolutivo dell allineamento multiplo In linea di principio esiste sempre un allineamento evolutivamente corretto. Tuttavia, non esiste un riferimento indipendente (come nel caso delle strutture cristallografiche), e la storia evolutiva delle sequenze deve anzi essere dedotta dall allineamento
9 Difficoltà dell allineamento multiplo Complessità del problema - Tempo di elaborazione Funzione oggettiva di punteggio - Peso da assegnare alle varie sequenze
10 Complessità dell allineamento multiplo Sulla superficie del cubo si hanno le matrici di confronto a coppie tra le sequenze A-B, B-C e A-C. L allineamento ottimale di tre sequenze (A-B-C) richiede il riempimento del cubo e la valutazione di tutte le possibili mosse all interno del cubo. La complessità di questo algoritmo è O(L N ), dove L è la lunghezza e N il numero delle sequenze. Per tre sequenze di 300 amino acidi il numero di confronti è 2.7 x Un algoritmo completo di programmazione dinamica è utilizzabile solo nel caso di tre sequenze.
11 Metodi per l allineamento multiplo Allineamento ottimale Programmazione dinamica (MSA) Allineamento euristico Progressivo globale (CLUSTALW,Pileup) locale (PIMA) Iterativo globale (PRRP) locale (DIALIGN)
12 Allineamento progressivo Allineamento di tre sequenze A, B, C per passi successivi Allineamento della coppia A-B DGEKFGPPQRSGQRSG.. DGERFGP-QRSGNRSG SeqA SeqB Aggiunta della coppia C al precedente allineamento consenso DHEKFGSSQRSGQRSG SeqC Allineamento multiplo DGEKFGPPQRSGQRSG.. DGERFGP-QRSGNRSG.... DHEKFGSSQRSGQRSG SeqA SeqB SeqC
13 Allineamento progressivo L'ordine di aggiunta delle sequenze è molto importante Il primo passo di un allineamento consiste nella scelta dell'ordine di aggiunta delle sequenze. Per questo vengono fatti tutti i possibili confronti a coppie (N*(N-1)/2) e viene costruito un albero per rappresentare le somiglianze. L'albero stabilisce l'ordine di aggiunta delle sequenze
14 Allineamento progressivo: CLUSTAL Higgins & Sharp 1988 Matrice di distanza ottenuta con confronti a coppie Albero (dendrogramma) di somiglianze costruito dalla matrice Allineamento progressivo delle coppie (sequenzasequenza, sequenzaprofilo, profilo -profilo) utilizzando l albero come guida
15 CLUSTALW e CLUSTALX Allineamento delle globine ottenuto con CLUSTALW CLUSTALX. Stesso algoritmo con interfaccia grafica (PC)
16 Allineamento iterativo Il primo allineamento multiplo viene usato per predire un nuovo albero, nuovi pesi e nuovi allineamenti fino a che non si ha più un miglioramento nel punteggio dell allineamento. Implementato in PRRP,DIALIGN
17 Visualizzare ed editare allineamenti: GeneDoc
18 Rappresentazioni dell allineamento multiplo: conservazione
19 Rappresentazioni dell allineamento multiplo: sostituzioni
20 Misure quantitative della conservazione dei residui in un A.M. Sequence logos Profili Modelli markoviani (profili HMM)
21 Sequence logos Rappresentazione grafica del grado di conservazione delle colonne dell allineamento. Nel grafico è rappresentata in ordinata la conservazione come diminuzione di incertezza (bits). La misura dell'incertezza è proporzionale al logaritmo in base 2 del numero di simboli. La massima conservazione in bits è 2 per i nucleotidi e 4.32 per gli amino acidi.
22 Profili Sequenza di consenso dell allineamento HBA_HUMAN...vga--hagey... HBB_HUMAN...v----nvdev... MYG_PHYCA...vea--dvag-... GLB3_CHITP...vkg------d... GLB5_PETMA...vys--tyets... LGB2_LUPLU...fna--nipkh... GLB1_GLYDI...iagadngagv... Penalità apertura gap Gribskov et al 1987 Penalità allungamento gap Cons A C D E F G H I K L M N P Q R S T V W Y Gap Len V A A A D N V A E V Un profilo rappresenta l informazione di un allineamento multiplo assegnando a ciascuna colonna dell allineamento punteggi specifici per ciascun amino acido e per i gap.
23 Uso dei profili per la ricerca di omologia: PSI-Blast PSI-Blast= Position Specific Iterated Blast 1) Ricerca di omologia con una sequenza 2) Costruzione di un profilo con le sequenze significativamente simili 3) Nuova ricerca di omologia utilizzando il profilo
24 Profili HMM (hidden Markov models) inserzione match delezione I modelli markoviani sono rappresentazioni matematiche di un allineamento. Gli HMM possono essere usati per modellare allineamenti multipli di sequenze di una data famiglia (profili HMM). Forniscono una descrizione più accurata delle probabilità associate ad un allineamento multiplo.
25 Protein family PFAM (acronimo di Protein Families) è un database di domini di proteine descritti con modelli markoviani. E diviso in due sezioni: pfam-a contiene allineamenti curati da esperti; pfam-b contiene sequenze che vengono automaticamente raggruppate.
26 Pfam
27 Pfam: family description
28 Ricerca di omologia in Pfam: anatomia delle proteine
Allineamento multiplo
 Allineamento multiplo Allineamenti multipli Vs. allineamenti a coppie A 1: 2: 3: 4: 5: 6: B 1: 2: 3: 4: 5: 6: Significato biologico dell allineamento multiplo L allineamento multiplo riassume La storia
Allineamento multiplo Allineamenti multipli Vs. allineamenti a coppie A 1: 2: 3: 4: 5: 6: B 1: 2: 3: 4: 5: 6: Significato biologico dell allineamento multiplo L allineamento multiplo riassume La storia
Allineamento multiplo
 Allineamento multiplo Allineamenti multipli Il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione analoga. I siti funzionalmente o strutturalmente
Allineamento multiplo Allineamenti multipli Il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione analoga. I siti funzionalmente o strutturalmente
Le sequenze consenso
 Le sequenze consenso Si definisce sequenza consenso una sequenza derivata da un multiallineamento che presenta solo i residui più conservati per ogni posizione riassume un multiallineamento. non è identica
Le sequenze consenso Si definisce sequenza consenso una sequenza derivata da un multiallineamento che presenta solo i residui più conservati per ogni posizione riassume un multiallineamento. non è identica
Allineamenti Multipli di Sequenze
 Allineamenti Multipli di Sequenze 1 Allineamento multiplo di sequenze: obiettivi di oggi Definire un allineamento multiplo di sequenze; com è generato; comprendere i principali metodi. Introdurre i database
Allineamenti Multipli di Sequenze 1 Allineamento multiplo di sequenze: obiettivi di oggi Definire un allineamento multiplo di sequenze; com è generato; comprendere i principali metodi. Introdurre i database
SAGA: sequence alignment by genetic algorithm. ALESSANDRO PIETRELLI Soft Computing
 SAGA: sequence alignment by genetic algorithm ALESSANDRO PIETRELLI Soft Computing Bologna, 25 Maggio 2007 Multi Allineamento di Sequenze (MSAs) Cosa sono? A cosa servono? Come vengono calcolati Multi Allineamento
SAGA: sequence alignment by genetic algorithm ALESSANDRO PIETRELLI Soft Computing Bologna, 25 Maggio 2007 Multi Allineamento di Sequenze (MSAs) Cosa sono? A cosa servono? Come vengono calcolati Multi Allineamento
Perché considerare la struttura 3D di una proteina
 Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
Algoritmi di Allineamento
 Algoritmi di Allineamento CORSO DI BIOINFORMATICA Corso di Laurea in Biotecnologie Università Magna Graecia Catanzaro Outline Similarità Allineamento Omologia Allineamento di Coppie di Sequenze Allineamento
Algoritmi di Allineamento CORSO DI BIOINFORMATICA Corso di Laurea in Biotecnologie Università Magna Graecia Catanzaro Outline Similarità Allineamento Omologia Allineamento di Coppie di Sequenze Allineamento
Laboratorio di Informatica 2004/ 2005 Corso di laurea in biotecnologie - Novara Viviana Patti Esercitazione 7 2.
 Laboratorio di Informatica 2004/ 2005 Corso di laurea in biotecnologie - Novara Viviana Patti patti@di.unito.it Esercitazione 7 1 Info&Bio Bio@Lab Allineamento di sequenze Esercitazione 7 2 1 Es2: Allineamento
Laboratorio di Informatica 2004/ 2005 Corso di laurea in biotecnologie - Novara Viviana Patti patti@di.unito.it Esercitazione 7 1 Info&Bio Bio@Lab Allineamento di sequenze Esercitazione 7 2 1 Es2: Allineamento
Allineamento multiplo di sequenze
 Allineamento multiplo di sequenze Bioinformatica a.a. 2008/2009 Letterio Galletta Università di Pisa 22 Maggio 2009 Letterio Galletta (Università di Pisa) Allineamento multiplo di sequenze 22 Maggio 2009
Allineamento multiplo di sequenze Bioinformatica a.a. 2008/2009 Letterio Galletta Università di Pisa 22 Maggio 2009 Letterio Galletta (Università di Pisa) Allineamento multiplo di sequenze 22 Maggio 2009
ALLINEAMENTI MULTIPLI
 ALLINEAMENTI MULTIPLI Identificazione di siti funzionalmente importanti Dimostrazione di omologia Filogenesi molecolare Ricerca di somiglianze deboli ma significative in banche dati Predizione di struttura
ALLINEAMENTI MULTIPLI Identificazione di siti funzionalmente importanti Dimostrazione di omologia Filogenesi molecolare Ricerca di somiglianze deboli ma significative in banche dati Predizione di struttura
FASTA: Lipman & Pearson (1985) BLAST: Altshul (1990) Algoritmi EURISTICI di allineamento
 Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Ricerca di omologia di sequenza
 Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Allineamenti multipli
 Allineamenti multipli Allineamenti multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte
Allineamenti multipli Allineamenti multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte
Relazione sequenza-struttura e funzione
 Biotecnologie applicate alla progettazione e sviluppo di molecole biologicamente attive A.A. 2010-2011 Modulo di Biologia Strutturale Relazione sequenza-struttura e funzione Marco Nardini Dipartimento
Biotecnologie applicate alla progettazione e sviluppo di molecole biologicamente attive A.A. 2010-2011 Modulo di Biologia Strutturale Relazione sequenza-struttura e funzione Marco Nardini Dipartimento
Alberi filogenetici. File: alberi_filogenetici.odp. Riccardo Percudani 02/03/04
 Alberi filogenetici The tree of life Albero filogenetico costruito con le sequenze della subunità piccola dell RNA ribosomale. Tutte le forme viventi condividono un comune ancestore (LCA, last common ancestor
Alberi filogenetici The tree of life Albero filogenetico costruito con le sequenze della subunità piccola dell RNA ribosomale. Tutte le forme viventi condividono un comune ancestore (LCA, last common ancestor
Banche Dati proteiche
 Banche Dati proteiche Un altro grande database è UniProt, The Universal Protein Resource (http://www.uniprot.org/) nel quale sono radunate le sequenze proteiche, e le annotazione delle stesse, ottenute
Banche Dati proteiche Un altro grande database è UniProt, The Universal Protein Resource (http://www.uniprot.org/) nel quale sono radunate le sequenze proteiche, e le annotazione delle stesse, ottenute
Allineamenti Multipli
 Allineamenti Multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione
Allineamenti Multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione
Allineamenti multipli
 Allineamenti multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione
Allineamenti multipli Finora ci siamo occupati di allineamenti a coppie (pairwise), ma il modo migliore per conoscere le caratteristiche di una determinata famiglia è allineare molte proteine a funzione
ALLINEAMENTO DI SEQUENZE
 ALLINEAMENTO DI SEQUENZE Procedura per comparare due o piu sequenze, volta a stabilire un insieme di relazioni biunivoche tra coppie di residui delle sequenze considerate che massimizzino la similarita
ALLINEAMENTO DI SEQUENZE Procedura per comparare due o piu sequenze, volta a stabilire un insieme di relazioni biunivoche tra coppie di residui delle sequenze considerate che massimizzino la similarita
FASTA. Lezione del
 FASTA Lezione del 10.03.2016 Omologia vs Similarità Quando si confrontano due sequenze o strutture si usano spesso indifferentemente i termini somiglianza o omologia per indicare che esiste un rapporto
FASTA Lezione del 10.03.2016 Omologia vs Similarità Quando si confrontano due sequenze o strutture si usano spesso indifferentemente i termini somiglianza o omologia per indicare che esiste un rapporto
Esempio di utilizzo del programma BLAST disponibile all NCBI Form di Nucleotide BLAST
 Esempio di utilizzo del programma BLAST disponibile all NCBI www.ncbi.nlm.nih.gov/blast Form di Nucleotide BLAST Per un uso più avanzato, si possono impostare parametri particolari (es. cost to open gap,
Esempio di utilizzo del programma BLAST disponibile all NCBI www.ncbi.nlm.nih.gov/blast Form di Nucleotide BLAST Per un uso più avanzato, si possono impostare parametri particolari (es. cost to open gap,
Bioinformatica ed applicazioni di bioinformatica strutturale!
 Bioinformatica ed applicazioni di bioinformatica strutturale! Bioinformatica! Le banche dati! Programmi per estrarre ed analizzare i dati! I numeri! Cellule nell uomo! Geni nell uomo! Genoma umano Il dogma
Bioinformatica ed applicazioni di bioinformatica strutturale! Bioinformatica! Le banche dati! Programmi per estrarre ed analizzare i dati! I numeri! Cellule nell uomo! Geni nell uomo! Genoma umano Il dogma
Lezione 2 (10/03/2010): Allineamento di sequenze (parte 1) Antonella Meloni:
 Lezione 2 (10/03/2010): Allineamento di sequenze (parte 1) Antonella Meloni: antonella.meloni@ifc.cnr.it Sequenza A= stringa formata da N simboli, dove i simboli apparterranno ad un certo alfabeto. A
Lezione 2 (10/03/2010): Allineamento di sequenze (parte 1) Antonella Meloni: antonella.meloni@ifc.cnr.it Sequenza A= stringa formata da N simboli, dove i simboli apparterranno ad un certo alfabeto. A
sono le unità monomeriche che costituiscono le proteine hanno tutti una struttura comune
 AMINO ACIDI sono le unità monomeriche che costituiscono le proteine sono 20 hanno tutti una struttura comune sono asimmetrici La carica di un amino acido dipende dal ph Classificazione amino acidi Glicina
AMINO ACIDI sono le unità monomeriche che costituiscono le proteine sono 20 hanno tutti una struttura comune sono asimmetrici La carica di un amino acido dipende dal ph Classificazione amino acidi Glicina
Come si sceglie l algoritmo di allineamento? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione?
 Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Esercizio: Ricerca di sequenze in banche dati e allineamento multiplo (adattato da una lezione del Prof. Paiardini)
 Esercizio: Ricerca di sequenze in banche dati e allineamento multiplo (adattato da una lezione del Prof. Paiardini) Collegatevi al sito www.ncbi.nlm.nih.gov/blast. Apparirà una pagina nella quale le versioni
Esercizio: Ricerca di sequenze in banche dati e allineamento multiplo (adattato da una lezione del Prof. Paiardini) Collegatevi al sito www.ncbi.nlm.nih.gov/blast. Apparirà una pagina nella quale le versioni
BLAST. W = word size T = threshold X = elongation S = HSP threshold
 BLAST Blast (Basic Local Aligment Search Tool) è un programma che cerca similarità locali utilizzando l algoritmo di Altschul et al. Anche Blast, come FASTA, funziona: 1. scomponendo la sequenza query
BLAST Blast (Basic Local Aligment Search Tool) è un programma che cerca similarità locali utilizzando l algoritmo di Altschul et al. Anche Blast, come FASTA, funziona: 1. scomponendo la sequenza query
ALLINEAMENTI MULTIPLI
 ALLINEAMENTI MULTIPLI Allineamento progressivo (Clustal) Metodi iterativi (Multalin) Metodi basati su zone comuni di sequenza conservate (Profili) Metodi statistici e modelli probabilistici (HMM) Motivazione
ALLINEAMENTI MULTIPLI Allineamento progressivo (Clustal) Metodi iterativi (Multalin) Metodi basati su zone comuni di sequenza conservate (Profili) Metodi statistici e modelli probabilistici (HMM) Motivazione
q xi Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p i >0;
 Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
Quarta lezione. 1. Ricerca di omologhe in banche dati. 2. Programmi per la ricerca: FASTA BLAST
 Quarta lezione 1. Ricerca di omologhe in banche dati. 2. Programmi per la ricerca: FASTA BLAST Ricerca di omologhe in banche dati Proteina vs. proteine Gene (traduzione in aa) vs. proteine Gene vs. geni
Quarta lezione 1. Ricerca di omologhe in banche dati. 2. Programmi per la ricerca: FASTA BLAST Ricerca di omologhe in banche dati Proteina vs. proteine Gene (traduzione in aa) vs. proteine Gene vs. geni
Metodi di Distanza. G.Allegrucci riproduzione vietata
 Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
Lezione 8. Ricerche in banche dati (databases) attraverso l uso di BLAST
 Lezione 8 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Lezione 8 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Lezione 8. Ricerche in banche dati (databases) attraverso l uso di BLAST
 Lezione 8 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Lezione 8 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Omologia di sequenze: allineamento e ricerca
 Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
Bellini Lara matricola: Tesina di Biologia Molecolare 2
 Bellini Lara matricola: 594736 Tesina di Biologia Molecolare 2 Argomento: Scegli una proteina di Drosophila e trovala in Uniprot.Descrivi le informazioni presenti nel record ed i collegamenti a risorse
Bellini Lara matricola: 594736 Tesina di Biologia Molecolare 2 Argomento: Scegli una proteina di Drosophila e trovala in Uniprot.Descrivi le informazioni presenti nel record ed i collegamenti a risorse
ALGORITMI DI OTTIMIZZAZIONE M Esercizi Parte I
 ALGORITMI DI OTTIMIZZAZIONE M Esercizi Parte I Esercizio 1 Dati n oggetti ed un contenitore, ad ogni oggetto j (j = 1,, n) sono associati un peso p j ed un costo c j (con p j e c j interi positivi). Si
ALGORITMI DI OTTIMIZZAZIONE M Esercizi Parte I Esercizio 1 Dati n oggetti ed un contenitore, ad ogni oggetto j (j = 1,, n) sono associati un peso p j ed un costo c j (con p j e c j interi positivi). Si
Allineamenti di sequenze: concetti e algoritmi
 Allineamenti di sequenze: concetti e algoritmi 1 globine: a- b- mioglobina Precoce esempio di allineamento di sequenza: globine (1961) H.C. Watson and J.C. Kendrew, Comparison Between the Amino-Acid Sequences
Allineamenti di sequenze: concetti e algoritmi 1 globine: a- b- mioglobina Precoce esempio di allineamento di sequenza: globine (1961) H.C. Watson and J.C. Kendrew, Comparison Between the Amino-Acid Sequences
Riconoscimento e recupero dell informazione per bioinformatica
 Riconoscimento e recupero dell informazione per bioinformatica Hidden Markov Models Manuele Bicego orso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Processi
Riconoscimento e recupero dell informazione per bioinformatica Hidden Markov Models Manuele Bicego orso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Processi
Sommario. Presentazione dell opera Ringraziamenti
 Sommario Presentazione dell opera Ringraziamenti XI XII Capitolo 1 Introduzione alla bioinformatica 1 1.1 Cenni introduttivi 1 1.2 Pietre miliari della bioinformatica 2 1.3 Infrastrutture bioinformatiche
Sommario Presentazione dell opera Ringraziamenti XI XII Capitolo 1 Introduzione alla bioinformatica 1 1.1 Cenni introduttivi 1 1.2 Pietre miliari della bioinformatica 2 1.3 Infrastrutture bioinformatiche
Percorsi di chimica organica - Soluzioni degli esercizi del testo
 ercorsi di chimica organica - Soluzioni degli esercizi del testo AITL 14 1. Il prefisso α negli α-amminoacidi sta ad indicare che il gruppo amminico, - 2, si trova sul carbonio alfa (carbonio legato al
ercorsi di chimica organica - Soluzioni degli esercizi del testo AITL 14 1. Il prefisso α negli α-amminoacidi sta ad indicare che il gruppo amminico, - 2, si trova sul carbonio alfa (carbonio legato al
E il server più utilizzato, permette di tracciare tutte le operazioni che svolge e di impostare alcuni parametri importanti per il risultato finale.
 Homology Modelling Homology modelling L omology modeling delle proteine è il tipo di predizione di struttura terziaria più semplice ed affidabile. Viene richiesta soltanto una (o più) sequenze di riferimento
Homology Modelling Homology modelling L omology modeling delle proteine è il tipo di predizione di struttura terziaria più semplice ed affidabile. Viene richiesta soltanto una (o più) sequenze di riferimento
BLAST: Basic Local Alignment Search Tool
 BLAST: Basic Local Alignment Search Tool 1 Outline della lezione di oggi BLAST Uso pratico Algoritmo Strategie Trovare proteine lontanamente legate: PSI-BLAST 2 Problema con gli algoritmi dinamici Gli
BLAST: Basic Local Alignment Search Tool 1 Outline della lezione di oggi BLAST Uso pratico Algoritmo Strategie Trovare proteine lontanamente legate: PSI-BLAST 2 Problema con gli algoritmi dinamici Gli
UNIVERSITÀ DEGLI STUDI DI MILANO. Bioinformatica. A.A semestre I. Allineamento veloce (euristiche)
 Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre I 3 Allineamento veloce (euristiche) Banche dati primarie e secondarie Esistono due categorie
Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre I 3 Allineamento veloce (euristiche) Banche dati primarie e secondarie Esistono due categorie
Lezione 6. Ricerche in banche dati (databases) attraverso l uso di BLAST
 Lezione 6 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Lezione 6 Ricerche in banche dati (databases) attraverso l uso di BLAST BLAST: Basic Local Alignment Search Tool Basic Local Alignment Search Tool. Altschul et al. 1990,1994,1997 Sviluppato per rendere
Università degli studi di Pisa
 Università degli studi di Pisa Nicola Guido PATTERNHUNTER: Faster and More Sensitive. Homology Search Seminario: Bioinformatica a.a. 2008/2009 Contenuto della presentazione Introduzione Scenario PatternHunter
Università degli studi di Pisa Nicola Guido PATTERNHUNTER: Faster and More Sensitive. Homology Search Seminario: Bioinformatica a.a. 2008/2009 Contenuto della presentazione Introduzione Scenario PatternHunter
Allineamento multiplo di sequenze
 Allineamento multiplo di sequenze Nicola Vitacolonna vitacolo@dimi.uniud.it http://www.dimi.uniud.it/~vitacolo Università degli Studi di Udine 16 aprile 2002 Two homologous sequences whisper... a full
Allineamento multiplo di sequenze Nicola Vitacolonna vitacolo@dimi.uniud.it http://www.dimi.uniud.it/~vitacolo Università degli Studi di Udine 16 aprile 2002 Two homologous sequences whisper... a full
Allineamenti a coppie
 Laboratorio di Bioinformatica I Allineamenti a coppie Dott. Sergio Marin Vargas (2014 / 2015) ExPASy Bioinformatics Resource Portal (SIB) http://www.expasy.org/ Il sito http://myhits.isb-sib.ch/cgi-bin/dotlet
Laboratorio di Bioinformatica I Allineamenti a coppie Dott. Sergio Marin Vargas (2014 / 2015) ExPASy Bioinformatics Resource Portal (SIB) http://www.expasy.org/ Il sito http://myhits.isb-sib.ch/cgi-bin/dotlet
Analisi della struttura primaria delle proteine
 Analisi della struttura primaria delle proteine Strumenti on-line La maggior parte degli strumenti per l analisi della struttura primaria si trovano on-line all indirizzo www.expasy.org Ottenere la sequenza
Analisi della struttura primaria delle proteine Strumenti on-line La maggior parte degli strumenti per l analisi della struttura primaria si trovano on-line all indirizzo www.expasy.org Ottenere la sequenza
PROTEINE DEFINIZIONE:
 Cap.4 Le PROTEINE DEFINIZIONE: Macromolecole formate di AA della serie L uniti tra loro da un legame peptidico. FUNZIONI DELLE PROTEINE Enzimi Proteine di riconoscimento Proteine di trasporto Proteine
Cap.4 Le PROTEINE DEFINIZIONE: Macromolecole formate di AA della serie L uniti tra loro da un legame peptidico. FUNZIONI DELLE PROTEINE Enzimi Proteine di riconoscimento Proteine di trasporto Proteine
MODELLO SCHEDA INSEGNAMENTO. II II Luigi Cerulo
 Corso di L/LM/LMCU Denominazione insegnamento: MODELLO SCHEDA INSEGNAMENTO Numero di Crediti: 6 Anno: Semestre: Docente Titolare: Scienze e Tecnologie Genetiche Bioinformatica II II Luigi Cerulo Dottorandi/assegnisti
Corso di L/LM/LMCU Denominazione insegnamento: MODELLO SCHEDA INSEGNAMENTO Numero di Crediti: 6 Anno: Semestre: Docente Titolare: Scienze e Tecnologie Genetiche Bioinformatica II II Luigi Cerulo Dottorandi/assegnisti
Computazione per l interazione naturale: Modelli dinamici
 Computazione per l interazione naturale: Modelli dinamici Corso di Interazione uomo-macchina II Prof. Giuseppe Boccignone Dipartimento di Scienze dell Informazione Università di Milano boccignone@dsi.unimi.it
Computazione per l interazione naturale: Modelli dinamici Corso di Interazione uomo-macchina II Prof. Giuseppe Boccignone Dipartimento di Scienze dell Informazione Università di Milano boccignone@dsi.unimi.it
4. Ricerca di sequenze in banche dati e allineamento multiplo
 4. Ricerca di sequenze in banche dati e allineamento multiplo Collegatevi al sito www.ncbi.nlm.nih.gov/blast. Apparirà una pagina nella quale le versioni di BLAST disponibili sono organizzate in base al
4. Ricerca di sequenze in banche dati e allineamento multiplo Collegatevi al sito www.ncbi.nlm.nih.gov/blast. Apparirà una pagina nella quale le versioni di BLAST disponibili sono organizzate in base al
Pairwise Sequence Alignment BIOINFORMATICA. Corso di Laurea in Ingegneria Informatica e Biomedica. Università Magna Graecia Catanzaro
 Pairwise Sequence Alignment BIOINFORMATICA Corso di Laurea in Ingegneria Informatica e Biomedica Università Magna Graecia Catanzaro Outline Similarità Allineamento Omologia Allineamento Esatto di Coppie
Pairwise Sequence Alignment BIOINFORMATICA Corso di Laurea in Ingegneria Informatica e Biomedica Università Magna Graecia Catanzaro Outline Similarità Allineamento Omologia Allineamento Esatto di Coppie
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Alcol + alcol etere R-OH + R -OH R-O-R + H 2 O Aldeide + alcol emiacetale R-CHO + R -OH R-CHOH-O-R Acido + Acido anidride R-COOH + R -COOH
 Nomenclatura AMIDI Alcol + alcol etere R-OH + R -OH R-O-R + H 2 O Aldeide + alcol emiacetale R-CHO + R -OH R-CHOH-O-R Acido + Acido anidride R-COOH + R -COOH R-CO-O-CO-R + H 2 O Alcol + Acido estere R-COOH
Nomenclatura AMIDI Alcol + alcol etere R-OH + R -OH R-O-R + H 2 O Aldeide + alcol emiacetale R-CHO + R -OH R-CHOH-O-R Acido + Acido anidride R-COOH + R -COOH R-CO-O-CO-R + H 2 O Alcol + Acido estere R-COOH
LE PROTEINE. SONO Polimeri formati dall unione di AMMINOACIDI (AA) Rende diversi i 20 AA l uno dall altro UN ATOMO DI C AL CENTRO
 LE PROTEINE SONO Polimeri formati dall unione di ATOMI DI C, H, N, O CHE SONO AMMINOACIDI (AA) Uniti tra loro dal Legame peptidico 20 TIPI DIVERSI MA HANNO STESSA STRUTTURA GENERALE CON Catene peptidiche
LE PROTEINE SONO Polimeri formati dall unione di ATOMI DI C, H, N, O CHE SONO AMMINOACIDI (AA) Uniti tra loro dal Legame peptidico 20 TIPI DIVERSI MA HANNO STESSA STRUTTURA GENERALE CON Catene peptidiche
La distribuzione dei veri e falsi positivi la ricerca della giusta soglia
 La distribuzione dei veri e falsi positivi la ricerca della giusta soglia BLAST Blast (Basic Local Aligment Search Tool) è un programma che cerca similarità locali utilizzando l algoritmo di Altschul et
La distribuzione dei veri e falsi positivi la ricerca della giusta soglia BLAST Blast (Basic Local Aligment Search Tool) è un programma che cerca similarità locali utilizzando l algoritmo di Altschul et
Informatica e biotecnologie
 Informatica e biotecnologie Ricerca di informazioni e analisi di sequenze CGCTTCGGACGAAATCGCATCAGCATACGATCGCATGCCGGGCGGGATAAC CGAAATCGCATCAGCATACGATCGCATGC Informatica e biotecnologie Strumenti per raccogliere
Informatica e biotecnologie Ricerca di informazioni e analisi di sequenze CGCTTCGGACGAAATCGCATCAGCATACGATCGCATGCCGGGCGGGATAAC CGAAATCGCATCAGCATACGATCGCATGC Informatica e biotecnologie Strumenti per raccogliere
Riconoscimento e recupero dell informazione per bioinformatica
 Riconoscimento e recupero dell informazione per bioinformatica Hidden Markov Models Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Processi
Riconoscimento e recupero dell informazione per bioinformatica Hidden Markov Models Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Processi
Bioinformatics more basic notions
 Bioinformatics more basic notions Alcune slides provengono dal materiale rilasciato da: Dr Sergio Marin Vargas - Verona Prof. Riccardo Percudari - Parma Bioinformatics Bio-inspired Computer science Gli
Bioinformatics more basic notions Alcune slides provengono dal materiale rilasciato da: Dr Sergio Marin Vargas - Verona Prof. Riccardo Percudari - Parma Bioinformatics Bio-inspired Computer science Gli
A W T V A S A V R T S I A Y T V A A A V R T S I A Y T V A A A V L T S I
 COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
RICERCA DI PATTERN E DI MOTIVI DEFINIZIONE DI MOTIVO
 RICERCA DI PATTERN E DI MOTIVI Uno dei primi scopi della biologia computazionale consiste nel rispondere alla domanda: data una nuova sequenza, cosa si può dire sulla funzione, o sulle funzioni, in essa
RICERCA DI PATTERN E DI MOTIVI Uno dei primi scopi della biologia computazionale consiste nel rispondere alla domanda: data una nuova sequenza, cosa si può dire sulla funzione, o sulle funzioni, in essa
Costituenti chimici della materia vivente
 Costituenti chimici della materia vivente Le macromolecole biologiche Macromolecole (dal greco macros = grande) biologiche. Classi di composti biologici multifunzionali: Polisaccaridi proteine acidi
Costituenti chimici della materia vivente Le macromolecole biologiche Macromolecole (dal greco macros = grande) biologiche. Classi di composti biologici multifunzionali: Polisaccaridi proteine acidi
Ricerche con BLAST (Laboratorio)
 Laboratorio di Bioinformatica I Ricerche con BLAST (Laboratorio) Dott. Sergio Marin Vargas (2014 / 2015) NCBI BLAST BLAST: Basic Local Alignment Search Tool http://blast.ncbi.nlm.nih.gov/blast.cgi NCBI
Laboratorio di Bioinformatica I Ricerche con BLAST (Laboratorio) Dott. Sergio Marin Vargas (2014 / 2015) NCBI BLAST BLAST: Basic Local Alignment Search Tool http://blast.ncbi.nlm.nih.gov/blast.cgi NCBI
La struttura covalente delle proteine (la sequenza amminoacidica)
 La struttura covalente delle proteine (la sequenza amminoacidica) Sequenza amminoacidica dell ormone insulina bovino (Frederick Sanger, 1953) Il primo passo per determinare la sequenza di un peptide è
La struttura covalente delle proteine (la sequenza amminoacidica) Sequenza amminoacidica dell ormone insulina bovino (Frederick Sanger, 1953) Il primo passo per determinare la sequenza di un peptide è
La ricerca di similarità in banche dati
 La ricerca di similarità in banche dati Uno dei problemi più comunemente affrontati con metodi bioinformatici è quello di trovare omologie di sequenza interrogando una banca dati. L idea di base è che
La ricerca di similarità in banche dati Uno dei problemi più comunemente affrontati con metodi bioinformatici è quello di trovare omologie di sequenza interrogando una banca dati. L idea di base è che
Riconoscimento e recupero dell informazione per bioinformatica
 Riconoscimento e recupero dell informazione per bioinformatica Clustering: introduzione Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Una definizione
Riconoscimento e recupero dell informazione per bioinformatica Clustering: introduzione Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Una definizione
Relazione Laboratorio di bioinformatica
 Relazione Laboratorio di bioinformatica Davide Cittaro La predizione della struttura tridimensionale di una proteina sulla base della sequenza è un risultato ambito. L utilizzo di sole considerazioni fisico-chimiche
Relazione Laboratorio di bioinformatica Davide Cittaro La predizione della struttura tridimensionale di una proteina sulla base della sequenza è un risultato ambito. L utilizzo di sole considerazioni fisico-chimiche
Indice generale. Nozioni fondamentali. Prefazione XIII
 Prefazione XIII A Nozioni fondamentali CAPITOLO 1 La biologia essenziale 3 1.1 Genomi, genomica e avvento della Bioinformatica 3 1.2 Genoma dei procarioti 5 1.2.1 Struttura e dimensioni 5 1.2.2 Proprietà
Prefazione XIII A Nozioni fondamentali CAPITOLO 1 La biologia essenziale 3 1.1 Genomi, genomica e avvento della Bioinformatica 3 1.2 Genoma dei procarioti 5 1.2.1 Struttura e dimensioni 5 1.2.2 Proprietà
Z-score. lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random
 Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Corso di Biochimica computazionale LS Ecologia. Analisi informatica di una proteina ipotetica
 Corso di Biochimica computazionale Greta Gandolfi LS Ecologia Analisi informatica di una proteina ipotetica ID proteina: EAA74842 Ricerca in banca dati NCBI (http://www.ncbi.nlm.nih.gov/genbank/index.html)
Corso di Biochimica computazionale Greta Gandolfi LS Ecologia Analisi informatica di una proteina ipotetica ID proteina: EAA74842 Ricerca in banca dati NCBI (http://www.ncbi.nlm.nih.gov/genbank/index.html)
BIOLOGIA MOLECOLARE CON ELEMENTI DI BIOINFORMATICA
 DIPARTIMENTO DI SCIENZE BIOLOGICHE, GEOLOGICHE E AMBIENTALI Corso di laurea magistrale in Biologia sanitaria e cellularemolecolare Anno accademico 2017/2018-1 anno - Curriculum Biologia cellulare e molecolare
DIPARTIMENTO DI SCIENZE BIOLOGICHE, GEOLOGICHE E AMBIENTALI Corso di laurea magistrale in Biologia sanitaria e cellularemolecolare Anno accademico 2017/2018-1 anno - Curriculum Biologia cellulare e molecolare
Le proteine sono polimeri lineari costituiti da unità base formate da oltre 40 amminoacidi. Possono assumere forme diverse a seconda della funzione
 Le proteine sono polimeri lineari costituiti da unità base formate da oltre 40 amminoacidi Hanno elevato PM Possono assumere forme diverse a seconda della funzione svolgono molteplici funzioni Tra le proteine
Le proteine sono polimeri lineari costituiti da unità base formate da oltre 40 amminoacidi Hanno elevato PM Possono assumere forme diverse a seconda della funzione svolgono molteplici funzioni Tra le proteine
ALLINEAMENTI MULTIPLI
 ALLINEAMENTI MULTIPLI Allineamento progressivo (Clustal) Metodi iterativi (Multalin) Metodi basati su zone comuni di sequenza conservate (Profili) Metodi statistici e modelli probabilistici (HMM) Motivazione
ALLINEAMENTI MULTIPLI Allineamento progressivo (Clustal) Metodi iterativi (Multalin) Metodi basati su zone comuni di sequenza conservate (Profili) Metodi statistici e modelli probabilistici (HMM) Motivazione
Problemi, istanze, soluzioni
 lgoritmi e Strutture di Dati II 2 Problemi, istanze, soluzioni Un problema specifica una relazione matematica tra dati di ingresso e dati di uscita. Una istanza di un problema è formata dai dati di un
lgoritmi e Strutture di Dati II 2 Problemi, istanze, soluzioni Un problema specifica una relazione matematica tra dati di ingresso e dati di uscita. Una istanza di un problema è formata dai dati di un
Complessità computazionale
 Complessità computazionale Alberto Policriti Dpt. of Mathematics and Informatics, University of Udine. Applied Genomics Institute December 16, 2009 Il problema computazionale Un problema specifica una
Complessità computazionale Alberto Policriti Dpt. of Mathematics and Informatics, University of Udine. Applied Genomics Institute December 16, 2009 Il problema computazionale Un problema specifica una
Distanza di Edit. Speaker: Antinisca Di Marco Data:
 Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
Riconoscimento e recupero dell informazione per bioinformatica
 Riconoscimento e recupero dell informazione per bioinformatica Filogenesi Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Introduzione alla
Riconoscimento e recupero dell informazione per bioinformatica Filogenesi Manuele Bicego Corso di Laurea in Bioinformatica Dipartimento di Informatica - Università di Verona Sommario Introduzione alla
Programmazione dinamica
 rogrammazione dinamica Fornisce l allineamento ottimale tra due sequenze semplici variazioni dell algoritmo producono allineamenti globali o locali l allineamento calcolato dipende dalla scelta di alcuni
rogrammazione dinamica Fornisce l allineamento ottimale tra due sequenze semplici variazioni dell algoritmo producono allineamenti globali o locali l allineamento calcolato dipende dalla scelta di alcuni
La struttura terziaria delle proteine
 La struttura terziaria delle proteine 1 La struttura terziaria L arrangiamento spaziale degli aminoacidi di una singola catena polipeptidica a formare la sua struttura tridimensionale a domini viene chiamata
La struttura terziaria delle proteine 1 La struttura terziaria L arrangiamento spaziale degli aminoacidi di una singola catena polipeptidica a formare la sua struttura tridimensionale a domini viene chiamata
Allineamento e similarità di sequenze
 Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Kangourou Italia Gara del 28 marzo 2008 Categoria Student Per studenti di quarta o quinta della secondaria di secondo grado
 Testi_08.qxp 9-0-008 :6 Pagina 8 Kangourou Italia Gara del 8 marzo 008 ategoria Per studenti di quarta o quinta della secondaria di secondo grado I quesiti dal N. al N. 0 valgono punti ciascuno. Nelle
Testi_08.qxp 9-0-008 :6 Pagina 8 Kangourou Italia Gara del 8 marzo 008 ategoria Per studenti di quarta o quinta della secondaria di secondo grado I quesiti dal N. al N. 0 valgono punti ciascuno. Nelle
Bioinformatica. Analisi del genoma
 Bioinformatica Analisi del genoma GABRIELLA TRUCCO CREMA, 5 APRILE 2017 Cosa è il genoma? Insieme delle informazioni biologiche, depositate nella sequenza di DNA, necessarie alla costruzione e mantenimento
Bioinformatica Analisi del genoma GABRIELLA TRUCCO CREMA, 5 APRILE 2017 Cosa è il genoma? Insieme delle informazioni biologiche, depositate nella sequenza di DNA, necessarie alla costruzione e mantenimento
Corso di Bioinformatica
 Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
UML Introduzione a UML Linguaggio di Modellazione Unificato. Corso di Ingegneria del Software Anno Accademico 2012/13
 UML Introduzione a UML Linguaggio di Modellazione Unificato Corso di Ingegneria del Software Anno Accademico 2012/13 1 Che cosa è UML? UML (Unified Modeling Language) è un linguaggio grafico per: specificare
UML Introduzione a UML Linguaggio di Modellazione Unificato Corso di Ingegneria del Software Anno Accademico 2012/13 1 Che cosa è UML? UML (Unified Modeling Language) è un linguaggio grafico per: specificare
LE PROTEINE -struttura tridimensionale-
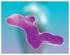 LE PROTEINE -struttura tridimensionale- Struttura generale di una proteina Ceruloplasmina Cosa sono??? Sono biopolimeri con forme ben definite. composti da molteplici amminoacidi, legati con legami peptidici
LE PROTEINE -struttura tridimensionale- Struttura generale di una proteina Ceruloplasmina Cosa sono??? Sono biopolimeri con forme ben definite. composti da molteplici amminoacidi, legati con legami peptidici
Modulo Laboratorio A.A. 2014/2015
 Biochimica - Laboratorio di Bioinformatica I (CdL. Bioinformatica) Bioinformatica e banche dati biologiche (CdL. Biotecnologie) Modulo Laboratorio A.A. 2014/2015 Docente: Dr. Sergio Marin Vargas Mail:
Biochimica - Laboratorio di Bioinformatica I (CdL. Bioinformatica) Bioinformatica e banche dati biologiche (CdL. Biotecnologie) Modulo Laboratorio A.A. 2014/2015 Docente: Dr. Sergio Marin Vargas Mail:
FONDAMENTI DI INTELLIGENZA ARTIFICIALE 13 Gennaio 2010 Tempo a disposizione 2h Risultato 32/32 punti
 FONDAMENTI DI INTELLIGENZA ARTIFICIALE Gennaio 2010 Tempo a disposizione 2h Risultato 32/32 punti Esercizio 1 (punti 6) Si modellino in logica dei predicati del I ordine le seguenti frasi (utilizzando
FONDAMENTI DI INTELLIGENZA ARTIFICIALE Gennaio 2010 Tempo a disposizione 2h Risultato 32/32 punti Esercizio 1 (punti 6) Si modellino in logica dei predicati del I ordine le seguenti frasi (utilizzando
Metodi predittivi di accessibilità
 Metodi predittivi di accessibilità ACCESSIBILITÀ AL SOLVENTE Una caratteristica interessante da predire, visto la forte dipendenza dalla sequenza, è l accessibilità al solvente dei residui che compongono
Metodi predittivi di accessibilità ACCESSIBILITÀ AL SOLVENTE Una caratteristica interessante da predire, visto la forte dipendenza dalla sequenza, è l accessibilità al solvente dei residui che compongono
Proprietà comuni. Il gruppo α-carbossilico b è un acido più forte del gruppo carbossilico degli acidi alifatici
 Gli aminoacidi Proprietà comuni Il gruppo α-carbossilico b è un acido più forte del gruppo carbossilico degli acidi alifatici paragonabili Il gruppo α-aminico è un acido più forte (o una base più debole
Gli aminoacidi Proprietà comuni Il gruppo α-carbossilico b è un acido più forte del gruppo carbossilico degli acidi alifatici paragonabili Il gruppo α-aminico è un acido più forte (o una base più debole
la edit distance tra X e Y è la distanza relativa all allineamento (o agli allineamenti) che minimizza tale distanza.
 Algoritmica 14/15 EDIT DISTANCE Il problema della edit distance (distanza di edizione, in una versione in italiano scarsamente usata) è alla base dei problemi di confronto fra sequenze perché il meccanismo
Algoritmica 14/15 EDIT DISTANCE Il problema della edit distance (distanza di edizione, in una versione in italiano scarsamente usata) è alla base dei problemi di confronto fra sequenze perché il meccanismo
Introduzione alla programmazione
 Introduzione alla programmazione Risolvere un problema Per risolvere un problema si procede innanzitutto all individuazione Delle informazioni, dei dati noti Dei risultati desiderati Il secondo passo consiste
Introduzione alla programmazione Risolvere un problema Per risolvere un problema si procede innanzitutto all individuazione Delle informazioni, dei dati noti Dei risultati desiderati Il secondo passo consiste
Paolo Mogorovich
 Sistemi Informativi Territoriali Paolo Mogorovich www.di.unipi.it/~mogorov .4 Dati vettoriali - Tecniche di rappresentazione Un layer areale può essere rappresentato utilizzando diverse tecniche. Per esempio:
Sistemi Informativi Territoriali Paolo Mogorovich www.di.unipi.it/~mogorov .4 Dati vettoriali - Tecniche di rappresentazione Un layer areale può essere rappresentato utilizzando diverse tecniche. Per esempio:
Database di sequenze. Dati di sequenza. Caratteristiche dei dati della biologia molecolare. I dati ed i problemi della bioinformatica
 I dati ed i problemi della bioinformatica Giorgio Valentini DSI Università degli Studi di Milano 1 Caratteristiche dei dati della biologia molecolare Diverse tipologie di dati bio-molecolari Per ogni tipo
I dati ed i problemi della bioinformatica Giorgio Valentini DSI Università degli Studi di Milano 1 Caratteristiche dei dati della biologia molecolare Diverse tipologie di dati bio-molecolari Per ogni tipo
a) un movimento contro gradiente di concentrazione che utilizza fonti primarie di energia
 1. Quale considerazione sulla struttura primaria di una proteina è vera? a) è caratteristica delle proteine insolubili b) i ponti S-S la stabilizzano c) i ponti H la stabilizzano d) la proteina assume
1. Quale considerazione sulla struttura primaria di una proteina è vera? a) è caratteristica delle proteine insolubili b) i ponti S-S la stabilizzano c) i ponti H la stabilizzano d) la proteina assume
I programmi Assembler e Linker
 Testo di riferimento: [Congiu] 10.1-10.5 (pg. 267 291) 11.a I programmi Assembler e Linker Produzione del software Istruzioni, direttive, simboli, ecc. Processo di produzione del software Analisi del problema
Testo di riferimento: [Congiu] 10.1-10.5 (pg. 267 291) 11.a I programmi Assembler e Linker Produzione del software Istruzioni, direttive, simboli, ecc. Processo di produzione del software Analisi del problema
UNIVERSITÀ DEGLI STUDI DI MILANO. Bioinformatica. A.A semestre II. 4 Evoluzione e filogenesi
 Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre II 4 Evoluzione e filogenesi FILOGENETICA CS Definzione Studio delle relazioni evolutive tra
Docente: Matteo Re UNIVERSITÀ DEGLI STUDI DI MILANO C.d.l. Informatica Bioinformatica A.A. 2013-2014 semestre II 4 Evoluzione e filogenesi FILOGENETICA CS Definzione Studio delle relazioni evolutive tra
Le macromolecole dei tessuti - 1
 Le macromolecole dei tessuti - 1 Che cosa sono le proteine? Sono macromolecole complesse ad alta informazione Sono costituite da una o più catene polipeptidiche Ogni catena peptidica è composta da centinaia
Le macromolecole dei tessuti - 1 Che cosa sono le proteine? Sono macromolecole complesse ad alta informazione Sono costituite da una o più catene polipeptidiche Ogni catena peptidica è composta da centinaia
