Tecniche per il sequenziamento degli acidi nucleici
|
|
|
- Rosalinda Fusco
- 8 anni fa
- Visualizzazioni
Transcript
1 Tecniche per il sequenziamento degli acidi nucleici
2 Nel 1965 viene determinata la prima sequenza completa di un Acido Nucleico: il trna dell alanina di lievito (78 nucleotidi) 1971: prima mappa di restrizione (individuazione della disposizione dei geni sul genoma mediante enzimi di restrizione) del DNA circolare di SV40 (11 frammenti di restrizione). Nel 1975 viene sequenziato il primo genoma completo: l RNA del fago MS2 (3568 nucleotidi). 3 geni separati da pochi nucleotidi Alle estremità 2 lunghe (129 e 174 basi) regioni non tradotte
3 Il Progetto Genoma (inizio fine 2003) Department of Energy and the National Institutes of Health Scopi del Progetto determinare le sequenze delle circa 3x10 9 coppie di basi di cui é costituito il DNA umano identificare tutti i circa ~10 5 geni geni del DNA umano registrare tutta l informazione in Banche Dati sviluppare strumenti per l'analisi dei dati raccolti trasferire le tecnologie sviluppate ai settori privati indirizzare le questioni etiche, sociali e legali che originino dallo svolgimento del progetto
4 Operazioni preliminari al sequenziamento Molecola troppo grande taglio in punti definiti DNasi completamente aspecifiche impossibile tagliare in punti specifici RNasi specificità strutturale Enzimi di restrizione
5 La scoperta degli enzimi di restrizione DNA E.coli di ceppo B in E.coli di ceppo C digestione 1966: si osserva la sopravvivenza di un DNA virale in E.coli enzima di modificazione basi metilate enzima di restrizione
6 sono stati identificati più di 150 siti di restrizione specifici sequenze specifiche di 4-8 basi casuale 1/256 1/ GTPy PuAC 3 3 CAPu PyTG 5 I frammenti di DNA tagliati dagli REs (restriction enzymes) possono essere separati, in base alle loro dimensioni relative, utilizzando l lettroforesi su gel di agaroso
7 I plasmidi sono utilizzati come vettori di clonaggio un frammento di DNA, proveniente da altra sorgente, é integrato nel plasmide (plasmide chimera)
8 Costruzione e clonazione di molecole di DNA ricombinante I passi sono: Costruzione di una molecola ricombinante (plasmide)
9
10 taglio al centro del sito di riconoscimento frammenti ad estremità piatte taglio sfalsato frammenti ad estremità coesive CCGC GG GG CGCC CCGC CGCC DNA ricombinante
11 Costruzione e clonazione di molecole di DNA ricombinante I passi sono: Costruzione di una molecola ricombinante (plasmide) Introduzione nelle cellule ospite (batteri mutanti che non degradino rapidamente il DNA estraneo) Selezione e clonazione delle cellule che portano il DNA ricombinante
12 Costruzione e clonazione di molecole di DNA ricombinante I passi sono: Costruzione di una molecola ricombinante (plasmide) Introduzione nelle cellule ospite (batteri mutanti che non degradino rapidamente il DNA estraneo) Selezione e clonazione delle cellule che portano il DNA ricombinante
13 moltiplicazione genica clonaggio molecolare DNA ricombinante utilizzando plasmidi e fagi come vettori Molti batteri contengono uno o piú plasmidi v Molecole di DNA circolare a doppia-elica v # coppie di basi (bp): da 3000 a v unitá autonoma di replicazione v opzionale per la cellula ospite (resistenza agli antibiotici)
14 Plasmidi come vettori di clonaggio un frammento di DNA, proveniente da altra sorgente, é integrato nel plasmide (plasmide chimera) plasmide modificato é inserito in una cellula di E.coli le cellule con il plasmide chimera sono individuate dalla loro resistenza ad un farmaco l insieme dei discendenti di queste cellule é un clone I plasmidi sono utilizzati come vettori d espressione esprimendo il gene inserito si può produrre una proteina di interesse medico (es: insulina)
15 Fotografie al microscopio elettronico Il plasmide psc101: il primo usato per clonare DNA Il fago filamentoso M13
16 Fago lambda come vettore di clonaggio
17
18 Nota bene: Amplificazione di DNA mediante PCR (Polymerase Chain Reaction) Si può amplificare una sequenza di DNA di qualsiasi origine (virus, batteri, organismi superiori) centinaia di milioni di volte in un ora (con la tecnica del DNA ricombinante ci sarebbero voluti molti giorni).
19 Polymerase Chain Reaction, PCR K.Mullis º Ciclo 2º Ciclo A partire dal 3º ciclo, ad ogni ciclo il numero di molecole di DNA a doppia elica raddoppia. All n-esimo ciclo ci saranno quindi N=2 n-2 molecole di DNA a doppia elica 3º Ciclo
20 Metodo di Maxam e Gilbert (o metodo del taglio chimico) marcatura del DNA ad una estremità con 32 P taglio a livello di uno dei 4 nucleotidi elettroforesi autoradiografia polinucleotide chinasi inserisce 32 P al terminale 5
21 Esempio Sequenza marcata 5-32 P- GCTACGTA-3 Frammenti radioattivi Taglio a livello di A: 32 P- GCT 32 P- GCTACGT Taglio a livello di G: 32 P- GCTAC Taglio a livello di C: 32 P- G 32 P- GCTA Taglio a livello di T: 32 P- GC 32 P- GCTACG
22 Diagramma schematico di un autoradiografia su gel dei frammenti radioattivi prodotti dai tagli specifici A G C T
23 Autoradiografia di gel che mostra frammenti marcati prodotti dal taglio chimico
24 Metodo di Sanger (o dell interruzione controllata) copiatura della sequenza a singolo filamento blocco della crescita ad opera di una miscela di incubazione contenente i 4 nucleosidi 3P marcati un analogo 2,3 -dideossi di uno di essi P-P-POCH 2 elettroforesi DNA polimerasi I e primer autoradiografia H H H O base H H 2 3 H
25 Variante del metodo di Sanger marcatura dei primer di ciascuna delle 4 miscele con sonde fluorescenti a λ diversi spettri di fluorescenza elettroforesi blu verde giallo viola
26 Batteri: ospiti ideali per l amplificazione di molecole di DNA produzione di numerose proteine procariotiche ed eucariotiche Molti geni eucariotici sono espressi correttamente solo in cellule eucariotiche L introduzione del DNA ricombinante in cellule eucariotiche permette di studiare, per esempio, in che modo i geni vengono attivati e disattivati durante lo sviluppo dell embrione (differenziazione)
27 DNA-computing
28 La macchina di Turing (Alan Turing, 1936)* Un meccanismo (finite control) si muove tra una coppia di nastri:. legge le istruzioni da un nastro (input tape). scrive il risultato sull altro nastro (output tape) La replicazione del DNA La DNA polimerasi scorre lungo un filamento del DNA. legge ciascuna base sul filamento. scrive il suo complemento sul filamento nuovo Leonard M. Adleman (1994): macchina di Turing DNA? *dieci anni prima della comparsa dei computers!! 1944: electronic discrete variable automatic computer (EDVAC)
29 La Tesi di Church &Turing (Alonzo Church & Alan Turing) Universalità : la macchina di Turing (= qualsiasi computer) può calcolare qualsiasi cosa sia calcolabile, nell ipotesi che ci sia sufficiente tempo e sufficiente memoria L algoritmo con cui viene fatto il calcolo deve soddisfare i seguenti requisiti 1. L algoritmo deve consistere di un set finito di istruzioni descritte con un numero finito di simboli 2. L algoritmo produce il risultato in un numero finito di steps 3. L algoritmo può, in teoria, essere operato da un umano con solo carta e penna 4. La sua esecuzione non richiede un intelligenza umana tranne quella necessaria a capire le istruzioni ed eseguirle
30 DNA Computing La sfida computazionele risolvere gli NP-problems P-problems possono essere risolti in un tempo polinomiale (N k ) NP-problems Non-standard Polynomial problems il tempo per testare una ipotesi (ansatz) è polinomiale, ma il numero di possibili prove è esponenziale (e N ) se disponessi di un computer parallelo con un numero esponenzialmente grande di nodi il tempo di calcolo sarebbe polinomiale Ovviamente P-problems NP-problems* il problema di decidere se l inclusione è stretta è uno dei problemi più importanti della matematica
31 Esempi di problemi NP (completi) Il problema del commesso viaggiatore (Hamilton path problem) Colorare un grafo connesso con 3 colori, senza vertici adiacenti dello stesso colore Problemi di consistenza (SAT-problems = SATisfability-problems) Minimizzazione di una hamiltoniana di spin completo se si trova un algoritmo (efficiente) per risolvere un problema NP completo questo potrà essere utilizzato per risolvere ogni altro problema NP
32
33 DNA computer permette di risolvere problemi NP in un tempo polinomiale strumenti molecolari naturali & artificiali appaiamento alla Watson-Crick DNA-polimerasi (+ primer) DNA-ligasi endonucleasi di restrizione sintesi (artificiale) di DNA elettroforesi su gel
34 Hamilton Path Problem Connessioni orientate Vertici Grafo Esempio di algoritmo (non efficiente) Dato un grafo con n vertici 1. generare un insieme di cammini casuali 2. per ciascun cammino a. verificare che cominci dal vertice di partenza e finisca nel vertice di arrivo; altrimenti rimuoverlo b. verificare che il cammino passi esattamente per n vertici; altrimenti rimuoverlo c. per ogni vertice verificare che il cammino passi per quel vertice; altrimenti rimuoverlo 3. Se l insieme non è vuoto: c è un cammino di Hamilton. Se l insieme è vuoto: no
35 Hamilton path: Roma-Parigi-Madrid-Berlino Madrid VOLO Berlino Roma Parigi 4 vertici (= città) 6 connessioni (= voli) Roma = vertice di partenza Berlino = vertice di arrivo CITTA SEQ. DNA COMPL ROMA ACTT GCAG TGAA CGTC PARIGI TCGG ACTG AGCC TGAC MADRID GGCT ATGT CCGA TACA BERLINO CCGA GCAA GGCT CGTT SEQUENZA # VOLO ROMA-PARIGI ROMA-BERLINO PARIGI-MADRID PARIGI-BERLINO PARIGI-ROMA MADRID-BERLINO GCAGTCGG GCAGCCGA ACTGGGCT ACTGCCGA ACTGACTT ATGTCCGA
36 Come si procede in pratica: 1. Sintesi* delle sequenze complementari ai nomi delle città e delle sequenze con i numeri dei voli (i nomi delle città non sono necessari) 2. Nella stessa provetta: un pizzico (circa molecole) di ognuna delle diverse sequenze, acqua, ligasi, sali, etc. In circa 1 sec si ha in mano il risultato * Occorrono pochi giorni per ricevere una provetta con circa molecole di DNA tutte (o in gran parte) aventi la sequenza richiesta. Una sequenza di 20 basi costa circa 25$. Si possono facilmente ottenere sequenze lunghe anche 100 basi.
37 Algoritmo: passo 1 Cosa succede nella provetta a. il volo Roma-Parigi (GCAGTCGG) ed il complemento del nome Parigi (AGCC TGAC) si incontrano per caso. b. la fine della 1 a e l inizio della 2 a sono complementari e si appaiano c. questo complesso incontra il volo Parigi-Madrid (ACTGGGCT) d. la 1 a parte di questo è complementare all ultima del complesso e si appaia e. e così via GCAGTCGG AGCCTGAC GCAGTCGGACTGGGCT AGCCTGAC... La provetta conterrà sequenze relative a cammini casuali.
38 Numero elevatissimo di cammini (sequenze) se esiste, almeno uno sarà quello di Hamilton tutti i cammini sono stati generati contemporaneamente (elevato parallelismo) ci sono (anche) circa molecole che traducono cammini che non sono quello di Hamilton devono essere eliminati
39 Algoritmo: passo 2a Eliminare le molecole che non cominciano con la città di partenza e non finiscono con la città di arrivo. Polymerase Chain Reaction (PCR) con molte copie delle sequenze. complemento del nome della città di partenza (TGAA). del cognome della città di arrivo (GCAA) [primers*] 5 ACTT 3 3 TGAA 5 si producono molte copie solo dei complementi di tutte quelle molecole la cui sequenza comincia con il nome della città di partenza * segnalano alla DNA polimerasi dove cominciare la replicazione alla Watson e Crick
40 5 CGTT TGAA 3 3 GCAA ACTT 5 Roma: ACTT GCAG Berlino: CCGA GCAA si producono molte copie solo dei complementi (prodotti nel precedente passo e che quindi cominciano tutti con il complemento del nome della città di partenza) di tutte quelle molecole la cui sequenza comincia con il complemento del cognome della città di arrivo le molecole moltiplicate da PCR sono quindi quelle che cominciano con il nome della città di partenza e finiscono con il cognome della città di arrivo
41 Algoritmo: passo 2b Con l elettroforesi seleziono solo quelle molecole che hanno la lunghezza (numero di vertici) giusta. Nell esempio: 24 basi Algoritmo: passo 2c i. Si attacca al complemento del nome di una delle città intermedie una microscopica pallina di ferro. ii. Si mette in soluzione e si promuove l appaiamento. iii. Si usa un magnete per attrarre solo le molecole attaccate alla pallina e le altre vengono lavate via. iv. Si stacca la pallina di ferro v. Si ripete la stessa operazione a partire da (i) per le altre città intermedie Algoritmo: passo 3 PCR seguito da elettroforesi e sequenziamento Nota: se la provetta è vuota: non ci sono cammini di Hamilton
42 Passo 2c
43 Vantaggi dei computers a DNA Capacità di Memoria: Parallelismo: 1gr DNA (1cc) = 10 9 CD numeri di volo connessi in 1 sec Efficienza energetica: DNA: 1 joule/10 19 operazioni di legame supercomputers: 1 joule/10 9 operazioni...però, per conoscere il risultato, ottenuto in 1 sec, ci è voluta circa 1 settimana!...e soprattutto, il numero di catene di DNA che devono essere prodotte a caso cresce esponenzialmente con il numero di variabili (nodi) i problemi NP non hanno veramente trovato una soluzione!
44 Perchè il computer a DNA non ha rimpiazzato quello al silicio? DNA = macchina di Turing? Abbastanza più complicato Vantaggi del DNA: parallelismo massivo, numero esponenzialmente grande di processori (più DNA nel beaker!!). Vantaggi del silicone: versatilità, disponibilità, durata, facilità d interfacciamento anni di sviluppo di algoritmi e tecnologie per il silicio non facilmente trasferibili al computer a DNA
La macchina di Turing (Alan Turing, 1936)*
 DNA-computing La macchina di Turing (Alan Turing, 1936)* Un meccanismo (finite control) si muove tra una coppia di nastri:. legge le istruzioni da un nastro (input tape). scrive il risultato sull altro
DNA-computing La macchina di Turing (Alan Turing, 1936)* Un meccanismo (finite control) si muove tra una coppia di nastri:. legge le istruzioni da un nastro (input tape). scrive il risultato sull altro
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Le molecole di RNA possono assumere particolari strutture che conferiscono loro particolari funzioni.
 Traduzione: t-rna Le molecole di RNA possono assumere particolari strutture che conferiscono loro particolari funzioni. Singola catena alta flessibilità ripiegamento su stessa legami deboli con se stessa
Traduzione: t-rna Le molecole di RNA possono assumere particolari strutture che conferiscono loro particolari funzioni. Singola catena alta flessibilità ripiegamento su stessa legami deboli con se stessa
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
Il metabolismo dell RNA. Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
 Il metabolismo dell RNA I vari tipi di RNA Il filamento di DNA che dirige la sintesi dello mrna è chiamato filamento stampo o filamento antisenso. L altro filamento che ha sequenza identica a quella dello
Il metabolismo dell RNA I vari tipi di RNA Il filamento di DNA che dirige la sintesi dello mrna è chiamato filamento stampo o filamento antisenso. L altro filamento che ha sequenza identica a quella dello
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
DNA - RNA. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi.
 DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Esperienza 3: clonaggio di un gene in un plasmide
 Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
Biotecnologie ed OGM. Prima parte: DNA ricombinante e microorganismi geneticamente modificati.
 Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
LE MOLECOLE INFORMAZIONALI. Lezioni d'autore Treccani
 LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
Il genoma dinamico: gli elementi trasponibili
 Il genoma dinamico: gli elementi trasponibili Anni trenta: studi sul mais ribaltano la visione classica secondo cui i geni si trovano solo in loci fissi sul cromosoma principale Esistono elementi genetici
Il genoma dinamico: gli elementi trasponibili Anni trenta: studi sul mais ribaltano la visione classica secondo cui i geni si trovano solo in loci fissi sul cromosoma principale Esistono elementi genetici
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Introduzione. Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD... 6
 Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
RNA polimerasi operone. L operatore è il tratto
 La regolazione genica nei procarioti Alcune proteine vengono prodotte dalla cellula ad un ritmo relativamente costante e l attività dei geni che codificano queste proteine non è regolata in modo sofisticato.
La regolazione genica nei procarioti Alcune proteine vengono prodotte dalla cellula ad un ritmo relativamente costante e l attività dei geni che codificano queste proteine non è regolata in modo sofisticato.
COME VIENE REALIZZATA UNA RICERCA SPERIMENTALE IN BIOLOGIA MOLECOLARE?
 COME VIENE REALIZZATA UNA RICERCA SPERIMENTALE IN BIOLOGIA MOLECOLARE? A Flusso di attività B - INPUT C Descrizione dell attività D RISULTATO E - SISTEMA PROFESSIONALE 0. RICHIESTA DI STUDIARE E/O INDIVIDUARE
COME VIENE REALIZZATA UNA RICERCA SPERIMENTALE IN BIOLOGIA MOLECOLARE? A Flusso di attività B - INPUT C Descrizione dell attività D RISULTATO E - SISTEMA PROFESSIONALE 0. RICHIESTA DI STUDIARE E/O INDIVIDUARE
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Attività 9. La città fangosa Minimal Spanning Trees
 Attività 9 La città fangosa Minimal Spanning Trees Sommario la nostra società ha molti collegamenti in rete: la rete telefonica, la rete energetica, la rete stradale. Per una rete in particolare, ci sono
Attività 9 La città fangosa Minimal Spanning Trees Sommario la nostra società ha molti collegamenti in rete: la rete telefonica, la rete energetica, la rete stradale. Per una rete in particolare, ci sono
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
REPLICAZIONE DEL DNA
 REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
Sommario. Definizione di informatica. Definizione di un calcolatore come esecutore. Gli algoritmi.
 Algoritmi 1 Sommario Definizione di informatica. Definizione di un calcolatore come esecutore. Gli algoritmi. 2 Informatica Nome Informatica=informazione+automatica. Definizione Scienza che si occupa dell
Algoritmi 1 Sommario Definizione di informatica. Definizione di un calcolatore come esecutore. Gli algoritmi. 2 Informatica Nome Informatica=informazione+automatica. Definizione Scienza che si occupa dell
LA GENETICA: DNA e RNA LA GENETICA. DNA e RNA. Prof. Daniele Verri
 LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
LA GENETICA DNA e RNA Prof. Daniele Verri L'acido desossiribonucleico o deossiribonucleico (DNA) è un acido nucleico che contiene le informazioni necessarie per la formazione di RNA e proteine. LA GENETICA:
LA MATERIA Suggerimenti didattici e schede
 LA MATERIA Suggerimenti didattici e schede Iniziamo il percorso chiedendo a un bambino di consegnarci alcune cose: una gomma, una penna, un capello. Domandiamo a un altro di consegnarci una gioia, una
LA MATERIA Suggerimenti didattici e schede Iniziamo il percorso chiedendo a un bambino di consegnarci alcune cose: una gomma, una penna, un capello. Domandiamo a un altro di consegnarci una gioia, una
Definizione di genoteca (o library) di DNA
 Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
Biosintesi non ribosomiale di metaboliti peptidici bioattivi
 Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
3 CENNI DI TEORIA DELLA COMPLESSITA COMPUTAZIONALE. E. Amaldi Fondamenti di R.O. Politecnico di Milano 1
 3 CENNI DI TEORIA DELLA COMPLESSITA COMPUTAZIONALE E. Amaldi Fondamenti di R.O. Politecnico di Milano 1 Scopo: Stimare l onere computazionale per risolvere problemi di ottimizzazione e di altra natura
3 CENNI DI TEORIA DELLA COMPLESSITA COMPUTAZIONALE E. Amaldi Fondamenti di R.O. Politecnico di Milano 1 Scopo: Stimare l onere computazionale per risolvere problemi di ottimizzazione e di altra natura
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
Prof.ssa Gamba Sabrina. Lezione 7: IL DNA. Duplicazione e sintesi delle proteine
 Prof.ssa Gamba Sabrina Lezione 7: IL DNA Duplicazione e sintesi delle proteine concetti chiave della lezione Costituzione fisico-chimica del DNA Basi azotate Duplicazione Concetto di geni Rna Trascrizione
Prof.ssa Gamba Sabrina Lezione 7: IL DNA Duplicazione e sintesi delle proteine concetti chiave della lezione Costituzione fisico-chimica del DNA Basi azotate Duplicazione Concetto di geni Rna Trascrizione
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Dal DNA all RNA. La trascrizione nei procarioti e negli eucarioti
 Dal DNA all RNA La trascrizione nei procarioti e negli eucarioti DOGMA CENTRALE DELLA BIOLOGIA MOLECOLARE Gene Regione di DNA che porta l informazione (= che CODIFICA) per una catena polipeptidica o per
Dal DNA all RNA La trascrizione nei procarioti e negli eucarioti DOGMA CENTRALE DELLA BIOLOGIA MOLECOLARE Gene Regione di DNA che porta l informazione (= che CODIFICA) per una catena polipeptidica o per
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Come funzionano gli oligo Antisenso? RNA WORLD. mrna. Regolare l espressione genica tramite molecole di RNA. Come funzionano gli oligo antisenso?
 RNA WORLD RNA Come funzionano gli oligo Antisenso? mrna Non coding RNA AAAAAAA rrna trna snrna snorna RNA Antisenso sirna Arresto della traduzione Proteina incompleta o nessuna sintesi MECCANISMO PASSIVO
RNA WORLD RNA Come funzionano gli oligo Antisenso? mrna Non coding RNA AAAAAAA rrna trna snrna snorna RNA Antisenso sirna Arresto della traduzione Proteina incompleta o nessuna sintesi MECCANISMO PASSIVO
Sequenziamento a minimo costo di commutazione in macchine o celle con costo lineare e posizione home (In generale il metodo di ottimizzazione
 Sequenziamento a minimo costo di commutazione in macchine o celle con costo lineare e posizione home (In generale il metodo di ottimizzazione presentato in questo file trova la seq. a costo minimo per
Sequenziamento a minimo costo di commutazione in macchine o celle con costo lineare e posizione home (In generale il metodo di ottimizzazione presentato in questo file trova la seq. a costo minimo per
8-06-2010. BLOTTING degli acidi nucleici: RT-PCR SOUTHERN BLOTTING NORTHERN BLOTTING. BLOTTING delle proteine: NORTHERN BLOTTING SOUTHERN BLOTTING
 8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
L informatica INTRODUZIONE. L informatica. Tassonomia: criteri. È la disciplina scientifica che studia
 L informatica È la disciplina scientifica che studia INTRODUZIONE I calcolatori, nati in risposta all esigenza di eseguire meccanicamente operazioni ripetitive Gli algoritmi, nati in risposta all esigenza
L informatica È la disciplina scientifica che studia INTRODUZIONE I calcolatori, nati in risposta all esigenza di eseguire meccanicamente operazioni ripetitive Gli algoritmi, nati in risposta all esigenza
Le parole dell informatica: modello di calcolo, complessità e trattabilità
 Le parole dell informatica: modello di calcolo, complessità e trattabilità Angelo Montanari Dipartimento di Matematica e Informatica Università degli Studi di Udine Ciclo di seminari su un Vocabolario
Le parole dell informatica: modello di calcolo, complessità e trattabilità Angelo Montanari Dipartimento di Matematica e Informatica Università degli Studi di Udine Ciclo di seminari su un Vocabolario
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas
 1 Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas Capitolo 7 Enzimi, le macchine della vita Piccole macchine regolano la funzione del corpo umano in un orchestrazione perfetta e a velocità
1 Tratto dal libro Come vivere 150 anni Dr. Dimitris Tsoukalas Capitolo 7 Enzimi, le macchine della vita Piccole macchine regolano la funzione del corpo umano in un orchestrazione perfetta e a velocità
Appunti sulla Macchina di Turing. Macchina di Turing
 Macchina di Turing Una macchina di Turing è costituita dai seguenti elementi (vedi fig. 1): a) una unità di memoria, detta memoria esterna, consistente in un nastro illimitato in entrambi i sensi e suddiviso
Macchina di Turing Una macchina di Turing è costituita dai seguenti elementi (vedi fig. 1): a) una unità di memoria, detta memoria esterna, consistente in un nastro illimitato in entrambi i sensi e suddiviso
Funzioni in C. Violetta Lonati
 Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Calcolatori Elettronici A a.a. 2008/2009
 Calcolatori Elettronici A a.a. 2008/2009 PRESTAZIONI DEL CALCOLATORE Massimiliano Giacomin Due dimensioni Tempo di risposta (o tempo di esecuzione): il tempo totale impiegato per eseguire un task (include
Calcolatori Elettronici A a.a. 2008/2009 PRESTAZIONI DEL CALCOLATORE Massimiliano Giacomin Due dimensioni Tempo di risposta (o tempo di esecuzione): il tempo totale impiegato per eseguire un task (include
Risposta: 2. Uracile. Risposta: 2. legami idrogeno. Adenina, Citosina e Guanina si trovano sia nell RNA che nel DNA.
 Risposta: 2. Uracile Adenina, Citosina e Guanina si trovano sia nell RNA che nel DNA. La Timina si trova soltanto nel DNA; l Uracile si sostituisce alla Timina nelle molecole dell RNA. Risposta: 2. legami
Risposta: 2. Uracile Adenina, Citosina e Guanina si trovano sia nell RNA che nel DNA. La Timina si trova soltanto nel DNA; l Uracile si sostituisce alla Timina nelle molecole dell RNA. Risposta: 2. legami
Corso di Informatica
 Corso di Informatica Modulo T3 1-Sottoprogrammi 1 Prerequisiti Tecnica top-down Programmazione elementare 2 1 Introduzione Lo scopo di questa Unità è utilizzare la metodologia di progettazione top-down
Corso di Informatica Modulo T3 1-Sottoprogrammi 1 Prerequisiti Tecnica top-down Programmazione elementare 2 1 Introduzione Lo scopo di questa Unità è utilizzare la metodologia di progettazione top-down
ARCHIVI E DATABASE (prof. Ivaldi Giuliano)
 ARCHIVI E DATABASE (prof. Ivaldi Giuliano) Archivio: è un insieme di registrazioni (o records) ciascuna delle quali è costituita da un insieme prefissato di informazioni elementari dette attributi (o campi).
ARCHIVI E DATABASE (prof. Ivaldi Giuliano) Archivio: è un insieme di registrazioni (o records) ciascuna delle quali è costituita da un insieme prefissato di informazioni elementari dette attributi (o campi).
Metodi Stocastici per la Finanza
 Metodi Stocastici per la Finanza Tiziano Vargiolu vargiolu@math.unipd.it 1 1 Università degli Studi di Padova Anno Accademico 2011-2012 Lezione 6 Indice 1 Il metodo bootstrap 2 Esercitazione 3 Interpolazione
Metodi Stocastici per la Finanza Tiziano Vargiolu vargiolu@math.unipd.it 1 1 Università degli Studi di Padova Anno Accademico 2011-2012 Lezione 6 Indice 1 Il metodo bootstrap 2 Esercitazione 3 Interpolazione
Esempi di algoritmi. Lezione III
 Esempi di algoritmi Lezione III Scopo della lezione Implementare da zero algoritmi di media complessità. Verificare la correttezza di un algoritmo eseguendolo a mano. Imparare a valutare le prestazioni
Esempi di algoritmi Lezione III Scopo della lezione Implementare da zero algoritmi di media complessità. Verificare la correttezza di un algoritmo eseguendolo a mano. Imparare a valutare le prestazioni
Studente: SANTORO MC. Matricola : 528
 CORSO di LAUREA in INFORMATICA Corso di CALCOLO NUMERICO a.a. 2004-05 Studente: SANTORO MC. Matricola : 528 PROGETTO PER L ESAME 1. Sviluppare una versione dell algoritmo di Gauss per sistemi con matrice
CORSO di LAUREA in INFORMATICA Corso di CALCOLO NUMERICO a.a. 2004-05 Studente: SANTORO MC. Matricola : 528 PROGETTO PER L ESAME 1. Sviluppare una versione dell algoritmo di Gauss per sistemi con matrice
PLASMIDI puc ALTRI TIPI DI VETTORI. VETTORI λ 17-06-2010
 PLASMIDI puc ALTRI TIPI DI VETTORI VETTORI λ (15-20 Kb) = vettori ottenuti apportando delle modifiche al genoma del batteriofago λ. COSMIDI (40-45 Kb) = plasmidi che contengono i siti cos di λ utili per
PLASMIDI puc ALTRI TIPI DI VETTORI VETTORI λ (15-20 Kb) = vettori ottenuti apportando delle modifiche al genoma del batteriofago λ. COSMIDI (40-45 Kb) = plasmidi che contengono i siti cos di λ utili per
Banca dati Professioniste in rete per le P.A. Guida all uso per le Professioniste
 Banca dati Professioniste in rete per le P.A. Guida all uso per le Professioniste versione 2.1 24/09/2015 aggiornamenti: 23-set-2015; 24-set-2015 Autore: Francesco Brunetta (http://www.francescobrunetta.it/)
Banca dati Professioniste in rete per le P.A. Guida all uso per le Professioniste versione 2.1 24/09/2015 aggiornamenti: 23-set-2015; 24-set-2015 Autore: Francesco Brunetta (http://www.francescobrunetta.it/)
introduzione I MICROCONTROLLORI
 introduzione I MICROCONTROLLORI Definizione Un microcontrollore è un dispositivo elettronico programmabile Può svolgere autonomamente diverse funzioni in base al programma in esso implementato Non è la
introduzione I MICROCONTROLLORI Definizione Un microcontrollore è un dispositivo elettronico programmabile Può svolgere autonomamente diverse funzioni in base al programma in esso implementato Non è la
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE
 LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
Soluzione dell esercizio del 2 Febbraio 2004
 Soluzione dell esercizio del 2 Febbraio 2004 1. Casi d uso I casi d uso sono riportati in Figura 1. Figura 1: Diagramma dei casi d uso. E evidenziato un sotto caso di uso. 2. Modello concettuale Osserviamo
Soluzione dell esercizio del 2 Febbraio 2004 1. Casi d uso I casi d uso sono riportati in Figura 1. Figura 1: Diagramma dei casi d uso. E evidenziato un sotto caso di uso. 2. Modello concettuale Osserviamo
www.fisiokinesiterapia.biz TRASCRIZIONE
 www.fisiokinesiterapia.biz TRASCRIZIONE TRASCRIZIONE Processo mediante il quale una sequenza di DNA (un gene) viene copiata in una sequenza di RNA Dalla trascrizione derivano gli mrna, che verranno tradotti
www.fisiokinesiterapia.biz TRASCRIZIONE TRASCRIZIONE Processo mediante il quale una sequenza di DNA (un gene) viene copiata in una sequenza di RNA Dalla trascrizione derivano gli mrna, che verranno tradotti
Macromolecole Biologiche. I domini (III)
 I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
Software di sistema e software applicativo. I programmi che fanno funzionare il computer e quelli che gli permettono di svolgere attività specifiche
 Software di sistema e software applicativo I programmi che fanno funzionare il computer e quelli che gli permettono di svolgere attività specifiche Software soft ware soffice componente è la parte logica
Software di sistema e software applicativo I programmi che fanno funzionare il computer e quelli che gli permettono di svolgere attività specifiche Software soft ware soffice componente è la parte logica
ALGEBRA DELLE PROPOSIZIONI
 Università di Salerno Fondamenti di Informatica Corso di Laurea Ingegneria Corso B Docente: Ing. Giovanni Secondulfo Anno Accademico 2010-2011 ALGEBRA DELLE PROPOSIZIONI Fondamenti di Informatica Algebra
Università di Salerno Fondamenti di Informatica Corso di Laurea Ingegneria Corso B Docente: Ing. Giovanni Secondulfo Anno Accademico 2010-2011 ALGEBRA DELLE PROPOSIZIONI Fondamenti di Informatica Algebra
Progetto della classe II C
 Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
DOMANDA FREQUENTE: QUALE E LA FUNZIONE DI UNA CERTA PROTEINA? SI AUMENTA O SI DIMINUISCE L ESPRESSIONE DELLA PROTEINA
 DOMANDA FREQUENTE: QUALE E LA FUNZIONE DI UNA CERTA PROTEINA? OVERESPRESSIONE DELLA PROTEINA ESPRESSIONE ECTOPICA CON UN VETTORE DI ESPRESSIONE ABOLIZIONE DELLA ESPRESSIONE DELLA PROTEINA INTERFERENZA
DOMANDA FREQUENTE: QUALE E LA FUNZIONE DI UNA CERTA PROTEINA? OVERESPRESSIONE DELLA PROTEINA ESPRESSIONE ECTOPICA CON UN VETTORE DI ESPRESSIONE ABOLIZIONE DELLA ESPRESSIONE DELLA PROTEINA INTERFERENZA
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
De-constructing and Reconstructing. Life. (smontare e costruire oggetti biologici)
 De-constructing and Reconstructing Life (smontare e costruire oggetti biologici) ogni oggetto biologico rappresenta un particolare stato di aggregazione e di organizzazione della materia, corrispondente
De-constructing and Reconstructing Life (smontare e costruire oggetti biologici) ogni oggetto biologico rappresenta un particolare stato di aggregazione e di organizzazione della materia, corrispondente
Maria Antonietta Lepore. Principali tecniche di biologia molecolare clinica
 Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Scopo della lezione. Informatica. Informatica - def. 1. Informatica
 Scopo della lezione Informatica per le lauree triennali LEZIONE 1 - Che cos è l informatica Introdurre i concetti base della materia Definire le differenze tra hardware e software Individuare le applicazioni
Scopo della lezione Informatica per le lauree triennali LEZIONE 1 - Che cos è l informatica Introdurre i concetti base della materia Definire le differenze tra hardware e software Individuare le applicazioni
Vettori di espressione
 Vettori di espressione Vengono usati per: 1.Generare sonde di RNA 2.Produrre la proteina codificata Per fare questo viene utilizzato un promotore che risiede sul vettore, modificato per ottimizzare l interazione
Vettori di espressione Vengono usati per: 1.Generare sonde di RNA 2.Produrre la proteina codificata Per fare questo viene utilizzato un promotore che risiede sul vettore, modificato per ottimizzare l interazione
SUPERAVVOLGIMENTO DEL DNA (ORGANIZZAZIONE TERZIARIA DEL DNA)
 SUPERAVVOLGIMENTO DEL DNA (ORGANIZZAZIONE TERZIARIA DEL DNA) ORGANIZZAZIONE TERZIARIA DEL DNA Il DNA cellulare contiene porzioni geniche e intergeniche, entrambe necessarie per le funzioni vitali della
SUPERAVVOLGIMENTO DEL DNA (ORGANIZZAZIONE TERZIARIA DEL DNA) ORGANIZZAZIONE TERZIARIA DEL DNA Il DNA cellulare contiene porzioni geniche e intergeniche, entrambe necessarie per le funzioni vitali della
PROVA INTRACORSO TRACCIA A Pagina 1 di 6
 PROVA INTRACORSO DI ELEMENTI DI INFORMATICA MATRICOLA COGNOME E NOME TRACCIA A DOMANDA 1 Calcolare il risultato delle seguenti operazioni binarie tra numeri interi con segno rappresentati in complemento
PROVA INTRACORSO DI ELEMENTI DI INFORMATICA MATRICOLA COGNOME E NOME TRACCIA A DOMANDA 1 Calcolare il risultato delle seguenti operazioni binarie tra numeri interi con segno rappresentati in complemento
Il flusso dell informazione genetica. DNA -->RNA-->Proteine
 Il flusso dell informazione genetica DNA -->RNA-->Proteine Abbiamo visto i principali esperimenti che hanno dimostrato che il DNA è la molecola depositaria dell informazione genetica nella maggior parte
Il flusso dell informazione genetica DNA -->RNA-->Proteine Abbiamo visto i principali esperimenti che hanno dimostrato che il DNA è la molecola depositaria dell informazione genetica nella maggior parte
METODI DI MARCATURA DEGLI ACIDI NUCLEICI
 METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
Note del Corso di Modelli Biologici Discreti: Un paio di algoritmi DNA per risolvere SAT
 Note del Corso di Modelli Biologici Discreti: Un paio di algoritmi DNA per risolvere SAT Giuditta Franco Corso di Laurea in Bioinformatica - AA 2012/2013 Uno dei più grossi risultati nell informatica degli
Note del Corso di Modelli Biologici Discreti: Un paio di algoritmi DNA per risolvere SAT Giuditta Franco Corso di Laurea in Bioinformatica - AA 2012/2013 Uno dei più grossi risultati nell informatica degli
TECNICHE DI SIMULAZIONE
 TECNICHE DI SIMULAZIONE INTRODUZIONE Francesca Mazzia Dipartimento di Matematica Università di Bari a.a. 2004/2005 TECNICHE DI SIMULAZIONE p. 1 Introduzione alla simulazione Una simulazione è l imitazione
TECNICHE DI SIMULAZIONE INTRODUZIONE Francesca Mazzia Dipartimento di Matematica Università di Bari a.a. 2004/2005 TECNICHE DI SIMULAZIONE p. 1 Introduzione alla simulazione Una simulazione è l imitazione
A intervalli regolari ogni router manda la sua tabella a tutti i vicini, e riceve quelle dei vicini.
 Algoritmi di routing dinamici (pag.89) UdA2_L5 Nelle moderne reti si usano algoritmi dinamici, che si adattano automaticamente ai cambiamenti della rete. Questi algoritmi non sono eseguiti solo all'avvio
Algoritmi di routing dinamici (pag.89) UdA2_L5 Nelle moderne reti si usano algoritmi dinamici, che si adattano automaticamente ai cambiamenti della rete. Questi algoritmi non sono eseguiti solo all'avvio
INTRODUZIONE AGLI ALGORITMI INTRODUZIONE AGLI ALGORITMI INTRODUZIONE AGLI ALGORITMI INTRODUZIONE AGLI ALGORITMI
 INTRODUZIONE AGLI ALGORITMI Prima di riuscire a scrivere un programma, abbiamo bisogno di conoscere un metodo risolutivo, cioè un metodo che a partire dai dati di ingresso fornisce i risultati attesi.
INTRODUZIONE AGLI ALGORITMI Prima di riuscire a scrivere un programma, abbiamo bisogno di conoscere un metodo risolutivo, cioè un metodo che a partire dai dati di ingresso fornisce i risultati attesi.
Fasi di creazione di un programma
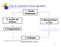 Fasi di creazione di un programma 1. Studio Preliminare 2. Analisi del Sistema 6. Manutenzione e Test 3. Progettazione 5. Implementazione 4. Sviluppo 41 Sviluppo di programmi Per la costruzione di un programma
Fasi di creazione di un programma 1. Studio Preliminare 2. Analisi del Sistema 6. Manutenzione e Test 3. Progettazione 5. Implementazione 4. Sviluppo 41 Sviluppo di programmi Per la costruzione di un programma
Corso di Informatica
 Corso di Informatica Modulo T2 3-Compilatori e interpreti 1 Prerequisiti Principi di programmazione Utilizzo di un compilatore 2 1 Introduzione Una volta progettato un algoritmo codificato in un linguaggio
Corso di Informatica Modulo T2 3-Compilatori e interpreti 1 Prerequisiti Principi di programmazione Utilizzo di un compilatore 2 1 Introduzione Una volta progettato un algoritmo codificato in un linguaggio
Architettura di un calcolatore
 2009-2010 Ingegneria Aerospaziale Prof. A. Palomba - Elementi di Informatica (E-Z) 7 Architettura di un calcolatore Lez. 7 1 Modello di Von Neumann Il termine modello di Von Neumann (o macchina di Von
2009-2010 Ingegneria Aerospaziale Prof. A. Palomba - Elementi di Informatica (E-Z) 7 Architettura di un calcolatore Lez. 7 1 Modello di Von Neumann Il termine modello di Von Neumann (o macchina di Von
Dispensa di Informatica I.1
 IL COMPUTER: CONCETTI GENERALI Il Computer (o elaboratore) è un insieme di dispositivi di diversa natura in grado di acquisire dall'esterno dati e algoritmi e produrre in uscita i risultati dell'elaborazione.
IL COMPUTER: CONCETTI GENERALI Il Computer (o elaboratore) è un insieme di dispositivi di diversa natura in grado di acquisire dall'esterno dati e algoritmi e produrre in uscita i risultati dell'elaborazione.
Dispense di Informatica per l ITG Valadier
 La notazione binaria Dispense di Informatica per l ITG Valadier Le informazioni dentro il computer All interno di un calcolatore tutte le informazioni sono memorizzate sottoforma di lunghe sequenze di
La notazione binaria Dispense di Informatica per l ITG Valadier Le informazioni dentro il computer All interno di un calcolatore tutte le informazioni sono memorizzate sottoforma di lunghe sequenze di
Excel. A cura di Luigi Labonia. e-mail: luigi.lab@libero.it
 Excel A cura di Luigi Labonia e-mail: luigi.lab@libero.it Introduzione Un foglio elettronico è un applicazione comunemente usata per bilanci, previsioni ed altri compiti tipici del campo amministrativo
Excel A cura di Luigi Labonia e-mail: luigi.lab@libero.it Introduzione Un foglio elettronico è un applicazione comunemente usata per bilanci, previsioni ed altri compiti tipici del campo amministrativo
Laboratorio di Informatica di Base Archivi e Basi di Dati
 Laboratorio di Informatica di Base Archivi e Basi di Dati Introduzione La memorizzazione dei dati è un aspetto molto importante dell informatica Oggi, mediante i computer, è possibile memorizzare e modificare
Laboratorio di Informatica di Base Archivi e Basi di Dati Introduzione La memorizzazione dei dati è un aspetto molto importante dell informatica Oggi, mediante i computer, è possibile memorizzare e modificare
4 3 4 = 4 x 10 2 + 3 x 10 1 + 4 x 10 0 aaa 10 2 10 1 10 0
 Rappresentazione dei numeri I numeri che siamo abituati ad utilizzare sono espressi utilizzando il sistema di numerazione decimale, che si chiama così perché utilizza 0 cifre (0,,2,3,4,5,6,7,8,9). Si dice
Rappresentazione dei numeri I numeri che siamo abituati ad utilizzare sono espressi utilizzando il sistema di numerazione decimale, che si chiama così perché utilizza 0 cifre (0,,2,3,4,5,6,7,8,9). Si dice
Università degli Studi di Ferrara - A.A. 2014/15 Dott. Valerio Muzzioli ORDINAMENTO DEI DATI
 ORDINAMENTO DEI DATI Quando si ordina un elenco (ovvero una serie di righe contenenti dati correlati), le righe sono ridisposte in base al contenuto di una colonna specificata. Distinguiamo due tipi di
ORDINAMENTO DEI DATI Quando si ordina un elenco (ovvero una serie di righe contenenti dati correlati), le righe sono ridisposte in base al contenuto di una colonna specificata. Distinguiamo due tipi di
Primi esercizi per gli studenti del corso di Statistica ed Elementi di Probabilita
 Primi esercizi per gli studenti del corso di Statistica ed Elementi di Probabilita NOTA 1 Gli esercizi sono presi da compiti degli scorsi appelli, oppure da testi o dispense di colleghi. A questi ultimi
Primi esercizi per gli studenti del corso di Statistica ed Elementi di Probabilita NOTA 1 Gli esercizi sono presi da compiti degli scorsi appelli, oppure da testi o dispense di colleghi. A questi ultimi
Le macchine di Turing
 Le macchine di Turing Alan Turing (1912-1954) 1954) Il problema della decisione i L Entscheidungsproblem [il problema della decisione] è risolto se si conosce una procedura che permette di decidere la
Le macchine di Turing Alan Turing (1912-1954) 1954) Il problema della decisione i L Entscheidungsproblem [il problema della decisione] è risolto se si conosce una procedura che permette di decidere la
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY.
 Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
La regolazione genica nei virus
 La regolazione genica nei virus Lic. Scientifico A. Meucci Aprilia Prof. Rolando Neri I VIRUS INDICE Caratteristiche dei virus: il capside e il genoma virale Classificazione virale Fasi del ciclo riproduttivo
La regolazione genica nei virus Lic. Scientifico A. Meucci Aprilia Prof. Rolando Neri I VIRUS INDICE Caratteristiche dei virus: il capside e il genoma virale Classificazione virale Fasi del ciclo riproduttivo
Dal DNA alle proteine: La trascrizione e la traduzione
 Dal DNA alle proteine: La trascrizione e la traduzione DNA RNA Trascrizione RNA PROTEINE Traduzione Dove avvengono? GLI EUCARIOTI I PROCARIOTI Cambell, Reece Biologia ZANICHELLI Trascrizione Sintesi di
Dal DNA alle proteine: La trascrizione e la traduzione DNA RNA Trascrizione RNA PROTEINE Traduzione Dove avvengono? GLI EUCARIOTI I PROCARIOTI Cambell, Reece Biologia ZANICHELLI Trascrizione Sintesi di
Corso di Biologia Molecolare
 Corso di Biologia Molecolare Dott.ssa Renata Tisi Dip. Biotecnologie e Bioscienze Ed. U4 Tel. 02 6448 3522 renata.tisi@unimib.it Acidi nucleici Il ruolo degli acidi nucleici è quello di custodire e trasmettere
Corso di Biologia Molecolare Dott.ssa Renata Tisi Dip. Biotecnologie e Bioscienze Ed. U4 Tel. 02 6448 3522 renata.tisi@unimib.it Acidi nucleici Il ruolo degli acidi nucleici è quello di custodire e trasmettere
Sistemi Web! per il turismo! - lezione 3 -
 Sistemi Web per il turismo - lezione 3 - I computer sono in grado di eseguire molte operazioni, e di risolvere un gran numero di problemi. E arrivato il momento di delineare esplicitamente il campo di
Sistemi Web per il turismo - lezione 3 - I computer sono in grado di eseguire molte operazioni, e di risolvere un gran numero di problemi. E arrivato il momento di delineare esplicitamente il campo di
Approccio stratificato
 Approccio stratificato Il sistema operativo è suddiviso in strati (livelli), ciascuno costruito sopra quelli inferiori. Il livello più basso (strato 0) è l hardware, il più alto (strato N) è l interfaccia
Approccio stratificato Il sistema operativo è suddiviso in strati (livelli), ciascuno costruito sopra quelli inferiori. Il livello più basso (strato 0) è l hardware, il più alto (strato N) è l interfaccia
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Perché abbiamo deciso di sequenziare il genoma umano
 L'immagine sopra rappresenta le tappe fondamentali per la scoperta del genoma umano. Una versione più interattiva della mappa è disponibile nel sito del progetto genoma umano, nella sezione dedicata alla
L'immagine sopra rappresenta le tappe fondamentali per la scoperta del genoma umano. Una versione più interattiva della mappa è disponibile nel sito del progetto genoma umano, nella sezione dedicata alla
Archivi e database. Prof. Michele Batocchi A.S. 2013/2014
 Archivi e database Prof. Michele Batocchi A.S. 2013/2014 Introduzione L esigenza di archiviare (conservare documenti, immagini, ricordi, ecc.) è un attività senza tempo che è insita nell animo umano Primi
Archivi e database Prof. Michele Batocchi A.S. 2013/2014 Introduzione L esigenza di archiviare (conservare documenti, immagini, ricordi, ecc.) è un attività senza tempo che è insita nell animo umano Primi
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
SINTESI DELL RNA. Replicazione. Trascrizione. Traduzione
 SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
