Corso di Struttura e Funzione dei Genomi (LM7) mutuato con il corso di Genomica e Trascrittomica (LM9) Genomi. Diapositive, articoli e dispense:
|
|
|
- Michele Mantovani
- 5 anni fa
- Visualizzazioni
Transcript
1 Corso di Struttura e Funzione dei Genomi (LM7) mutuato con il corso di Genomica e Trascrittomica (LM9) Genomi Diapositive, articoli e dispense:
2 Vettori di clonaggio I vettori di clonaggio sono brevemente trattati ai soli fini di un «ripasso» delle loro caratteristiche, dato che ad essi si fa riferimento in diversi argormenti trattati nell ambito del Corso Per lo studente con scarse conoscenze, informazioni dettagliate sono disponibili su: Tecnologie Biomolecolari 2010.pdf
3 Plasmidi
4 Cosmidi
5 Fagi
6
7 Fagi
8 Yeast Artificial Chromosome (YAC)
9 BAC Il vettore BAC è costituito da: geni oris e repe (Mediano le replicazione unidirezionale del fattore F) geni para e parb (Mantengono il numero di copie del fattore ad un basso livello (1 copia).un alto numero di copie potrebbe causare problemi di instabilità degli inserti per delezioni e riarrangiamenti del DNA clonato a causa di eventi di ricombinazione omologa) gene per la resistenza ad un antibiotico sito di clonaggio: È fiancheggiato dai promotori T7 e SP6 che generano sonde di DNA per il chromosome walking e per il sequenziamento d'inserti di DNA. cosn e loxp sono siti di taglio specifici per terminasi del batteriofago l e consentono la formazione di estremità che possono essere usate, nelle mappe di restrizione, per riarrangiare i cloni. I cloni ricombinanti sono selezionati mediante colorazione bianco/blu.
10 Comparazione fra YAC and BAC Cloning System Features YAC BAC Configuration Linear Circular Host Yeast Bacteria Copy Number / Cell Cloning Capacity Unlimited up-to 350 kb Transformation Spheroplast (10^7 T/ug) Electroporation (10^10 T/ug) Chimerism up to 40% None to low DNA Isolation Pulsed-field-gelelectrophoresis - Gel Isolation Insert Stability Unstable Stable Standard Plasmid Miniprep
11 Mappe genetiche e fisiche Le mappe sono brevemente trattate ai soli fini di un «ripasso» delle loro caratteristiche, dato che ad esse si fa riferimento in diversi argormenti trattati nell ambito del Corso
12 Mappe fisiche e mappe genetiche Mappa genetica o Mappa di concatenazione: Rappresentazione della distanza che separa i geni, basata sui dati di ricombinazione genetica Mappa fisica: Rappresentazione della distanza che si basa sulla distanza fisica lungo il cromosoma (sovente espressa in bp) Relazione tra mappa genetica e mappa fisica La distanza della mappa genetica per le 3000Mb del genoma umano e' di circa 3000cM e pertanto 1cM corrisponde approssimativamente a una distanza di mappa fisica di 1Mb. In realta' il rapporto delle distanze di mappa genetica e fisica sui segmenti cromosomici spesso deviano da questo valore medio a causa della localizzazione non casuale dei chiasmi. I segmenti cromosomici contenenti gli "hot spots" di ricombinazione mostrano una piu' alta frequenza di ricombinazione e per tanto marcatori molecolari localizzati in questa zona sembrano piu' distanti del reale. In generale c'e' una frequenza di crossingover piu' alta in corrispondenza delle regioni subtelomeriche rispetto a quelle centromeriche.
13 Mappa genetica
14 Mappa fisica
15
16 Overview genetic markers Molecular markers abundance level of polymorphism locus specificity codominance of alleles reproducibility Allozymes low low yes yes high low RFLP high medium yes yes high high Minisatellites medium high no/yes no/yes high high PCR-sequencing low low yes yes high high RAPD high medium no no low low Microsatellites high high yes yes high low ISSR medium-high medium no no medium-high low labor-intensity SSCP low low yes yes medium low-medium CAPS low low-medium yes yes high low-medium SCAR low medium yes yes/no high low AFLP high medium no no/yes high medium TaqMan low yes no/yes high low RAD
17 Isoenzymes ( Allozymes are allelic variants of enzymes encoded by structural genes. Enzymes are proteins consisting of amino acids, some of which are electrically charged. As a result, enzymes have a net electric charge, based on the stretch of amino acids comprising the protein. When due to mutation an amino acid has been replaced, the net electric charge of the protein may have been altered. Because changes in electric charge affect the migration rate of proteins in an electric field, allelic variation can be detected by gel-electrophoresis and subsequent specific enzymatic staining. Per enzyme usually two or more loci can be distinguished that have been termed isoloci. Therefore, allozyme variation is also referred to as isozyme variation. Synonyms Isozymes Analytical procedures Preparation of tissue homogenates Separation of the polymorphisms by polyacrylamide or starch gel-electrophoresis Visualization of the polymorphisms by specific enzymatic staining Main Requirements Laboratory setup for gel-electrophoresis Facilities/equipment to maintain gels at 2-5 C during electrophoresis Advantages No DNA extraction involved No primers or probes required Quick and easy to assay Low costs involved Zymograms can be interpreted in terms of loci and alleles Codominance of alleles High reproducibility Disadvantages Low abundance Low level of polymorphism Zymograms sometimes difficult to interpret due to complex banding profiles Proteins with identical electrophoretic mobility (co-migration) may not be homologous Profilo enzimatico influenzato dall ambiente (es. perossidasi indotte in condizioni di stress ossidativo)
18 RFLP (Restriction Fragment Length Polymorphism) RFLPs are fragments of restricted DNA (usually within the 2-10 kb range) separated by gel electrophoresis and detected by subsequent Southern blot hybridization to a radiolabeled DNA probe consisting of a sequence homologous to a specific chromosomal region. The locus specific probes, consisting of a sequence of unknown identity or part of the sequence of a cloned gene, are obtained by molecular cloning and isolation of suitable DNA fragments. Sequence variation affecting the occurrence (absence or presence) of endonuclease recognition sites is considered to be main cause of length polymorphisms. RFLPs are generally found to be moderately polymorphic and can be applied in comparisons ranging from the individual level to closely related species. Because of their high genomic abundance and random distribution throughout the genome, RFLPs have frequently been used in gene mapping studies. Analytical procedures Extraction of DNA Digestion of DNA by endonuclease restriction Separation of the fragments by agarose gel-electrophoresis Transference of the fragments to a nylon filter by Southern blotting Hybridization of fragments to a locus specific radiolabeled DNA probe Visualization of the polymorphisms by autoradiography Advantages High genomic abundance Random distribution throughout the genome Band profiles can be interpreted in terms of loci and alleles Codominance of alleles High reproducibility Filters can be washed and reprobed several times Disadvantages Large quantities of purified, high molecular weight DNA required (5-10 µg) Laborious and technically demanding High costs involved Additional development costs in case suitable probes are unavailable Not amenable to automation
19 RFLP (restriction fragment length polymorphism )
20 RFLP (restriction fragment length polymorphism )
21 CAPS (Cleaved Amplified Polymorphic Sequence) CAPS are DNA fragments amplified by the Polymerase Chain Reaction (PCR) using specific bp primers, followed by digestion with a restriction endonuclease. Subsequently, length polymorphisms resulting from variation in the occurrence of restriction sites are identified by gel-electrophoresis of the digested products. In comparison with RFLP-analysis, polymorphisms are more difficult to identify because of the limited size of the amplified fragments ( bp). CAPS-analysis, however, does not require time-consuming Southern blot hybridization and radioactive detection. CAPS have been applied predominantly in gene mapping studies. Analytical procedures Extraction of DNA Amplification of DNA fragments by PCR Digestion of the amplified fragments by endonuclease restriction Separation of the fragments by agarose or polyacrylamide gel-electrophoresis Visualization of the polymorphisms by ethidium-bromide staining and ultraviolet light (agarose gels) or silver staining (polyacrylamide gels) Advantages Low quantities of template DNA required ( ng per reaction) Band profiles can be interpreted in terms of loci and alleles Codominance of alleles High reproducibility Disadvantages Sequence data required for primer construction
22 marker B4 is a cleaved amplified polymorphism sequence (CAPS) marker.
23 RAPD (Random Amplified Polymorphic DNA) RAPDs are DNA fragments amplified by the Polymerase Chain Reaction (PCR) using short (generally 10 bp) synthetic primers of random sequence. These oligonucleotides serve as both forward and reverse primer and usually are able to amplify fragments from 3-10 genomic sites simultaneously. Amplified fragments (within the kb range) are separated by gel-electrophoresis and polymorphisms are detected as the presence or absence of bands of particular size. These polymorphisms are considered to be primarily due to variation in the primer annealing sites. RAPDs have been used for many purposes, ranging from studies at the individual level (e.g. genetic identity) to studies involving closely related species. Due to their very high genomic abundance, RAPDs have also been applied in gene mapping studies. Analytical procedures Extraction of DNA Amplification of DNA fragments by PCR Separation of the polymorphisms by agarose gel-electrophoresis (AP-PCR and DAF fragments are usually separated on polyacrylamide gels) Visualization of the polymorphisms by ethidium-bromide staining and ultraviolet light (AP-PCR and DAF fragments are usually visualized by silver staining or autoradiography) Advantages Low quantities of template DNA required (5-50 ng per reaction) No sequence data for primer construction required Low costs involved Very high genomic abundance Random distribution throughout the genome Generation of multiple bands per reaction Amenable to automation Disadvantages Purified, high molecular weight DNA required Precautions are needed to avoid contamination of DNA because short, random primers are used that are able to amplify DNA fragments in a variety of organisms Highly standardized experimental procedures are needed because of sensitivity to reaction conditions Dominance of alleles Low reproducibility Similar sized fragments may not be homologous
24 RAPD (Random amplified polymorphic DNA )
25 RAPD (Random amplified polymorphic DNA )
26 SCAR (Sequence Characterized Amplified Region) SCARs are DNA fragments amplified by the Polymerase Chain Reaction (PCR) using specific bp primers, designed from nucleotide sequences established in cloned RAPD (Random Amplified Polymorphic DNA) fragments linked to a trait of interest. By using longer PCR primers, SCARs do not face the problem of low reproducibility generally encountered with RAPDs. SCARs are locus specific and have been widely applied in gene mapping studies and marker assisted selection. Analytical procedures Extraction of DNA Amplification of DNA fragments by PCR Separation of the polymorphisms by agarose gel-electrophoresis Visualization of the polymorphisms by ethidium-bromide staining and ultraviolet light Advantages Low quantities of template DNA required ( ng per reaction) Easy and quick to assay High reproducibility Amenable to automation Disadvantages Sequence data required for primer construction
27 AFLP (Amplified Fragment Length Polymorphism) AFLP TM is a trademark of Keygene (Wageningen). AFLPs are DNA fragments ( bp) obtained from endonuclease restriction, followed by ligation of oligonucleotide adapters to the fragments and selective amplification by the Polymerase Chain Reaction (PCR). The PCR-primers consist of a core sequence (part of the adapter), a restriction enzyme specific sequence and 1-3 selective nucleotides. The AFLP-technique simultaneously generates fragments from many genomic sites (usually fragments per reaction) that are separated by gel-electrophoresis and generally scored as a dominant marker. However, by using automatic gel scanners, heterozygotes may be distinguished from homozygotes based on band intensity differences. Because of the highly informative fingerprinting profiles generally obtained, AFLPs can be applied in studies involving genetic identity, parentage and identification of clones and cultivars. Due to their high genomic abundance and random distribution throughout the genome, AFLPs are also considered relevant markers in gene mapping studies. Analytical procedures Extraction of DNA Digestion of DNA by endonuclease restriction and ligation of oligonucleotide adapters to the DNA fragments Selective amplification of part of the DNA fragments Separation of the fragments by polyacrylamide gel-electrophoresis Visualization of the polymorphisms by autoradiography, silver staining or fluorescence Advantages No sequence data for primer construction required High genomic abundance Random distribution throughout the genome, although clustering around centromers has been reported Generation of many informative bands per reaction High reproducibility Amenable to automation Disadvantages Purified, high molecular weight DNA required Dominance of alleles Similar sized fragments may not be homologous
28 Amplified Fragment Length Polymorphisms (AFLPs)
29
30 SSCP (Single-Strand Conformation Polymorphism) SSCPs are DNA fragments ( bp) amplified by the Polymerase Chain Reaction (PCR) using specific bp primers, followed by gel-electrophoresis of single-strand DNA to detect nucleotide sequence variation. The method is based on the fact that the electrophoretic mobility of single-strand DNA highly depends on the secondary structure (conformation) of the molecule, which changes significantly in case of mutation. SSCP provides a method to detect nucleotide variation without the need to sequence DNA samples. SSCPs have been applied to detect mutations in genes using gene sequence information for primer construction. Related techniques DGGE (Denaturing Gradient Gel Electrophoresis): separation of DNA fragments due to mobility differences under increasing denaturing conditions (formamide/urea concentrations) TGGE (Thermal Gradient Gel Electrophoresis): similar to DGGE, but subjected to heath-denaturing conditions Analytical procedures Extraction of DNA Amplification of DNA fragments by PCR Denaturation of the amplified fragments to a single-strand form Separation of the polymorphisms by polyacrylamide gel-electrophoresis Visualization of the polymorphisms by silver staining or autoradiography Advantages Low quantities of template DNA required ( ng per reaction) Band profiles can be interpreted in terms of loci and alleles Codominance of alleles Disadvantages Sequence data required for primer construction Highly standardized electrophoretical conditions are needed to obtain reproducible results Absence of mutation cannot be proven because some mutations may remain undetected
31 Denaturing Gradient Gel Electrophoresis (DGGE) PCR primer with a 5' tail consisting of a sequence of 40 GC!
32
33 Microsatellites Microsatellites are molecular marker loci consisting of tandem repeat units of very short (1-5 basepairs) nucleotide motif. In case the nucleotide sequences in the flanking regions of the microsatellite are known, specific primers (generally bp) can be designed to amplify the microsatellite by the Polymerase Chain Reaction (PCR). Polymerase slippage during DNA replication (or slipped strand mispairing) is considered to be the main cause of variation in the number of repeat units, resulting in length polymorphisms that can be detected by gel-electrophoresis. Due to their high level of polymorphism, microsatellites are informative markers that can be used for many population genetic purposes, ranging from the individual level (e.g. clone and strain identification) to closely related species. Synonyms SSLP (Simple Sequence Length Polymorphisms); SSR (Simple Sequence Repeats); STMS (Sequence Tagged Microsatellites) Analytical procedures Extraction of DNA Amplification of DNA fragments by PCR Separation of the polymorphisms by polyacrylamide Visualization of the polymorphisms by autoradiography, silver staining or fluorescence (polyacrylamide gels), or ethidium-bromide staining and ultraviolet light (agarose gels) Advantages Low quantities of template DNA required ( ng per reaction) High genomic abundance Random distribution throughout the genome High level of polymorphism Codominance of alleles Allele sizes can be determined with an accuracy of 1 bp, allowing accurate comparison across different gels High reproducibility Different microsatellites may be multiplexed in PCR or on gel Wide range of applications Amenable to automation Disadvantages High development costs in case primers are not yet available Heterozygotes may be misclassified as homozygotes when null-alleles occur due to mutation in the primer annealing sites
34 Microsatelliti Microsatellite DNA is thus a class of repetitive DNA based on dinucleotide repeats. The most common type consists of repeats of CA and its complement GT, as in the following example:
35 Figura SSR When the repeating unit is less than four, the VNTR is called a microsatellite and when the repeating unit is longer it is a minisatellite. I microsatelliti sono anche chiamati Simple Sequence Repeat (SSR)
36 Minisatellites Disadvantages Large quantities of purified, high molecular weight DNA required (5-10 µg) Laborious and technically demanding High costs involved Distribution across the genome may be non-random Similar sized fragments may not be homologous (in case of multilocus probes) Difficulties in comparing polymorphisms across different gels Minisatellites are molecular marker loci consisting of tandem repeat units of a base motif, flanked by conserved endonuclease restriction sites. They are detected by gel electrophoresis of restricted DNA and subsequent Southern blot hybridization to a radiolabeled DNA probe containing multiple copies of the minisatellite core sequence. A minisatellite profile consisting of many bands (within the 4-20 kb range) is generated by using common multilocus probes that are able to hybridize to minisatellite sequences in different species. Locus specific probes can be developed by molecular cloning of DNA restriction fragments and subsequent screening with a multilocus minisatellite probe. Variation in the number of repeat units is considered to be the main cause of length polymorphisms. Due to the high mutation rate of minisatellites, the level of polymorphism is substantial, generally resulting in unique multilocus profiles. Therefore, minisatellites are particularly useful in studies involving genetic identity, parentage, clonal growth and structure, and identification of varieties and cultivars Synonyms DNA fingerprinting; VNTR (Variable Number of Tandem Repeats) Analytical procedures Extraction of DNA Digestion of DNA by endonuclease restriction Separation of the fragments by agarose gel-electrophoresis Transference of the fragments to a nylon filter by Southern blotting Hybridization of fragments to a radiolabeled minisatellite probe Visualization of the polymorphisms by autoradiography Advantages High level of polymorphism Generation of many informative bands per reaction (in case of multilocus probes) High reproducibility
37 Obtaining a DNA fingerprint by using a VNTR probe. (a) Preparation of the probe. The first intron of the myoglobin gene has four repeats of the sequence shown, which contains a 13- bp core sequence (shown in boldface). This core sequence is found at other VNTR loci, labeled VNTR I, II, and III in this simple diagrammatic representation. (b) The number of repeats at the three VNTR loci with the core sequence. The Southern blot has been probed with the 33-bp repeat in part a and shows the DNA fingerprints of three people.
38 Fig. 5 Détection des réarrangements du minisatellite CEB1-1.8 et CEB1-0.6 dans les cellules rad27δ de S. cerevisiae. L'instabilité se manifeste par l'apparition de variants de nouvelles tailles, plus courtes ou plus longues que celles des allèles parentaux (indiqués par des flèches).
39 SNP
40 SNP (single nucleotide polymorphism Is a DNA sequence variation occurring when a single nucleotide A, T, C, or G in the genome (or other shared sequence) differs between members of a species (or between paired chromosomes in an individual). For example, two sequenced DNA fragments from different individuals, AAGCCTA to AAGCTTA, contain a difference in a single nucleotide. In this case we say that there are two alleles : C and T. Almost all common SNPs have only two alleles. Variations in the DNA sequences of humans can affect how humans develop diseases and respond to pathogens, chemicals, drugs, vaccines, and other agents. SNPs are also thought to be key enablers in realizing the concept of personalized medicine. [3] However, their greatest importance in biomedical research is for comparing regions of the genome between cohorts (such as with matched cohorts with and without a disease). The study of single-nucleotide polymorphisms is also important in crop and livestock breeding programs
41
42 Advantages and disadvantages of various DNA fingerprinting methods Type Advantages Disadvantages Isozymes RAPD AFLP SSR RFLP SNP Inexpensive Co-dominant Expressed genes Inexpensive Simple High discrimination Many markers High discrimination Highly reproducible Co-dominant Characterized DNA Highly reproducible Co-dominant Characterized DNA High discrimination Co-dominant The small number of isozymes available can lead to poor discrimination capacity between samples. Poor resolution of electrophoretic bands. Markers can be difficult to differentiate because of complex banding patterns of oligomeric isozymes and co-migration in electrophoresis Uncharacterized DNA which is amplified in RAPD provides no additional information, which is sometimes a useful property of other marker systems. Low stringency of the PCR process used in RAPD can result in profile reproducibility problems. Unknown gene function Expensive Complex procedure Unknown gene function Complex procedure to establish initially for given taxa. Expensive (unless using extant SSR primers). Taxaspecific, there are limitations to the transferability of SSR markers between taxa; only successful with closely related genera Unknown gene function (unless EST-derived) Complex procedure Laborious Complex to establish initially for given taxa
43 Restriction site Associated DNA (RAD) markers Restriction site associated DNA (RAD) markers can be identified and typed by detecting differential hybridization patterns of RAD tags on a microarray. Genomic DNA samples S1 and S2 contain the recognition sequence for various restriction enzymes at locations throughout the genome. Dark blue triangles represent restriction sites of a particular enzyme. Some of these restriction sites are only present in one sample because of polymorphisms that disrupt the recognition sequence (red asterisks). The two samples are separately digested with a particular restriction enzyme and then ligated to biotinylated linkers (light blue ellipses). The DNA is randomly sheared leaving only the fragments that were directly flanking a restriction site attached to biotin linkers. These fragments are purified using streptavidin beads and released by digestion at the original restriction site. Loci containing polymorphisms, such as the left locus of S2 or the right locus of S1, will not contain tags for that locus in the purified RAD-tag sample, thus resulting in differential hybridization patterns of RAD tags on a microarray. Miller M R et al. Genome Res. 2007;17:
44 Enriched RAD marker microarray production and characterization Enriched RAD marker microarray production and characterization. RAD-tag samples S1 and S2 contain polymorphic sets of RAD tags. RAD tags that are present in both individuals will not serve as informative markers. In order to produce an array that types a large number of informative markers, subtractive hybridization is used to enrich for sample-specific RAD tags. RAD-tag clone libraries are generated from these enriched samples. These clone libraries are used as templates for PCR, the products of which are spotted to produce RAD marker microarrays. To identify informative markers, RAD-tag samples S1 and S2 are fluorescently labeled and competitively hybridized directly against each other to the array. Miller M R et al. Genome Res. 2007;17:
45 The process of RADSeq The process of RADSeq (A) Genomic DNA is sheared with a restriction enzyme of choice (SbfI in this example). (B) P1 adapter is ligated to SbfI-cut fragments. The P1 adapter is adapted from the Illumina sequencing adapter (full sequence not shown here), with a molecular identifier (MID; CGATA in this example) and a cut site overhang at the end (TGCA in this example). (C) Samples from multiple individuals are pooled together and all fragments are randomly sheared. Only a subset of the resulting fragments contains restriction sites and P1 adapters. (D) P2 adapter is ligated to all fragments. The P2 adapter has a divergent end. (E) PCR amplification with P1 and P2 primers. The P2 adapter will be completed only in the fragments ligated with P1 adapter, and so only these fragments will be fully amplified. (F) Pooled samples with different MIDs are separated bioinformatically and SNPs called (C/G SNP underlined). (G) As fragments are sheared randomly, paired end sequences from each sequenced fragment will cover a bp region downstream of the restriction site. Davey J W, Blaxter M L Briefings in Functional Genomics 2010;9:
46 Gene tagging Transposon tagging T-DNA tagging Activation tagging
47 Activation TAGGING Transposon tagging
48
49
50 TAIL-PCR is very simple, efficient, and highly specific. Since no other manipulations apart from PCR are required, TAIL-PCR is especially suitable for isolation of targeted unknown sequences from a large number of samples. This technique has been used to recover insert ends from rice BAC clones for chromosome walking and mapping.
51 AIMS (Amplification of insertion mutagenised sites)
52 Amplification of insertion mutagenised sites (AIMS)
53 Reverse genetics Reverse genetics is an approach to discovering the function of a gene that proceeds oppositely to how such discoveries typically unfold in classical genetics, or in forward genetics. By the classical approach, geneticists first look for rare individuals with unusual traits or phenotypes, and then they trace these traits to an underlying faulty allele or gene. Locating the gene on its chromosome is the end point of an investigation. With the readily performed modern techniques of DNA sequencing and as a result of the sequencing of many whole genomes, many genetic sequences are discovered in advance of any other information about them. To learn the influence a sequence has on phenotype, or to discover its biological function, researchers may engineer a: Change or disruption by site-directed mutagenesis, for example, or by deletion of a gene by gene knockout (as can be done in some organisms, such as yeast and mice) -and only afterwards look for the effect of such alterations in the whole organism. The discovery of gene silencing using double stranded RNA also known as RNA interference has also made this approach very promising. A third reverse genetics technique is the creation of transgenic organisms that overexpress a gene of interest. The resulting phenotype may reflect the normal function of the gene.
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Marcatori molecolari nelle piante
 Marcatori molecolari nelle piante Mappa genetica: rappresentazione grafica dei singoli gruppi di associazione con l indicazione dell ordine e della posizione relativa dei geni lungo il cromosoma determinata
Marcatori molecolari nelle piante Mappa genetica: rappresentazione grafica dei singoli gruppi di associazione con l indicazione dell ordine e della posizione relativa dei geni lungo il cromosoma determinata
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
DNA Profiling e Genetica Forense. Silvia Fuselli Lezione 1
 DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
Marcatori molecolari per l iden3ficazione varietale. Tracciabilità molecolare
 Marcatori molecolari per l iden3ficazione varietale Tracciabilità molecolare Regolamento Europeo 178/2002 Nella tracciabilità rientrano le a.vità volte a iden1ficare le sostanze che cos1tuiscono il cibo
Marcatori molecolari per l iden3ficazione varietale Tracciabilità molecolare Regolamento Europeo 178/2002 Nella tracciabilità rientrano le a.vità volte a iden1ficare le sostanze che cos1tuiscono il cibo
IBRIDAZIONE DNA:DNA. Tassonomia molecolare CHEMOTAXONOMY
 IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
MARCATORI MOLECOLARI
 MARCATORI MOLECOLARI MARCATORI ENZIMATICI A B D E C Marcatori biochimici: (A) fosfo-gluco-mutasi (PGM); (B) lipoossigenasi (LOX) in soia; (C) esterasi (EXT) in poa pratense; (D,E) rappresentazioni schematiche
MARCATORI MOLECOLARI MARCATORI ENZIMATICI A B D E C Marcatori biochimici: (A) fosfo-gluco-mutasi (PGM); (B) lipoossigenasi (LOX) in soia; (C) esterasi (EXT) in poa pratense; (D,E) rappresentazioni schematiche
Diagnostica molecolare dei disordini genomici
 Diagnostica molecolare dei disordini genomici MLPA (Multiplex Ligation-dependent Probe Amplification) Riconoscimento di variazioni del numero di copie in più di 40 distinte sequenze genomiche mediante
Diagnostica molecolare dei disordini genomici MLPA (Multiplex Ligation-dependent Probe Amplification) Riconoscimento di variazioni del numero di copie in più di 40 distinte sequenze genomiche mediante
SAFE - SCUOLA DI SCIENZE AGRARIE, FORESTALI, ALIMENTARI ED AMBIENTALI
 Programma di insegnamento per l anno accademico 2015/2016 Programma dell insegnamento di GENETICA ANIMALE E TRACCIABILITÀ Course title: ANIMAL GENETICS AND TRACEABILITY SSD dell insegnamento AGR/17 CFU
Programma di insegnamento per l anno accademico 2015/2016 Programma dell insegnamento di GENETICA ANIMALE E TRACCIABILITÀ Course title: ANIMAL GENETICS AND TRACEABILITY SSD dell insegnamento AGR/17 CFU
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
in Linkage Disequilibrium con RFLP 4p16.3 (1983) Gene clonato nel 1993 (!!)
 Còrea di Huntington descritta da George Huntington nel 1872, è detta anche còrea che in greco indica la danza alla base vi è una degenerazione programmata geneticamente dei neuroni dei gangli basali e
Còrea di Huntington descritta da George Huntington nel 1872, è detta anche còrea che in greco indica la danza alla base vi è una degenerazione programmata geneticamente dei neuroni dei gangli basali e
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
TILLING (Targeting Induced Local Lesions In Genomes) La mutagenesi tradizionale incontra la genomica funzionale
 TILLING (Targeting Induced Local Lesions In Genomes) La mutagenesi tradizionale incontra la genomica funzionale Nell era genomica attuale, le potenti strategie di reverse genetics sono necessarie per studiare
TILLING (Targeting Induced Local Lesions In Genomes) La mutagenesi tradizionale incontra la genomica funzionale Nell era genomica attuale, le potenti strategie di reverse genetics sono necessarie per studiare
DNA revolution. Microsatellites SNPs. Sequenziamento massivo (Next Generation Sequencing NGS)
 DNA revolution Microsatellites SNPs Sequenziamento massivo (Next Generation Sequencing NGS) NGS NGS NGS Non-Sanger-based high-throughput DNA sequencing technology Increasing the throughput Minimizing the
DNA revolution Microsatellites SNPs Sequenziamento massivo (Next Generation Sequencing NGS) NGS NGS NGS Non-Sanger-based high-throughput DNA sequencing technology Increasing the throughput Minimizing the
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Analysis of MOlecular VAriance (AMOVA) Alessandra Nardi
 Analysis of MOlecular VAriance (AMOVA) Alessandra Nardi alenardi@mat.uniroma2.it 1 Vediamo come gli elementi base dell Analisi delle Varianza possano essere utilizzati per studiare la variabilità di dati
Analysis of MOlecular VAriance (AMOVA) Alessandra Nardi alenardi@mat.uniroma2.it 1 Vediamo come gli elementi base dell Analisi delle Varianza possano essere utilizzati per studiare la variabilità di dati
ANALISI MOLECOLARE DEL GENOMA
 ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
CORSO GENOPOM PRO. Marcatori molecolari. Docente: Pasquale Termolino
 CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
Single-rate three-color marker (srtcm)
 3. Markers Pag. 1 The Single Rate Three Color Marker (srtcm) can be used as component in a Diffserv traffic conditioner The srtcm meters a traffic stream and marks its packets according to three traffic
3. Markers Pag. 1 The Single Rate Three Color Marker (srtcm) can be used as component in a Diffserv traffic conditioner The srtcm meters a traffic stream and marks its packets according to three traffic
Introduction to High Resolution Virulence Allelic Profiling (HReVAP)
 Introduction to High Resolution Virulence Allelic Profiling (HReVAP) Valeria Michelacci NGS course, June 2016 The E. coli pangenome Genomic plasticity Huge pangenome Van Elsas J.D. et al., 2011 Pangenome
Introduction to High Resolution Virulence Allelic Profiling (HReVAP) Valeria Michelacci NGS course, June 2016 The E. coli pangenome Genomic plasticity Huge pangenome Van Elsas J.D. et al., 2011 Pangenome
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Marcatori genetici delle piante
 Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI
 Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Medicina Molecolare e Modelli Animali di Malattia
 Medicina Molecolare e Modelli Animali di Malattia PROF. AF. CAMPESE LEZIONE 1 Generazione, screening e mantenimento di modelli murini geneticamente modificati convenzionali Dept. of Molecular Medicine
Medicina Molecolare e Modelli Animali di Malattia PROF. AF. CAMPESE LEZIONE 1 Generazione, screening e mantenimento di modelli murini geneticamente modificati convenzionali Dept. of Molecular Medicine
Scheduling. Scheduler. Class 1 Class 2 Class 3 Class 4. Scheduler. Class 1 Class 2 Class 3 Class 4. Scheduler. Class 1 Class 2 Class 3 Class 4
 Course of Multimedia Internet (Sub-course Reti Internet Multimediali ), AA 2010-2011 Prof. 4. Scheduling Pag. 1 Scheduling In other architectures, buffering and service occur on a per-flow basis That is,
Course of Multimedia Internet (Sub-course Reti Internet Multimediali ), AA 2010-2011 Prof. 4. Scheduling Pag. 1 Scheduling In other architectures, buffering and service occur on a per-flow basis That is,
UNIVERSITA' DEGLI STUDI DELLA BASILICATA DIPARTIMENTO DI SCIENZE
 Insegnamento: Biotecnologie genetiche Corso di studio: Anno di Corso: Periodo didattico: Tipologia: Totale Crediti: Tipo Esame: Valutazione: Lingua di insegnamento: Laurea in Biotecnologie (L2) III I semestre
Insegnamento: Biotecnologie genetiche Corso di studio: Anno di Corso: Periodo didattico: Tipologia: Totale Crediti: Tipo Esame: Valutazione: Lingua di insegnamento: Laurea in Biotecnologie (L2) III I semestre
Le zone e la tipicità
 Conegliano, 9/6/2018 LABORATORIO ENOCHIMICO Le zone e la tipicità Meneghetti S. PREMESSA Il genere Vitis è caratterizzato da una grande variabilità a livello morfologico, fisiologico e genetico; I viticoltori,
Conegliano, 9/6/2018 LABORATORIO ENOCHIMICO Le zone e la tipicità Meneghetti S. PREMESSA Il genere Vitis è caratterizzato da una grande variabilità a livello morfologico, fisiologico e genetico; I viticoltori,
REGOLAZIONE DELL ESPRESSIONE GENICA. Controllo trascrizionale in E. coli. Esempio: Lac operon
 REGOLAZIONE DELL ESPRESSIONE GENICA Controllo trascrizionale in E. coli Esempio: Lac operon Nel genoma di un batterio ci sono circa 4000 geni Nel genoma umano ci sono circa 25000 geni. Espressione costitutiva:
REGOLAZIONE DELL ESPRESSIONE GENICA Controllo trascrizionale in E. coli Esempio: Lac operon Nel genoma di un batterio ci sono circa 4000 geni Nel genoma umano ci sono circa 25000 geni. Espressione costitutiva:
Corso di Immunologia A.A Immunoglobuline. Isotipi, allotipi e idiotipi
 Corso di Immunologia A.A. 2011-12 Immunoglobuline Isotipi, allotipi e idiotipi 1 Immunoglobulin Isotypes Definition - Antigenic determinants that characterize the classes and subclasses of heavy chains
Corso di Immunologia A.A. 2011-12 Immunoglobuline Isotipi, allotipi e idiotipi 1 Immunoglobulin Isotypes Definition - Antigenic determinants that characterize the classes and subclasses of heavy chains
Constant Propagation. A More Complex Semilattice A Nondistributive Framework
 Constant Propagation A More Complex Semilattice A Nondistributive Framework 1 The Point Instead of doing constant folding by RD s, we can maintain information about what constant, if any, a variable has
Constant Propagation A More Complex Semilattice A Nondistributive Framework 1 The Point Instead of doing constant folding by RD s, we can maintain information about what constant, if any, a variable has
Utilizzo e valutazione dell'offerta pubblica e privata di servizi radiotelevisivi nel 2017
 Utilizzo e valutazione dell'offerta pubblica e privata di servizi radiotelevisivi nel 2017 Rapporto finale Benjamin Fretwurst, Céline Murri, Thomas Friemel, Heinz Bonfadelli Panoramica grafica 3 Fretwurst/Murri/Friemel/Bonfadelli
Utilizzo e valutazione dell'offerta pubblica e privata di servizi radiotelevisivi nel 2017 Rapporto finale Benjamin Fretwurst, Céline Murri, Thomas Friemel, Heinz Bonfadelli Panoramica grafica 3 Fretwurst/Murri/Friemel/Bonfadelli
Graphs: Cycles. Tecniche di Programmazione A.A. 2012/2013
 Graphs: Cycles Tecniche di Programmazione Summary Definitions Algorithms 2 Definitions Graphs: Cycles Cycle A cycle of a graph, sometimes also called a circuit, is a subset of the edge set of that forms
Graphs: Cycles Tecniche di Programmazione Summary Definitions Algorithms 2 Definitions Graphs: Cycles Cycle A cycle of a graph, sometimes also called a circuit, is a subset of the edge set of that forms
Studio sull ibridazione tra le specie Anisakis simplex s.s. e Anisakis pegreffii in aree di simpatria
 UNIVERSITA DEGLI STUDI DI PALERMO SCUOLA DELLE SCIENZE DI BASE ED APPLICATE CORSO DI LAUREA IN BIODIVERSITA ED EVOLUZIONE Studio sull ibridazione tra le specie Anisakis simplex s.s. e Anisakis pegreffii
UNIVERSITA DEGLI STUDI DI PALERMO SCUOLA DELLE SCIENZE DI BASE ED APPLICATE CORSO DI LAUREA IN BIODIVERSITA ED EVOLUZIONE Studio sull ibridazione tra le specie Anisakis simplex s.s. e Anisakis pegreffii
Accesso Mul*plo - modelli
 Accesso Mul*plo - modelli Conceptual Model of Mul/ple Access A B C D Station A Station B Station C Station D Master Channel The Master does not know if and how many packets are present in each queue (i.e.,
Accesso Mul*plo - modelli Conceptual Model of Mul/ple Access A B C D Station A Station B Station C Station D Master Channel The Master does not know if and how many packets are present in each queue (i.e.,
CRA- Centro di Ricerca per la Genomica e la Postgenomica Animale e Vegetale Fiorenzuola d Arda (PC) Tesi di Dottorato di Ricerca
 Study of malting quality and frost tolerance in the winter x spring 'Nure x Tremois doubled haploid population and development of a Marker-Assisted Selection programme Giovanni Laidò Università degli
Study of malting quality and frost tolerance in the winter x spring 'Nure x Tremois doubled haploid population and development of a Marker-Assisted Selection programme Giovanni Laidò Università degli
PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay)
 Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Electronic Supplementary Information for: protein KLHL40 changes upon nemaline myopathy. associated mutation
 Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 2016 Electronic Supplementary Information for: The dynamics of the β-propeller domain in Kelch protein
Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 2016 Electronic Supplementary Information for: The dynamics of the β-propeller domain in Kelch protein
Tavole del Cielo 1. Plates of the Sky. versione 02.00
 Tavole del Cielo 1 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Tavole del Cielo 1 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Obiettivi della genomica
 Obiettivi della genomica Stabilire database ed interfaccie di ricerca per le analisi genomiche. Ottenere e combinare mappe fisiche e genetiche del genoma Generare ed ordinare sequenze genomiche e sequenze
Obiettivi della genomica Stabilire database ed interfaccie di ricerca per le analisi genomiche. Ottenere e combinare mappe fisiche e genetiche del genoma Generare ed ordinare sequenze genomiche e sequenze
NOTICE. Palladium Securities 1 S.A. Series 112 EUR 100,000,000 Fixed to Floating Rate Instruments due 2023 (with EUR
 NOTICE Palladium Securities 1 S.A. Series 112 EUR 100,000,000 Fixed to Floating Rate Instruments due 2023 (with EUR 14,500,000 in principal amount outstanding) (the Instruments ) ISIN Code: XS0890631913
NOTICE Palladium Securities 1 S.A. Series 112 EUR 100,000,000 Fixed to Floating Rate Instruments due 2023 (with EUR 14,500,000 in principal amount outstanding) (the Instruments ) ISIN Code: XS0890631913
La GENETICA DELLE POPOLAZIONI. studia con modelli matematici, a livello di gruppi di individui, variabilità genetica
 La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
I marcatori molecolari. Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze
I marcatori molecolari Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze
lezione giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
Bell, Nature 421: 414, 2003. Tratta da lezioni di genetica Prof. PF Pignatti
 La scoperta della doppia elica mezzo secolo fa ha coinvolto la pratica medica con lentezza, ma sono probabili trasformazioni significative nei prossimi 50 anni. Bisogna cambiare la pratica medica e la
La scoperta della doppia elica mezzo secolo fa ha coinvolto la pratica medica con lentezza, ma sono probabili trasformazioni significative nei prossimi 50 anni. Bisogna cambiare la pratica medica e la
REGISTRATION GUIDE TO RESHELL SOFTWARE
 REGISTRATION GUIDE TO RESHELL SOFTWARE INDEX: 1. GENERAL INFORMATION 2. REGISTRATION GUIDE 1. GENERAL INFORMATION This guide contains the correct procedure for entering the software page http://software.roenest.com/
REGISTRATION GUIDE TO RESHELL SOFTWARE INDEX: 1. GENERAL INFORMATION 2. REGISTRATION GUIDE 1. GENERAL INFORMATION This guide contains the correct procedure for entering the software page http://software.roenest.com/
Applicazioni scientifiche
 Applicazioni scientifiche Gli enzimi di restrizione possono essere utilizzati per analizzare il DNA genomico mediante la tecnica del Southern blot. Il campione viene digerito con uno o più l'enzimi di
Applicazioni scientifiche Gli enzimi di restrizione possono essere utilizzati per analizzare il DNA genomico mediante la tecnica del Southern blot. Il campione viene digerito con uno o più l'enzimi di
Radar signal contains 2 types of information: intensity and phase intensity lets us discriminate among materials
 Radar signal contains types of information: intensity and phase intensity lets us discriminate among materials Phase is used to measure topography and ground motion Interferometry: interaction between
Radar signal contains types of information: intensity and phase intensity lets us discriminate among materials Phase is used to measure topography and ground motion Interferometry: interaction between
SRT064 BTH SRT051 BTH SRT052 BTH
 KIT FOR TRUCK BRAKE TESTERS SRT051 BTH SRT052 BTH OPERATOR S MANUAL SRT064BTH SRT051BTH SRT052BTH CONTENTS 1. INTRODUCTION...1 2. Description of SRT064BTH Kit...2 3. Description of SRT051BTH Kit...2 4.
KIT FOR TRUCK BRAKE TESTERS SRT051 BTH SRT052 BTH OPERATOR S MANUAL SRT064BTH SRT051BTH SRT052BTH CONTENTS 1. INTRODUCTION...1 2. Description of SRT064BTH Kit...2 3. Description of SRT051BTH Kit...2 4.
Genome variability part II
 Genome variability part II I MARCATORI POLIMORFICI 1. RFLPs Restriction Fragment Lenght Polymorphism (bi-allelic).tipizzabili mediante PCR or Southern based screening. Studi di associazione. 2. SNPs Single
Genome variability part II I MARCATORI POLIMORFICI 1. RFLPs Restriction Fragment Lenght Polymorphism (bi-allelic).tipizzabili mediante PCR or Southern based screening. Studi di associazione. 2. SNPs Single
GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Decode NGS data: search for genetic features
 Decode NGS data: search for genetic features Valeria Michelacci NGS course, June 2015 Blast searches What we are used to: online querying NCBI database for the presence of a sequence of interest ONE SEQUENCE
Decode NGS data: search for genetic features Valeria Michelacci NGS course, June 2015 Blast searches What we are used to: online querying NCBI database for the presence of a sequence of interest ONE SEQUENCE
ANALISI DEI PERICOLI Hazard Analysis
 7.4 ANALISI DEI PERICOLI Hazard Analysis Identificazione Valutazione Misure di Controllo Identification Assessment Control Measures Taccani 1 7.4.2 Identificare i pericoli e i livelli accettabili Identify
7.4 ANALISI DEI PERICOLI Hazard Analysis Identificazione Valutazione Misure di Controllo Identification Assessment Control Measures Taccani 1 7.4.2 Identificare i pericoli e i livelli accettabili Identify
TRE PAROLE CHIAVE DELLA GENETICA
 TRE PAROLE CHIAVE DELLA GENETICA Questo documento è pubblicato sotto licenza Creative Commons Attribuzione Non commerciale Condividi allo stesso modo http://creativecommons.org/licenses/by-nc-sa/2.5/deed.it
TRE PAROLE CHIAVE DELLA GENETICA Questo documento è pubblicato sotto licenza Creative Commons Attribuzione Non commerciale Condividi allo stesso modo http://creativecommons.org/licenses/by-nc-sa/2.5/deed.it
Technical Guidelines GON % Italian production. sports car oriented
 The rubber nozzle mod GON (Gas Oval Nozzle) has the intake with 210 mm x 105 mm dimensions and has been developed by WORKY in order to be more SPORTS CAR oriented. It has been studied for vehicles with
The rubber nozzle mod GON (Gas Oval Nozzle) has the intake with 210 mm x 105 mm dimensions and has been developed by WORKY in order to be more SPORTS CAR oriented. It has been studied for vehicles with
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) AFLP Amplified restriction fragment lenght polymorphisms Ogni genoma ha un certo
lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) AFLP Amplified restriction fragment lenght polymorphisms Ogni genoma ha un certo
Finite Model Theory / Descriptive Complexity: bin
 , CMPSCI 601: Recall From Last Time Lecture 19 Finite Model Theory / Descriptive Compleity: Th: FO L DSPACE Fagin s Th: NP SO. bin is quantifier-free.!#"$&% ('*), 1 Space 0 1 ) % Time $ "$ $ $ "$ $.....
, CMPSCI 601: Recall From Last Time Lecture 19 Finite Model Theory / Descriptive Compleity: Th: FO L DSPACE Fagin s Th: NP SO. bin is quantifier-free.!#"$&% ('*), 1 Space 0 1 ) % Time $ "$ $ $ "$ $.....
LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI
 Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Fiori di campo. Conoscere, riconoscere e osservare tutte le specie di fiori selvatici più note
 Fiori di campo. Conoscere, riconoscere e osservare tutte le specie di fiori selvatici più note M. Teresa Della Beffa Click here if your download doesn"t start automatically Fiori di campo. Conoscere, riconoscere
Fiori di campo. Conoscere, riconoscere e osservare tutte le specie di fiori selvatici più note M. Teresa Della Beffa Click here if your download doesn"t start automatically Fiori di campo. Conoscere, riconoscere
AVERE 30 ANNI E VIVERE CON LA MAMMA BIBLIOTECA BIETTI ITALIAN EDITION
 AVERE 30 ANNI E VIVERE CON LA MAMMA BIBLIOTECA BIETTI ITALIAN EDITION READ ONLINE AND DOWNLOAD EBOOK : AVERE 30 ANNI E VIVERE CON LA MAMMA BIBLIOTECA BIETTI ITALIAN EDITION PDF Click button to download
AVERE 30 ANNI E VIVERE CON LA MAMMA BIBLIOTECA BIETTI ITALIAN EDITION READ ONLINE AND DOWNLOAD EBOOK : AVERE 30 ANNI E VIVERE CON LA MAMMA BIBLIOTECA BIETTI ITALIAN EDITION PDF Click button to download
BIOCHIMICA MOLECOLARE CLINICA
 Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
Strumenti della Genetica Molecolare Umana (4)
 Strumenti della Genetica Molecolare Umana (4) Capitoli 6-7-8 SNPs La sostituzione di una base puo provocare la perdita o l insorgenza di un sito di restrizione, utilizzando PCR e digestione dell amplificato
Strumenti della Genetica Molecolare Umana (4) Capitoli 6-7-8 SNPs La sostituzione di una base puo provocare la perdita o l insorgenza di un sito di restrizione, utilizzando PCR e digestione dell amplificato
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Resources and Tools for Bibliographic Research. Search & Find Using Library Catalogues
 Resources and Tools for Bibliographic Research Search & Find Using Library Catalogues November 28, 2011 Donata Pieri Index Definition University of Padova Library System Catalogue CaPerE E-journals Catalogue
Resources and Tools for Bibliographic Research Search & Find Using Library Catalogues November 28, 2011 Donata Pieri Index Definition University of Padova Library System Catalogue CaPerE E-journals Catalogue
6.5 RNA Secondary Structure
 6.5 RNA Secondary Structure Struttura di una biomolecola Biomolecola: DNA, RNA Struttura primaria: descrizione esatta della sua composizione atomica e dei legami presenti fra gli atomi Struttura secondaria:
6.5 RNA Secondary Structure Struttura di una biomolecola Biomolecola: DNA, RNA Struttura primaria: descrizione esatta della sua composizione atomica e dei legami presenti fra gli atomi Struttura secondaria:
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo INGEGNERIA GENETICA
 CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più.
 Piante transgeniche prive di geni marker I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più. Possibili problemi una volta in
Piante transgeniche prive di geni marker I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più. Possibili problemi una volta in
Trombosi in sedi atipiche: il ruolo delle nuove mutazioni. Walter Ageno Dipartimento di Medicina Clinica Università dell Insubria Varese
 Trombosi in sedi atipiche: il ruolo delle nuove mutazioni Walter Ageno Dipartimento di Medicina Clinica Università dell Insubria Varese Trombosi venose in sedi atipiche Trombosi venose in sedi diverse
Trombosi in sedi atipiche: il ruolo delle nuove mutazioni Walter Ageno Dipartimento di Medicina Clinica Università dell Insubria Varese Trombosi venose in sedi atipiche Trombosi venose in sedi diverse
RNA Post-transcriptional processing
 RNA Post-transcriptional processing ESE: exonic splicing enhancer Spliceosoma Negli organismi superiori gli esoni non codificano solamente per le sequenze aminoacidiche ma contengono anche ESE Mutazioni
RNA Post-transcriptional processing ESE: exonic splicing enhancer Spliceosoma Negli organismi superiori gli esoni non codificano solamente per le sequenze aminoacidiche ma contengono anche ESE Mutazioni
Lezione 5. Next Generation Sequencing
 Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Tavole del Cielo 3. Plates of the Sky. versione 02.00
 Tavole del Cielo 3 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Tavole del Cielo 3 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Tavole del Cielo 2. Plates of the Sky. versione 02.00
 Tavole del Cielo 2 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Tavole del Cielo 2 Plates of the Sky versione 02.00 Questa guida è parte di una collana dedicata all insegnamento dell astronomia in specifiche manifestazioni ed eventi. This guide is part of a series
Estendere Lean e Operational Excellence a tutta la Supply Chain
 Estendere Lean e Operational Excellence a tutta la Supply Chain Prof. Alberto Portioli Staudacher www.lean-excellence.it Dipartimento Ing. Gestionale Politecnico di Milano alberto.portioli@polimi.it Lean
Estendere Lean e Operational Excellence a tutta la Supply Chain Prof. Alberto Portioli Staudacher www.lean-excellence.it Dipartimento Ing. Gestionale Politecnico di Milano alberto.portioli@polimi.it Lean
Sequenziamento del DNA: strutture dei nucleotidi
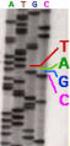 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Estensioni della genetica mendeliana
 Estensioni della genetica mendeliana Basi molecolari della dominanza e mutazioni Basi molecolari della dominanza e mutazioni Variazioni della dominanza ANIMAZIONE Dominanza incompleta Variazioni della
Estensioni della genetica mendeliana Basi molecolari della dominanza e mutazioni Basi molecolari della dominanza e mutazioni Variazioni della dominanza ANIMAZIONE Dominanza incompleta Variazioni della
Miglioramento genetico assistito dalla genomica
 Miglioramento genetico assistito dalla genomica Miglioramento genetico assistito dalla genomica Selezione delle piante sulla base del loro profilo ai marcatori genetici molecolari e non sulla base del
Miglioramento genetico assistito dalla genomica Miglioramento genetico assistito dalla genomica Selezione delle piante sulla base del loro profilo ai marcatori genetici molecolari e non sulla base del
EU opportunities, Italian success A revolutionary diagnostic device for lung cancer
 EU opportunities, Italian success A revolutionary diagnostic device for lung cancer 1 Dicembre 2016 ore 10:00 Diatech Pharmacogenetics Via Ignazio Silone 1b, Jesi (AN) The main scope of LEONID project
EU opportunities, Italian success A revolutionary diagnostic device for lung cancer 1 Dicembre 2016 ore 10:00 Diatech Pharmacogenetics Via Ignazio Silone 1b, Jesi (AN) The main scope of LEONID project
ISTITUTO SUPERIORE DELLE COMUNICAZIONI E DELLE TECNOLOGIE DELL INFORMAZIONE ORGANISMO NOTIFICATO 0648 NOTIFIED BODY
 ISTITUTO SUPERIORE DELLE COMUNICAZIONI E DELLE TECNOLOGIE DELL INFORMAZIONE ORGANISMO NOTIFICATO 0648 NOTIFIED BODY Richiesta di Parere Application form for Expert Opinion Riferimento: Reference: Allegati
ISTITUTO SUPERIORE DELLE COMUNICAZIONI E DELLE TECNOLOGIE DELL INFORMAZIONE ORGANISMO NOTIFICATO 0648 NOTIFIED BODY Richiesta di Parere Application form for Expert Opinion Riferimento: Reference: Allegati
Laurea Magistrale in Biotecnologie molecolari e bioinformatica
 Laurea Magistrale in Biotecnologie molecolari e bioinformatica Approfondisce le conoscenze dei meccanismi molecolari dei sistemi viventi e le loro applicazioni con i metodi di indagine avanzati della biologia
Laurea Magistrale in Biotecnologie molecolari e bioinformatica Approfondisce le conoscenze dei meccanismi molecolari dei sistemi viventi e le loro applicazioni con i metodi di indagine avanzati della biologia
ISTI ISTI Pisa. Trasportare algoritmi di partizionamento di oggetti in ambito teoria dei concetti. Elvira Locuratolo & Jari Palomaki.
 ISTI ISTI Pisa Tampere University of Technology / Pori Intension Extension Trasportare algoritmi di partizionamento di oggetti in ambito teoria dei concetti Elvira Locuratolo & Jari Palomaki Concept Theory
ISTI ISTI Pisa Tampere University of Technology / Pori Intension Extension Trasportare algoritmi di partizionamento di oggetti in ambito teoria dei concetti Elvira Locuratolo & Jari Palomaki Concept Theory
Keep calm, observe and assess
 Keep calm, observe and assess Using the video sitcoms in Just Right to assess competences Data: 2 febbraio, 2017 Relatore: Roy Bennett 1 Just Right! & competences 2 Support Pearson Academy 3 SESSION AIMS
Keep calm, observe and assess Using the video sitcoms in Just Right to assess competences Data: 2 febbraio, 2017 Relatore: Roy Bennett 1 Just Right! & competences 2 Support Pearson Academy 3 SESSION AIMS
CALANDRINO. 4 People, 2 Bedrooms, 2 Bathrooms, Balcony - Air Co, WiFi - II Floor (38 steps) a month
 Y o u r R e n t a l s i n L u c c a 4 People, 2 Bedrooms, 2 Bathrooms, Balcony - Air Co, WiFi - II Floor (38 steps) - 1200 a month - 2 BEDROOMS - 2 FULL BATHROOMS - FURNISHED BALCONY Fully Air Conditioned,
Y o u r R e n t a l s i n L u c c a 4 People, 2 Bedrooms, 2 Bathrooms, Balcony - Air Co, WiFi - II Floor (38 steps) - 1200 a month - 2 BEDROOMS - 2 FULL BATHROOMS - FURNISHED BALCONY Fully Air Conditioned,
AVVISO n Settembre 2009 AIM Italia
 AVVISO n.14112 08 Settembre 2009 AIM Italia Mittente del comunicato : Borsa Italiana Societa' oggetto : dell'avviso Oggetto : Modifiche al mercato AIM Italia: orari di negoziazione/amendments to the AIM
AVVISO n.14112 08 Settembre 2009 AIM Italia Mittente del comunicato : Borsa Italiana Societa' oggetto : dell'avviso Oggetto : Modifiche al mercato AIM Italia: orari di negoziazione/amendments to the AIM
R.M.Dorizzi, Strumenti per interpretare gli esami di laboratorio? Forlì, 9 ottobre 2012
 1 Half of what you are taught as medical students will in 10 years have been shown to be wrong. And the trouble is, none of your teachers know which half. Sydney Burwell, Dean Harvard Medical School 1935-1949
1 Half of what you are taught as medical students will in 10 years have been shown to be wrong. And the trouble is, none of your teachers know which half. Sydney Burwell, Dean Harvard Medical School 1935-1949
Esperienze nel settore Automotive
 Esperienze nel settore Automotive Tekna Automazione e Controllo lavora nel campo dell automazione industrial fornendo prodotti, Sistemi e servizi per l automazione ed il controllo delle line di produzione.
Esperienze nel settore Automotive Tekna Automazione e Controllo lavora nel campo dell automazione industrial fornendo prodotti, Sistemi e servizi per l automazione ed il controllo delle line di produzione.
SYRINGE FILTER general
 SYRINGE FILTER general Not every filter is suitable for every application. In order to give you a rough overview of the appli-cation possibilities of our filters, we have put together an overview, which
SYRINGE FILTER general Not every filter is suitable for every application. In order to give you a rough overview of the appli-cation possibilities of our filters, we have put together an overview, which
Research Collection. Statistical decisions based directly on the likelihood function. Doctoral Thesis. ETH Library. Author(s): Cattaneo, Marco E.G.V.
 Research Collection Doctoral Thesis Statistical decisions based directly on the likelihood function Author(s): Cattaneo, Marco E.G.V. Publication Date: 2007 Permanent Link: https://doi.org/10.3929/ethz-a-005463829
Research Collection Doctoral Thesis Statistical decisions based directly on the likelihood function Author(s): Cattaneo, Marco E.G.V. Publication Date: 2007 Permanent Link: https://doi.org/10.3929/ethz-a-005463829
Materiali Intelligenti: introduzione. Corso Materiali Intelligenti e Biomimetici 1/03/2018
 Materiali Intelligenti: introduzione Corso Materiali Intelligenti e Biomimetici 1/03/2018 Organizzazione corso Materiali Intelligenti A.A. 2017/18 Parte Teorica 1. Definizione e classificazione 2. Proprietà
Materiali Intelligenti: introduzione Corso Materiali Intelligenti e Biomimetici 1/03/2018 Organizzazione corso Materiali Intelligenti A.A. 2017/18 Parte Teorica 1. Definizione e classificazione 2. Proprietà
Screening e Counseling genetico: quando e per quale paziente Anna Maria Di Blasio
 Screening e Counseling genetico: quando e per quale paziente Anna Maria Di Blasio Responsabile del Laboratorio di Genetica Medica Instituto Auxologico Italiano Milano Malattie Mendeliane o Monogeniche
Screening e Counseling genetico: quando e per quale paziente Anna Maria Di Blasio Responsabile del Laboratorio di Genetica Medica Instituto Auxologico Italiano Milano Malattie Mendeliane o Monogeniche
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
UNIVERSITA' DEGLI STUDI DELLA BASILICATA DIPARTIMENTO DI SCIENZE
 Programma di insegnamento per l a.a. 2015-2016 Insegnamento: BIOTECNOLOGIE MEDICO DIAGNOSTICHE AVANZATE Docente: ANGELA OSTUNI Corso di studio: LAUREA MAGISTRALE PER LA DIAGNOSTICA MEDICA, FARMACEUTICA
Programma di insegnamento per l a.a. 2015-2016 Insegnamento: BIOTECNOLOGIE MEDICO DIAGNOSTICHE AVANZATE Docente: ANGELA OSTUNI Corso di studio: LAUREA MAGISTRALE PER LA DIAGNOSTICA MEDICA, FARMACEUTICA
Biochimica e Biotecnologia molecolare
 Biochimica e Biotecnologia molecolare Tecniche di manipolazione del DNA Endonucleasi di restrizione. Separazione elettroforetica del DNA. Sintesi chimica e sequenziamento del DNA. Tecniche di blotting.
Biochimica e Biotecnologia molecolare Tecniche di manipolazione del DNA Endonucleasi di restrizione. Separazione elettroforetica del DNA. Sintesi chimica e sequenziamento del DNA. Tecniche di blotting.
Data Exploitation della missione PRISMA, precursore delle missioni iperspettrali nazionali
 Data Exploitation della missione PRISMA, precursore delle missioni iperspettrali nazionali Sede Agenzia Spaziale Italiana Roma 01-03/03/2017 Valutazione dell impatto ambientale di incendi: comparazione
Data Exploitation della missione PRISMA, precursore delle missioni iperspettrali nazionali Sede Agenzia Spaziale Italiana Roma 01-03/03/2017 Valutazione dell impatto ambientale di incendi: comparazione
