GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
|
|
|
- Fabiola Landi
- 6 anni fa
- Visualizzazioni
Transcript
1 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti Microarray e RNA-seq 2. La mappatura dei genomi Metil-seq Mappatura genetica Chip-seq Mappatura fisica 3. Il sequenziamento automatico del DNA Il principio del sequenziamento secondo Sanger Il sequenziamento su larga scala La lettura dei tracciati di sequenziamento 4. Il sequenziamento del genoma I nuovi metodi di sequenziamento Il sequenziamento gerarchico Il sequenziamento shogun Piattaforme di sequenziamento di interi genomi La verifica delle sequenze e assemblaggio dei contig 5. L annotazione del genoma Il sequenziamento delle EST Le caratteristiche funzionali delle sequenze genomiche 6. I Progetti Genoma Il Progetto Genoma Umano Il Progetto Genoma Animali Il Progetto Genoma Piante Il Progetto Genoma Microrganismi 7. Genotipizzazione Gli SNP e la variazione La genotipizzazione degli SNP
2
3 Sequenziamento per shotgun gerarchici Sequenziamento per shotgun dell intero genoma
4 High-throughputSequencing 1. First Generation Sequencing 2. Second (Next) Generation Sequencing 3. Third Generation Sequencing Cyclic-array sequencing based
5 First Generation Sequencing Sanger sequencing based For shotgun sequencing DNA is fragmented Clonedintoa plasmidvector Cyclic sequencing reaction Separation by electrophoresis Readout with fluorescent tags many clonal copies of a single plasmid insert within a spatially isolated bacterial colony
6 Next Generation Sequencingg Cyclic-array sequencing method For shotgun sequencing DNA is fragmented Adaptors ligated to fragments ArrayofPCR colonies Enzymatic extension with fluorescently tagged nucleotides Cyclic readout by imaging array manypcr ampliconspresentwithinsingle reaction volume
7 Polimerase colony Èuna amplificazione clonale, in vitro, di una singola molecola di DNA
8 Polony Polonies are colonies of PCR amplicons derived from a single molecule of nucleic acid. Thousands to millions of spatially distinct polonies, each with an effective reaction size on the order of nanoliters (10-9 L) to femtoliters (10-15 L), can be amplified simultaneously within an acrylamide gel. Enzymatic reactions on polonies (e.g. in situ hybridization; minisequencing) can be subsequently performed in parallel. PCR in emulsione PCR tramite ponti
9 Emulsion PCR Fragments, with adaptors, are PCR amplified within a water drop in oil. One primer is attached to the surface of a bead. Used by 454, SOLiDand Polonator.
10 Bridge PCR DNA fragments are flanked with adaptors. A flat surface coated with two types of primers, corresponding to the adaptors. Amplification proceeds in cycles, with one end of each bridge tethered to the surface. Used by Solexa(Illumina).
11 454 sequencing based on pyrosequencing chemistry (sequencing by synthesis),, uses an emulsion-based method to isolate and amplify DNA fragments in vitro. Fragments of nt in length are ligated to special DNA Capture Beads, one fragment per bead. The beads are captured in water droplets of a heat-stable water-in-oil emulsion; one bead per droplet. Amplification occurs in the droplets, containing PCR-reagents (microreactors). The 454 GS FLX platform gives read lengths of ~250bp, providing about 400,000 sequences and Mb per run.
12
13
14
15
16
17
18
19 Illumina Solexa sequencingtechnology is a platform based on massively parallel sequencing of millions of fragments using reversible terminator-based sequencing chemistry (modified Sanger). This technology relies on fragmented genomic DNA arranged on a planar, optically transparent surface. Attached DNA fragments are amplified to create an ultra-high density sequencing flow cell with Illumina Sole xa seq uenc ing techno logy is a platform based on massively parall el seq uenc ing of millions of fragmen ts using reversible terminatorbased sequenc ing of chem istry the (mod ified same Sanger). This techno template logy relies on fragmen(up ted genom ic to DN A arra ten nged on a million planar, op tically transpsingle-molecule arent surface. ~1,000 copies Attached DN A fragmen ts amplified to create an ultra -high dens ity sequencing flow cell with ~1,000 cop ies of the same temp late (up to ten million clusters per single-molecule clusters per square These temp lates are seq uenced using a four-color DN A seq uenc ing-by-syn thes is technology that emp loys square reve rsible terminators centimetre). with remov able fluoresc dyes. The These Solexa platform provides templates up to 70bp pe r read, with are a paired sequenced end capability of using a ~3050 million reads and 24G b per run. four-color DNA sequencing-by-synthesis technology that employs reversible terminators with removable fluorescent dyes. The Solexa platform provides up to bp per read, with a paired end capability of ~3050 million reads and 24Gb per run.
20
21
22
23
24 Illumina Flow Cell One flow cell Eight 1.4-mm wide channels Each channel can run up to twelve differently tagged libraries (Multiplexed Sequencing) Input requirement: μg (single- and paired-end reads), 10 μg(mate Pair reads) million reads (clusters) per flow cell, each containing ~1,000 copies of the same template
25
26 ABI SOLiD System is a highly accurate, massively parallel genomic analysis platform. Based on sequencing by ligation, generates DNA by measuring the serial ligation of an oligonucleotide. The SOLiD System generates over 20 gigabases and 400M tags per run, with system accuracy greater than 99.94%, due to 2 base encoding which enables unique error checking capability, providing higher confidence ineach call.
27 Library preparation
28 Emulsion PCR/Bead Enrichment
29 Bead Deposition
30 Sequencing by Ligation/Data Analysis
31
32
33 Sequenze corte Sequenzecorte, ma tecnologiain continua evoluzione: 454: 100 basi ? Solid: 25 basi ? Illumina: 32 basi ? Difficoltà di assemblare sequenze corte de novo, soprattutto per il problema delle sequenze ripetute complicato ancora di più rispettoa Sanger (lunghezzamedia 600bp)
34
35 The Polonator
36 SequenziamentoSangerad alta processività PREPARAZIONE DELLA LIBRERIA Frammentazione casuale del DNA genomico clonazione e trasformazione in batteri 7-10 giorni assumendo di possedere una piattaforma robotica per alta processività Raccolta delle colonie Purificazione del DNA dalle colonie Sequenziamento Sanger Elettroforesi capillare Settimane-anni, dipendentemente dalle dimensione del genoma (e copertura richiesta) e dal numero di sequenziatori capillari a disposizione Mappatura delle reads su un genoma di riferimento (o assemblaggio de novo)
37 Sequenziamentodi Nuova Generazione PREPARAZIONE DELLA LIBRERIA Frammentazione casuale del DNA genomico Ligazione degli adattatori 1-3 giorni Amplificazione clonale dei frammenti Sequenziamento nuova generazione Processamento delle immagini 1-7 giorni Mappatura delle reads su un genoma di riferimento (o assemblaggio de novo)
38 High-throughput Sequencing 1. First Generation Sequencing 2. Second (Next) Generation Sequencing 3. Third Generation Sequencing
39 Nanopore sequencing Nucleic acids driven through a nanopore Differences in conductance of pore provide readout Real-time monitoring of PCR activity Read-out by fluorescence resonance energy transfer between polymerase and nucleotides (VisiGen Biotechnologies approach) Waveguides allow direct observation of polymerase and fluorescently labeled nucleotides (Single Molecule SMRT sequencing)
40 Ion semiconductor sequencing DNA nanoball sequencing Helioscope(TM) single molecule sequencing Single Molecule SMRT(TM) sequencing Single Molecule real time(rnap) sequencing Nanopore DNA sequencing VisiGen Biotechnologies approach
41 cyclodextrin α-hemolysin Nanopore sequencing
42 IonTorrentSequencing
43 HeliScopeSingle MoleculeSequencertechnology True Single Molecule Sequencing (tsms) is a powerful new method capable of directly measuring single DNA molecules, without amplification. Helioscope sequencing uses DNA fragments with added polya tail adapters, which are attached to the flow cell surface. The next steps involve extension-based sequencing with cyclic washes of the flow cell with fluorescently labeled nucleotides (one nucleotide type at a time, as with the Sanger method). The incorporated nucleotides emit light that is detected by the HeliScope Single Molecule Sequencer. Tracking nucleotide incorporation on each strand determines the exact sequence of each individual DNA molecule. The reads are short, up to 55 bases per run
GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Lezione 5. Next Generation Sequencing
 Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Next Generation Sequencers: from the bacterial culture to raw data. Valeria Michelacci NGS course, June 2015
 Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
Summer School 2015. Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi
 Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS)
 Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS) Per il sequenziamento di genomi la metodica principalmente utilizzata fino a pochi anni fa era basata su una strategia enzimatica, metodo
Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS) Per il sequenziamento di genomi la metodica principalmente utilizzata fino a pochi anni fa era basata su una strategia enzimatica, metodo
NEXT- GENERATION DNA SEQUENCING
 NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
David Caramelli Dipartimento di Biologia Evoluzionistica Laboratori di Antropologia. david.caramelli@unifi.it
 Nuovi metodi di analisi negli studi sul DNA antico David Caramelli Dipartimento di Biologia Evoluzionistica Laboratori di Antropologia UnitàdiAntropologia Molecolare /Paleogenetica, Università difirenze
Nuovi metodi di analisi negli studi sul DNA antico David Caramelli Dipartimento di Biologia Evoluzionistica Laboratori di Antropologia UnitàdiAntropologia Molecolare /Paleogenetica, Università difirenze
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo INGEGNERIA GENETICA
 CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
IBRIDAZIONE DNA:DNA. Tassonomia molecolare CHEMOTAXONOMY
 IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
Genomica Servizio Sequenziamento DNA
 Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Basi Teoriche e Applicazioni delle Nuove Tecnologie Genomiche
 Corsi di laurea magistrale in: Biotecnologie agrarie e ambientali (LM-7) Biologia cellulare e molecolare (LM-6) Sicurezza e qualitàagroalimentare (LM-69 & LM-70) insegnamento di Basi Teoriche e Applicazioni
Corsi di laurea magistrale in: Biotecnologie agrarie e ambientali (LM-7) Biologia cellulare e molecolare (LM-6) Sicurezza e qualitàagroalimentare (LM-69 & LM-70) insegnamento di Basi Teoriche e Applicazioni
Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei
 ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI a) sequenziamento gerarchico (clone by clone) b) sequenziamento shotgun
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI a) sequenziamento gerarchico (clone by clone) b) sequenziamento shotgun
Tecniche biofisiche basate su nanopori
 Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
Materiale del corso: http://bioinformatica.uniroma2.it/gc2014/
 Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Avanzamento dei sistemi di sequenziamento
 Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Aggiornamenti di biologia molecolare nella diagnostica microbiologica. Pier Sandro Cocconcelli
 Aggiornamenti di biologia molecolare nella diagnostica microbiologica Pier Sandro Cocconcelli Istituto di Microbiologia Centro Ricerche Biotecnologiche Università Cattolica del Sacro Cuore Piacenza-Cremona
Aggiornamenti di biologia molecolare nella diagnostica microbiologica Pier Sandro Cocconcelli Istituto di Microbiologia Centro Ricerche Biotecnologiche Università Cattolica del Sacro Cuore Piacenza-Cremona
La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria
 La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria Il 15% del DNA associato ai centromeri, costituito da sequenze altamente ripetute (DNA satellite e
La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria Il 15% del DNA associato ai centromeri, costituito da sequenze altamente ripetute (DNA satellite e
Tecniche di biologia molecolare. Utilizzi delle endonucleasi e polimerasi
 Tecniche di biologia molecolare Utilizzi delle endonucleasi e polimerasi 4 PRINCIPALI TIPI DI ENZIMI AGISCONO SUL DNA: - NUCLEASI (idrolisi del legame fosfodiestere in oligonucleotidi) - LIGASI (formazione
Tecniche di biologia molecolare Utilizzi delle endonucleasi e polimerasi 4 PRINCIPALI TIPI DI ENZIMI AGISCONO SUL DNA: - NUCLEASI (idrolisi del legame fosfodiestere in oligonucleotidi) - LIGASI (formazione
Sequenziamento e analisi di genomi completi
 Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Digital PCR: the evolution of PCR. https://www.youtube.com/watch?v=wu3qkhiuc54
 Digital PCR: the evolution of PCR https://www.youtube.com/watch?v=wu3qkhiuc54 Digital PCR: the evolution of PCR The rare target may not be amplified by PCR because of competition for reagents from the
Digital PCR: the evolution of PCR https://www.youtube.com/watch?v=wu3qkhiuc54 Digital PCR: the evolution of PCR The rare target may not be amplified by PCR because of competition for reagents from the
PCR. (Reazione a Catena della Polimerasi) Kary Mullis
 POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
Sequenziamento del DNA: strutture dei nucleotidi
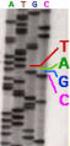 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Lezione XXII-XXllI martedì 24-XI-2011
 Lezione XXII-XXllI martedì 24-XI-2011 corso di genomica Laurea Magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 libro consigliato Zanichelli Introduz.
Lezione XXII-XXllI martedì 24-XI-2011 corso di genomica Laurea Magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 libro consigliato Zanichelli Introduz.
ncdna Per ncdna si intende il DNA intronico, intergenico e altre zone non codificanti del genoma.
 ncdna Per ncdna si intende il DNA intronico, intergenico e altre zone non codificanti del genoma. ncdna è caratteristico degli eucarioti: Sequenze codificanti 1.5% del genoma umano Introni in media 95-97%
ncdna Per ncdna si intende il DNA intronico, intergenico e altre zone non codificanti del genoma. ncdna è caratteristico degli eucarioti: Sequenze codificanti 1.5% del genoma umano Introni in media 95-97%
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
Lezione 4. Il sequenziamento del DNA, Sanger e il progetto genoma umano
 Lezione 4 Il sequenziamento del DNA, Sanger e il progetto genoma umano Stranneheim and Lundeberg 2012 Un po di storia 25 Aprile 1953 James D. Watson e Francis Crick pubblicano la struttura del DNA (Watson
Lezione 4 Il sequenziamento del DNA, Sanger e il progetto genoma umano Stranneheim and Lundeberg 2012 Un po di storia 25 Aprile 1953 James D. Watson e Francis Crick pubblicano la struttura del DNA (Watson
Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari
 Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari Roberto Bresciani Dipartimento di Medicina Molecolare e Traslazionale Università degli Studi di Brescia Biologia cellulare Biologia
Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari Roberto Bresciani Dipartimento di Medicina Molecolare e Traslazionale Università degli Studi di Brescia Biologia cellulare Biologia
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Lezione IX-X martedì 25-X-2011
 Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) AFLP Amplified restriction fragment lenght polymorphisms Ogni genoma ha un certo
lezione 19-20 martedi 13 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) AFLP Amplified restriction fragment lenght polymorphisms Ogni genoma ha un certo
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Marcatori molecolari nelle piante
 Marcatori molecolari nelle piante Mappa genetica: rappresentazione grafica dei singoli gruppi di associazione con l indicazione dell ordine e della posizione relativa dei geni lungo il cromosoma determinata
Marcatori molecolari nelle piante Mappa genetica: rappresentazione grafica dei singoli gruppi di associazione con l indicazione dell ordine e della posizione relativa dei geni lungo il cromosoma determinata
Esperienze nel settore Automotive
 Esperienze nel settore Automotive Tekna Automazione e Controllo lavora nel campo dell automazione industrial fornendo prodotti, Sistemi e servizi per l automazione ed il controllo delle line di produzione.
Esperienze nel settore Automotive Tekna Automazione e Controllo lavora nel campo dell automazione industrial fornendo prodotti, Sistemi e servizi per l automazione ed il controllo delle line di produzione.
Elettroforesi in campo pulsato su ceppi di S. aureus: presentazione del protocollo
 Elettroforesi in campo pulsato su ceppi di S. aureus: presentazione del protocollo Pulsed Field Gel Electrophoresis on strains of S. aureus: presentation of the protocol Walter Vencia IZS Piemonte, Liguria
Elettroforesi in campo pulsato su ceppi di S. aureus: presentazione del protocollo Pulsed Field Gel Electrophoresis on strains of S. aureus: presentation of the protocol Walter Vencia IZS Piemonte, Liguria
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010. Percorso nº 3: Clonaggio di segmenti di DNA
 Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010 Percorso nº 3: Clonaggio di segmenti di DNA 11-5-2010 I plasmidi: un mondo da esplorare. Elementi genetici capaci di replicarsi autonomamente
Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010 Percorso nº 3: Clonaggio di segmenti di DNA 11-5-2010 I plasmidi: un mondo da esplorare. Elementi genetici capaci di replicarsi autonomamente
PCR. Polymerase Chain Reaction
 PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
Tecniche di sequenziamento del DNA
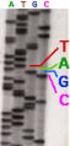 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Principali classi di mutazioni
 Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
UNIVERSITA' DEGLI STUDI DELLA BASILICATA DIPARTIMENTO DI SCIENZE
 Programma di insegnamento per l a.a. 2015-2016 Insegnamento: BIOTECNOLOGIE MEDICO DIAGNOSTICHE AVANZATE Docente: ANGELA OSTUNI Corso di studio: LAUREA MAGISTRALE PER LA DIAGNOSTICA MEDICA, FARMACEUTICA
Programma di insegnamento per l a.a. 2015-2016 Insegnamento: BIOTECNOLOGIE MEDICO DIAGNOSTICHE AVANZATE Docente: ANGELA OSTUNI Corso di studio: LAUREA MAGISTRALE PER LA DIAGNOSTICA MEDICA, FARMACEUTICA
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
L enigma del XXI secolo: decifrare il codice della vita
 L enigma del XXI secolo: decifrare il codice della vita David Horner Dipar9mento di Bioscienze Università degli Studi di Milano Via Celoria 26 Milano 20133 david.horner@unimi.it Regola di Chargaff %A =
L enigma del XXI secolo: decifrare il codice della vita David Horner Dipar9mento di Bioscienze Università degli Studi di Milano Via Celoria 26 Milano 20133 david.horner@unimi.it Regola di Chargaff %A =
Next-generation sequencing, annotazione, ed espressione genica. Giulio Pavesi Dip. Bioscienze Università di Milano giulio.pavesi@unimi.
 Next-generation sequencing, annotazione, ed espressione genica Giulio Pavesi Dip. Bioscienze Università di Milano giulio.pavesi@unimi.it Il primo passo... Abbiamo la sequenza completa del DNA di un organismo:
Next-generation sequencing, annotazione, ed espressione genica Giulio Pavesi Dip. Bioscienze Università di Milano giulio.pavesi@unimi.it Il primo passo... Abbiamo la sequenza completa del DNA di un organismo:
Biodiversità Molecolare: aspetti di base e nuove tecnologie
 Biodiversità Molecolare: aspetti di base e nuove tecnologie Graziano PESOLE Università di Bari & IBBE-CNR, Bari, Italy Seminari sulla Biodiversità Bari, 18 Febbraio 2011 La Biodiversità Per biodiversità
Biodiversità Molecolare: aspetti di base e nuove tecnologie Graziano PESOLE Università di Bari & IBBE-CNR, Bari, Italy Seminari sulla Biodiversità Bari, 18 Febbraio 2011 La Biodiversità Per biodiversità
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
Ibridazione degli acidi nucleici
 Ibridazione degli acidi nucleici L ibridazione consiste nel porre a contatto acidi nucleici a singolo filamento provenienti da fonti diverse: Sonda di solito una popolazione omogenea di molecole note.
Ibridazione degli acidi nucleici L ibridazione consiste nel porre a contatto acidi nucleici a singolo filamento provenienti da fonti diverse: Sonda di solito una popolazione omogenea di molecole note.
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Applicazioni scientifiche
 Applicazioni scientifiche Gli enzimi di restrizione possono essere utilizzati per analizzare il DNA genomico mediante la tecnica del Southern blot. Il campione viene digerito con uno o più l'enzimi di
Applicazioni scientifiche Gli enzimi di restrizione possono essere utilizzati per analizzare il DNA genomico mediante la tecnica del Southern blot. Il campione viene digerito con uno o più l'enzimi di
Metodica fu ideata nel 1983
 Metodica fu ideata nel 1983 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi
Metodica fu ideata nel 1983 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi
Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico Padano Pietro Piffanelli, Pamela Abbruscato, barbara Menin
 Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
PCR. Polymerase Chain Reaction
 PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
La diagnostica biomolecolare a supporto dei controlli ufficiali per le api regine d importazione
 La diagnostica biomolecolare a supporto dei controlli ufficiali per le api regine d importazione Raniero Lorenzetti Biotecnologie Istituto Zooprofilattico Sperimentale delle Regioni Lazio e Toscana PREMESSA
La diagnostica biomolecolare a supporto dei controlli ufficiali per le api regine d importazione Raniero Lorenzetti Biotecnologie Istituto Zooprofilattico Sperimentale delle Regioni Lazio e Toscana PREMESSA
A NOVEL FEM METHOD FOR PREDICTING INSTABILITY
 Presented at the COMSOL Conference 2009 Milan POLITECNICO DI BARI A NOVEL FEM METHOD FOR PREDICTING THERMOACOUSTIC COMBUSTION INSTABILITY G. Campa S.M. Camporeale COMSOL Conference 2009, 14-1616 October
Presented at the COMSOL Conference 2009 Milan POLITECNICO DI BARI A NOVEL FEM METHOD FOR PREDICTING THERMOACOUSTIC COMBUSTION INSTABILITY G. Campa S.M. Camporeale COMSOL Conference 2009, 14-1616 October
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Marcatori molecolari per l iden3ficazione varietale. Tracciabilità molecolare
 Marcatori molecolari per l iden3ficazione varietale Tracciabilità molecolare Regolamento Europeo 178/2002 Nella tracciabilità rientrano le a.vità volte a iden1ficare le sostanze che cos1tuiscono il cibo
Marcatori molecolari per l iden3ficazione varietale Tracciabilità molecolare Regolamento Europeo 178/2002 Nella tracciabilità rientrano le a.vità volte a iden1ficare le sostanze che cos1tuiscono il cibo
Lezione XLI-XLII martedì 17-1-2012
 Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
SRT064 BTH SRT051 BTH SRT052 BTH
 KIT FOR TRUCK BRAKE TESTERS SRT051 BTH SRT052 BTH OPERATOR S MANUAL SRT064BTH SRT051BTH SRT052BTH CONTENTS 1. INTRODUCTION...1 2. Description of SRT064BTH Kit...2 3. Description of SRT051BTH Kit...2 4.
KIT FOR TRUCK BRAKE TESTERS SRT051 BTH SRT052 BTH OPERATOR S MANUAL SRT064BTH SRT051BTH SRT052BTH CONTENTS 1. INTRODUCTION...1 2. Description of SRT064BTH Kit...2 3. Description of SRT051BTH Kit...2 4.
sottobasi per valvole a spola
 sottobasi per valvole a spola manifolds for spool valves Sottobasi modulari per valvole a spola /8 e /4 Multiple sub-bases for /8 and /4 spool valves Sottobasi a posti fissi per valvole a spola /8 e /4
sottobasi per valvole a spola manifolds for spool valves Sottobasi modulari per valvole a spola /8 e /4 Multiple sub-bases for /8 and /4 spool valves Sottobasi a posti fissi per valvole a spola /8 e /4
lezione martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
SAFE - SCUOLA DI SCIENZE AGRARIE, FORESTALI, ALIMENTARI ED AMBIENTALI
 Programma di insegnamento per l anno accademico 2015/2016 Programma dell insegnamento di GENETICA ANIMALE E TRACCIABILITÀ Course title: ANIMAL GENETICS AND TRACEABILITY SSD dell insegnamento AGR/17 CFU
Programma di insegnamento per l anno accademico 2015/2016 Programma dell insegnamento di GENETICA ANIMALE E TRACCIABILITÀ Course title: ANIMAL GENETICS AND TRACEABILITY SSD dell insegnamento AGR/17 CFU
I CAMBIAMENTI PROTOTESTO-METATESTO, UN MODELLO CON ESEMPI BASATI SULLA TRADUZIONE DELLA BIBBIA (ITALIAN EDITION) BY BRUNO OSIMO
 I CAMBIAMENTI PROTOTESTO-METATESTO, UN MODELLO CON ESEMPI BASATI SULLA TRADUZIONE DELLA BIBBIA (ITALIAN EDITION) BY BRUNO OSIMO READ ONLINE AND DOWNLOAD EBOOK : I CAMBIAMENTI PROTOTESTO-METATESTO, UN MODELLO
I CAMBIAMENTI PROTOTESTO-METATESTO, UN MODELLO CON ESEMPI BASATI SULLA TRADUZIONE DELLA BIBBIA (ITALIAN EDITION) BY BRUNO OSIMO READ ONLINE AND DOWNLOAD EBOOK : I CAMBIAMENTI PROTOTESTO-METATESTO, UN MODELLO
Leggere il Dna. Innovazione. Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica.
 Innovazione Leggere il Dna Rosario Iacono Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica. Nel 1977 fu pubblicato il metodo per il sequenziamento delle
Innovazione Leggere il Dna Rosario Iacono Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica. Nel 1977 fu pubblicato il metodo per il sequenziamento delle
Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016. Docente: Silvia Fuselli fss@unife.it
 Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016 Docente: Silvia Fuselli fss@unife.it Possibili testi di riferimento Introduction to Genomics, A.M. Lesk, Oxford Capitoli 1, 3,
Introduzione al corso di bioinformatica e analisi dei genomi AA 2015-2016 Docente: Silvia Fuselli fss@unife.it Possibili testi di riferimento Introduction to Genomics, A.M. Lesk, Oxford Capitoli 1, 3,
ENPCOM European network for the promotion of the Covenant of Mayors
 ENPCOM European network for the promotion of the Covenant of Mayors Censimento e monitoraggio dei consumi energetici e comportamento dei cittadini Controllo Energetico dei Consumi degli Edifici Monitoraggio
ENPCOM European network for the promotion of the Covenant of Mayors Censimento e monitoraggio dei consumi energetici e comportamento dei cittadini Controllo Energetico dei Consumi degli Edifici Monitoraggio
INSTALLAZIONE INSTALLATION
 INSTALLAZIONE INSTALLATION KIT SOFFIONI MySlim A CONTROSOFFITTO KIT MySlim SHOWER HEADS FOR FALSE CEILING Art. KIT00006 AVVERTENZE: L impianto deve essere effettuato da personale autorizzato, certificato
INSTALLAZIONE INSTALLATION KIT SOFFIONI MySlim A CONTROSOFFITTO KIT MySlim SHOWER HEADS FOR FALSE CEILING Art. KIT00006 AVVERTENZE: L impianto deve essere effettuato da personale autorizzato, certificato
Genotipizzazione virale
 Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Next-generation sequencing : dalla ricerca di base alla diagnostica*
 Next-generation sequencing : dalla ricerca di base alla diagnostica* Karl V. Voelkerding 1,2, Shale A. Dames 1, Jacob D. Durtschi 1 1ARUP Institute for Experimental and Clinical Pathology, Salt Lake City,
Next-generation sequencing : dalla ricerca di base alla diagnostica* Karl V. Voelkerding 1,2, Shale A. Dames 1, Jacob D. Durtschi 1 1ARUP Institute for Experimental and Clinical Pathology, Salt Lake City,
EVOLUZIONE MOLECOLARE. Silvia Fuselli
 EVOLUZIONE MOLECOLARE Silvia Fuselli silvia.fuselli@unife.it TESTI Organizzazione del corso Graur and Li, Fundamentals of molecular evolution, Sinauer 2000 Michael Lynch, The Origins of Genome Architecture,
EVOLUZIONE MOLECOLARE Silvia Fuselli silvia.fuselli@unife.it TESTI Organizzazione del corso Graur and Li, Fundamentals of molecular evolution, Sinauer 2000 Michael Lynch, The Origins of Genome Architecture,
Modificazione del 1 gennaio 2017
 Ornanza del DFI del 29 settembre 1995 sulle prestazioni dell assicurazione obbligatoria delle cure meco-sanitarie (Ornanza sulle prestazioni, OPre, allegato 3) RS 832.112.31 Moficazione del 1 gennaio 2017
Ornanza del DFI del 29 settembre 1995 sulle prestazioni dell assicurazione obbligatoria delle cure meco-sanitarie (Ornanza sulle prestazioni, OPre, allegato 3) RS 832.112.31 Moficazione del 1 gennaio 2017
Appendice A. Conduttori elettrici, sezioni e diametri Appendix A. Wires, Sizes and AWG diameters
 Appendice A. Conduttori elettrici, sezioni e diametri Appendix A. Wires, Sizes and AWG diameters A.1 Misura dei conduttori elettrici, sezioni e diametri AWG and kcmil wires sizes measurement L America
Appendice A. Conduttori elettrici, sezioni e diametri Appendix A. Wires, Sizes and AWG diameters A.1 Misura dei conduttori elettrici, sezioni e diametri AWG and kcmil wires sizes measurement L America
Mission IGA. associazione senza scopo di lucro fondata il 13 aprile 2006 riconoscimento giuridico FVG 4 agosto 2008
 Mission IGA promuovere la ricerca della diversità genetica comprendere le basi genetiche dei caratteri complessi sviluppare algoritmi per l analisi dei sistemi biologici promuovere la cultura scientifica
Mission IGA promuovere la ricerca della diversità genetica comprendere le basi genetiche dei caratteri complessi sviluppare algoritmi per l analisi dei sistemi biologici promuovere la cultura scientifica
