Applicazioni biotecnologiche in systems biology
|
|
|
- Gerardo Mazzoni
- 8 anni fa
- Visualizzazioni
Transcript
1 Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013
2 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore delle idee) Tel:
3 Systems biology
4 Principali tecnologie di sequenziamento e controllo qualità Lezione #2 Dr. Marco Galardini AA 2012/2013
5 Sequenziamento del genoma Genoma: intera sequenza nucleotidica di un organismo Sequenziamento: determinazione di una sequenza nucleotidica Le tecniche di sequenziamento Generazione 0 Sequenziamento chimico Generazione 1 Dye-terminator Generazione 2 NGS con pre-amplificazione Generazione 3 NGS su singola molecola NGS: Next Generation Sequencing
6 Sequenziamento del genoma Le tecniche di sequenziamento Generazione 0 - Maxam & Gilbert Generazione 1 - Sanger Generazione 2 Pyrosequencing (454), Solexa-Illumina, Solid, Ion Torrent Generazione 3 PACBIO RS, Nanopore
7 Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil Green and Brent Ewing Viene pubblicato il softwere Phred per l analisi di sequenze di DNA 1986 Leeroy E.Hoods Prima macchina semiautomatica per il sequenziamento del DNA 1996 Pål Nyrén Nascita del pyrosequencing 2000 Lynx Therapeutics Lancio sul mercato del MPSS e inizio del sequenziamento di nuova generazione (NGS)
8 Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA Quantità di dati nell unità di tempo 1998 Phil Green and Brent Ewing Viene pubblicato il softwere Phred per l analisi di sequenze di DNA 1986 Leeroy E.Hoods Prima macchina semiautomatica per il sequenziamento del DNA Qualità e controllo dei dati ottenuti Riduzione del costo 1996 Pål Nyrén Nascita del pyrosequencing 2000 Lynx Therapeutics Lancio sul mercato del MPSS e inizio del sequenziamento di nuova generazione (NGS)
9 Automatismi di sequenziamento?
10 Automatismi di sequenziamento Maggiore automazione, industrializzazione del sequenziamento Concentrazione di sequenziatori presso centri genomici (economia di scala) Riduzione del numero di singoli sequenziatori presso laboratori di ricerca Utilizzo della genomica per applicazioni cliniche, farmaceutiche, industriali
11 Incremento della complessità Sanger sequencing Quantità di basi lette Nell unità di tempo Risorse per la lettura ed immagazzinamento delle sequenze NGS
12 Sequenziamento del genoma Le tecniche di sequenziamento Generazione 0 - Maxam & Gilbert Generazione 1 - Sanger Generazione 2 Pyrosequencing (454), Solexa-Illumina, Solid, Ion Torrent Generazione 3 PACBIO RS, Nanopore
13 Generazione 1 - Sanger ovvero l idea geniale dei dideossi!
14 Sanger sequencing: Cycle Sequencing Molecola segnale La bassa percentuale di ddntp fa sì passi del tempo prima che la sintesi venga interrotta dnalc
15 Sequenziamento manuale Di-deossi nucleotidi radioattivi Una lane per ogni nucleotide Esistono sequenziatori automatici che leggono automaticamente i gel di poliacrilamide
16 Sequenziamento automatico Di-deossi nucleotidi fluorescenti Un colore per ogni nucleotide Una singola lane per i 4 nucleotidi Separazione attraverso elettroforesi capillare Lettura nucleotidi tramite laser Macchinari multi-capillari per letture in parallelo
17 Cromatogramma Lunghezza dei frammenti raggiunta: fino a 1000 bp
18 Cromatogramma possibili errori Sovrapposizione fra i picchi Omonucleotidi Difficile separazione fra i picchi Sovrapposizione fra i picchi Necessario un approccio algoritmico per determinare la qualità dei cromatogrammi ed individuare la giusta sequenza
19 Generazione 2 NGS con pre-amplificazione Pyrosequencing (454) Solexa-Illumina Solid (Anche la tecnica Sanger prevede un amplificazione del dna prima del sequenziamento, con successiva ligazione di un primer)
20 NGS, seconda generazione
21 NGS, seconda generazione Viene commercializzata la prima macchina automatica per il pirosequenziamento Prima pubblicazione sul pirosequenziamento Primo sequenziamento automatico Primo sequenziamento semi-automatico (Sanger dye-terminator)
22 NGS, seconda generazione 454 pyrosequencing Viene commercializzata la prima macchina automatica per il pirosequenziamento Illumina (Solexa) Prima pubblicazione sul pirosequenziamento Primo sequenziamento automatico Primo sequenziamento semi-automatico (Sanger dye-terminator) SOLiD Ion semiconductor DNA nanoball Helioscope SMRT/RNAP
23 NGS, seconda generazione 454 pyrosequencing Viene commercializzata la prima macchina automatica per il pirosequenziamento Illumina (Solexa) Prima pubblicazione sul pirosequenziamento Primo sequenziamento automatico Primo sequenziamento semi-automatico (Sanger dye-terminator) SOLiD Ion semiconductor DNA nanoball Helioscope SMRT/RNAP
24 Il Pyrosequencing (o 454) La tecnologia 454 Da 400 a 700 basi per read Circa la metà rispetto al Sanger megabasi ogni 10 ore Più veloce del Sanger Costi medio alti Impossibilità di sequenziare correttamente più di 8 basi identiche consecutive (omonucleotidi)
25 Il Pyrosequencing (o 454) La tecnologia 454 Amplificazione tramite emulsion PCR (empcr) Multiple reazioni di PCR nella stessa provetta Sospensioni acquose in olio Frantumazione DNA Primer di PCR legati a microsfere Intrappolamento microsfere in micropiastra Una microsfera per pozzetto
26 Il Pyrosequencing (o 454) La tecnologia 454 Pirosequenziamento Inserimento di un nucleotide per volta Incorporazione di un nucleotide da parte della DNA polimerasi comporta lo sviluppo di luce Rilevazione da parte di una videocamera
27
28 Importante: Cosa succede quando aggiungo ATP?
29 Importante: Cosa succede quando aggiungo ATP? Al posto dell ATP si usa d-alpha-thio triphosphate (non utilizzata dalla luciferasi)
30 Il Solexa-Illumina La tecnologia Solexa, cioè sequencing on-a-chip Circa 100 basi per read 10 milioni di reads per spot Ridotti costi di sequenziamento Profondità di sequenziamento elevata Video Overview
31 Frammentazione + ligazione adattatori Annealing sul chip Amplificazione tramite bridge PCR
32 Amplificazione tramite bridge PCR Formazione di cluster densi e separati Ogni cluster contiene una singola sequenza
33 Aggiunta di nucleotidi fluorescenti Terminatori reversibili (il fluoroforo può essere clivato) Lettura di una base per volta, ma in parallelo su molti cluster
34 Lunghezza dei frammenti raggiunta: 100 basi (in pair-end)
35 Il SOliD Sequencing by ligation No DNA polimerasi Utilizzo dell enzima ligasi Reads di 150 bp massimo (generalmente 35 bp) Massima accuratezza Vengono utilizzati sonde con due basi Ogni base della sequenza viene «letta» due volte Costi bassi Pre-amplificazione con empcr su microsfere Ligazione delle microsfere su un vetrino
36 Sequencing by Ligation 1. Primers hybridize to the P1 adapter sequence on the templated beads. 2. A set of four fluorescently labeled di-base probes compete for ligation to the sequencing primer. Specificity of the di-base probe is achieved by interrogating every 1st and 2nd base in each ligation reaction. 3. Multiple cycles of ligation, detection and cleavage are performed with the number of cycles determining the eventual read length. 4. Following a series of ligation cycles, the extension product is removed and the template is reset with a primer complementary to the n-1 position for a second round of ligation cycles. Lunghezza dei frammenti raggiunta: basi
37 Video
38 Ion Torrent
39 Amplificazione tramite empcr su microsfere (intrappolate in micropozzetti) Utilizzo di normali dntp L incorporazione di un dntp rilascia un protone (H+) Rilevazione della differenza di voltaggio (ΔV) ΔV proporzionale al numero di nucleotidi incorporati
40 Lunghezza dei frammenti raggiunta: oltre 100 basi Previsto nuovo CMOS chip da 400 basi entro dicembre 2011
41 Generazione 3 Oxford Nanopore PacBio Single Molecule Real Time sequencing (SMRT) Sequenziamento a singola molecola di DNA Amplificazione iniziale tramite PCR non necessaria Riduzione di bias dovuti all amplificazione (amplificazione differenziale di alcune parti della sequenza)
42 Oxford Nanopore Tecnologia ancora molto sperimentale Vari metodi per determinare i singoli nucleotidi
43 PacBio RS - SMRT Technology
44 Filmato SMRT Pozzetti con volume litri (zeptolitri) Aggiunta sequenziale di dntp marcati con fluorofori Determinazione incorporazione tramite ZMW (zero-mode waveguide)
45 Lunghezza dei frammenti raggiunta: bp (esperimenti con circa 2.5 kb)
46
47 Qualità dei sequenziamenti Dalla reazione alla sequenza
48 Qualità dei sequenziamenti Sequence:??????????????????????????????????
49 Qualità dei sequenziamenti Come è possibile risalire alla sequenza di DNA sequenziata? Necessità di trovare un modo univoco per l interpretazione dei cromatogrammi. E possibile ripetere il processo per milioni di volte? Necessità di sviluppare algoritmi in grado di assegnare ad ogni picco una determinata base in modo automatico.
50 Qualità dei sequenziamenti
51 Qualità dei sequenziamenti G
52 Qualità dei sequenziamenti G C
53 Qualità dei sequenziamenti G C A
54 Qualità dei sequenziamenti G C A G
55 Qualità dei sequenziamenti G C A G C G A Durante l elettroforesi capillare frammenti di lunghezza diversa possono non essere ben separati Lettura simultanea di basi diverse Man mano che il sequenziamento procede verso la fine del segmento di DNA, la qualità andrà a diminuire (anche all inizio) Una bassa qualità può significare una sequenza diversa da quella reale
56 Base-calling algorithm Algoritmo che, data una serie di picchi cromatografici, sia in grado di risalire alla corretta sequenza di DNA che li ha generati ed assegnare ad ogni base una certa «qualità» Phred E l algoritmo più utilizzato. Ad oggi la qualità delle sequenze di DNA viene espressa utilizzando il suo score
57 Base-calling algorithm Vengono presi in considerazione i principali parametri relativi alla forma ed all ampiezza dei picchi; in base a questi viene calcolato un certo valore di qualità relativo ad ogni base.
58 Base-calling algorithm Vengono presi in considerazione i principali parametri relativi alla forma ed all ampiezza dei picchi; in base a questi viene calcolato un certo valore di qualità relativo ad ogni base. La qualità così ottenuta è strettamente legata alla probabilità di ottenere quella determinata base in quel punto del cromatogramma Base A C Quality 40 30
59 Qualità e Probabilità Q = Qualità (Phred Score) P = Probabilità di errore Q = -10log 10 (P) P = 10 -Q/10 BASES A C T G ERROR QUALITY
60 Qualità e Probabilità Q = Qualità (Phred Score) P = Probabilità di errore Q = -10log 10 (P) P = 10 -Q/10 Ad esempio: una qualità pari a 20 si traduce in una probabilità di errore dell 1%, mentre una qualità pari a 30 si traduce in una probabilità dello 0.1% BASES A C T G ERROR 1% 0.1% 0.01% 0.001% QUALITY
61 Qualità e Probabilità BASES A C T G ERROR 1% 0.1% 0.01% 0.001% QUALITY Base scartata Qualità generalmente accettata Importante: Una probabilità d errore dell 1% può sembrare bassa Bisogna tenere conto che vengono sequenziate milioni di basi 1% = basi errate ogni Mb Queste basi errate possono essere confuse per mutazioni -> implicazioni cliniche gravi!
62 Dal sequenziamento al genoma Dalle reads alle sequenze (più o meno) complete
63 Tutte le tecniche di sequenziamento portano alla produzione di molte sequenze corte ( bp) Il genoma batterico più corto è circa 1Mb (500 volte la reads più lunga) Un gene batterico è lungo in media 300 bp Necessario unire insieme le varie reads per ottenere un genoma Trimming, Assembling, Scaffolding
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
DNA revolution. Microsatellites SNPs. Sequenziamento massivo (Next Generation Sequencing NGS)
 DNA revolution Microsatellites SNPs Sequenziamento massivo (Next Generation Sequencing NGS) NGS NGS NGS Non-Sanger-based high-throughput DNA sequencing technology Increasing the throughput Minimizing the
DNA revolution Microsatellites SNPs Sequenziamento massivo (Next Generation Sequencing NGS) NGS NGS NGS Non-Sanger-based high-throughput DNA sequencing technology Increasing the throughput Minimizing the
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Avanzamento dei sistemi di sequenziamento
 Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Tecniche di sequenziamento del DNA
 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #6 Dr. Marco Galardini AA 2012/2013 Gene regulation analysis Lezione #6 Dr. Marco Galardini AA 2012/2013 Regolazione genica Elementi molecolari e
Applicazioni biotecnologiche in systems biology Lezione #6 Dr. Marco Galardini AA 2012/2013 Gene regulation analysis Lezione #6 Dr. Marco Galardini AA 2012/2013 Regolazione genica Elementi molecolari e
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
La farmacogeneticanella gestione del paziente con tumore colorettale, l esperienza traslazionaleal CRO di Aviano
 SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
Un sistema così integrato, rende più semplice e preciso il trasferimento di dati dalla prima alla seconda fase del processo.
 CAD-CAM significa impiegare insieme sistemi di software per la progettazione assistita da computer e per la fabbricazione assistita da computer mediante macchinari a controllo numerico. Un sistema così
CAD-CAM significa impiegare insieme sistemi di software per la progettazione assistita da computer e per la fabbricazione assistita da computer mediante macchinari a controllo numerico. Un sistema così
Analisi qualitativa con Agilent BioAnalyzer. cdna, RNA Totale e Messaggero e small RNA
 Il Servizio di Biologia Molecolare si è di recente dotato di uno strumento per l analisi qualitativa e quantitativa degli acidi nucleici Agilent Bioanalyzer 2100 e ha implementato tale tecnologia nell
Il Servizio di Biologia Molecolare si è di recente dotato di uno strumento per l analisi qualitativa e quantitativa degli acidi nucleici Agilent Bioanalyzer 2100 e ha implementato tale tecnologia nell
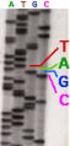
Immagine di una sequenza ben riuscita. Sequences troubleshooting
 Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Francesca Ceroni. Biotecnologie tradizionali. Biologia Sintetica. F. Ceroni 16/09/2010. Bressanone GNB 2010 1. 1) DNA ricombinante 2) PCR
 XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
Real Time PCR. La PCR Real Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico.
 eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
scaricato da www.sunhope.it LA TECNOLOGIA DEI MICROARRAYS
 LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
Analisi di sequenziamento degli acidi nucleici
 Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Librerie digitali. Video. Gestione di video. Caratteristiche dei video. Video. Metadati associati ai video. Metadati associati ai video
 Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
Bioinformatica: DNA e Algoritmi
 Bioinformatica: DNA e Algoritmi Alberto Policriti Dpt. of Mathematics and Informatics, University of Udine. Applied Genomics Institute Di cosa parleremo In generale Deniamo i termini: DNA & Algoritmi Tecnologie
Bioinformatica: DNA e Algoritmi Alberto Policriti Dpt. of Mathematics and Informatics, University of Udine. Applied Genomics Institute Di cosa parleremo In generale Deniamo i termini: DNA & Algoritmi Tecnologie
Inflazione e Produzione. In questa lezione cercheremo di rispondere a domande come queste:
 Inflazione e Produzione In questa lezione cercheremo di rispondere a domande come queste: Da cosa è determinata l Inflazione? Perché le autorità monetarie tendono a combatterla? Attraverso quali canali
Inflazione e Produzione In questa lezione cercheremo di rispondere a domande come queste: Da cosa è determinata l Inflazione? Perché le autorità monetarie tendono a combatterla? Attraverso quali canali
PCR. (Reazione a catena della polimerasi) ESTRAZIONE del DNA da gel di AGAROSIO. Corso di INGEGNERIA GENETICA, Prof. Renato Fani,
 PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di. DNA umano. 22-26 giugno 2009
 Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
SAFETY & MOVING. Miglioramento delle pratiche di efficienza produttiva. 6 maggio 2014. ing. Marco Lanciai
 SAFETY & MOVING Miglioramento delle pratiche di efficienza produttiva 6 maggio 2014 ing. Marco Lanciai Indice Assemblaggio Gestione dei magazzini Movimentazione di merci e materiali 2 Contesto storico
SAFETY & MOVING Miglioramento delle pratiche di efficienza produttiva 6 maggio 2014 ing. Marco Lanciai Indice Assemblaggio Gestione dei magazzini Movimentazione di merci e materiali 2 Contesto storico
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Calcolatori Elettronici A a.a. 2008/2009
 Calcolatori Elettronici A a.a. 2008/2009 PRESTAZIONI DEL CALCOLATORE Massimiliano Giacomin Due dimensioni Tempo di risposta (o tempo di esecuzione): il tempo totale impiegato per eseguire un task (include
Calcolatori Elettronici A a.a. 2008/2009 PRESTAZIONI DEL CALCOLATORE Massimiliano Giacomin Due dimensioni Tempo di risposta (o tempo di esecuzione): il tempo totale impiegato per eseguire un task (include
Che cosa e come valutano le prove di matematica e con quali risultati. nell A.S. 2008 2009
 Che cosa e come valutano le prove di matematica e con quali risultati nell A.S. 2008 2009 Presentazione a cura di Roberta Michelini Casalpusterlengo, 8 gennaio 2010 http://www.invalsi.it/esamidistato0809/
Che cosa e come valutano le prove di matematica e con quali risultati nell A.S. 2008 2009 Presentazione a cura di Roberta Michelini Casalpusterlengo, 8 gennaio 2010 http://www.invalsi.it/esamidistato0809/
Cultura generale. per tutti i produttori di ghiaccio. Qualità dell acqua / qualità del ghiaccio. www.krebastore.com
 Qualità dell acqua / qualità del ghiaccio Il ghiaccio, un potenziale concentratore di batteri Cultura generale È evidente che un cubetto o una scaglia di ghiaccio concentra al suo interno tutti gli odori
Qualità dell acqua / qualità del ghiaccio Il ghiaccio, un potenziale concentratore di batteri Cultura generale È evidente che un cubetto o una scaglia di ghiaccio concentra al suo interno tutti gli odori
Esperienza 3: clonaggio di un gene in un plasmide
 Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Informatica - A.A. 2010/11
 Ripasso lezione precedente Facoltà di Medicina Veterinaria Corso di laurea in Tutela e benessere animale Corso Integrato: Matematica, Statistica e Informatica Modulo: Informatica Esercizio: Convertire
Ripasso lezione precedente Facoltà di Medicina Veterinaria Corso di laurea in Tutela e benessere animale Corso Integrato: Matematica, Statistica e Informatica Modulo: Informatica Esercizio: Convertire
GESTIONE RICERCA & SVILUPPO LEZIONE 5. Angelo BONOMI
 GESTIONE RICERCA & SVILUPPO LEZIONE 5 Angelo BONOMI INDUSTRIALIZZAZIONE DELL INNOVAZIONE TECNOLOGICA RAPPRESENTA LA FASE DI TRANSIZIONE TRA R&S E PRODUZIONE ED E CARATTERIZZATA DAL LAVORO DI LbyD PER LO
GESTIONE RICERCA & SVILUPPO LEZIONE 5 Angelo BONOMI INDUSTRIALIZZAZIONE DELL INNOVAZIONE TECNOLOGICA RAPPRESENTA LA FASE DI TRANSIZIONE TRA R&S E PRODUZIONE ED E CARATTERIZZATA DAL LAVORO DI LbyD PER LO
METODI ALTERNATIVI. PCR digitale per la rilevazione e quantificazione. assoluta del DNA
 METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
Perché il logaritmo è così importante?
 Esempio 1. Perché il logaritmo è così importante? (concentrazione di ioni di idrogeno in una soluzione, il ph) Un sistema solido o liquido, costituito da due o più componenti, (sale disciolto nell'acqua),
Esempio 1. Perché il logaritmo è così importante? (concentrazione di ioni di idrogeno in una soluzione, il ph) Un sistema solido o liquido, costituito da due o più componenti, (sale disciolto nell'acqua),
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY.
 Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Quantificazione di. legionella pneumophila. mediante metodo biomolecolare
 Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
Genomica Servizio Sequenziamento DNA
 Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Esempi di algoritmi. Lezione III
 Esempi di algoritmi Lezione III Scopo della lezione Implementare da zero algoritmi di media complessità. Verificare la correttezza di un algoritmo eseguendolo a mano. Imparare a valutare le prestazioni
Esempi di algoritmi Lezione III Scopo della lezione Implementare da zero algoritmi di media complessità. Verificare la correttezza di un algoritmo eseguendolo a mano. Imparare a valutare le prestazioni
GUIDA AL CALCOLO DEI COSTI DELLE ATTIVITA DI RICERCA DOCUMENTALE
 GUIDA AL CALCOLO DEI COSTI DELLE ATTIVITA DI RICERCA DOCUMENTALE L applicazione elaborata da Nordest Informatica e disponibile all interno del sito è finalizzata a fornirvi un ipotesi dell impatto economico
GUIDA AL CALCOLO DEI COSTI DELLE ATTIVITA DI RICERCA DOCUMENTALE L applicazione elaborata da Nordest Informatica e disponibile all interno del sito è finalizzata a fornirvi un ipotesi dell impatto economico
CALCOLATORI ELETTRONICI A cura di Luca Orrù. Lezione n.7. Il moltiplicatore binario e il ciclo di base di una CPU
 Lezione n.7 Il moltiplicatore binario e il ciclo di base di una CPU 1 SOMMARIO Architettura del moltiplicatore Architettura di base di una CPU Ciclo principale di base di una CPU Riprendiamo l analisi
Lezione n.7 Il moltiplicatore binario e il ciclo di base di una CPU 1 SOMMARIO Architettura del moltiplicatore Architettura di base di una CPU Ciclo principale di base di una CPU Riprendiamo l analisi
Elaborazione di segnali biologici
 Modulo 4 Elaborazione di segnali biologici Bioingegneria per fisioterapisti Univ.degli Studi di Siena Laurea Univ. in Fisioterapia Ing. A. Rossi Sistemi acquisizione dati Conversione da segnale Analogico
Modulo 4 Elaborazione di segnali biologici Bioingegneria per fisioterapisti Univ.degli Studi di Siena Laurea Univ. in Fisioterapia Ing. A. Rossi Sistemi acquisizione dati Conversione da segnale Analogico
manifatturiera e per i servizi
 CAPITOLO 7 Tecnologie per la produzione manifatturiera e per i servizi Agenda Tecnologia e core technology Processi core ed ausiliari Tecnologia e struttura organizzativa Tecnologia core manifatturiera
CAPITOLO 7 Tecnologie per la produzione manifatturiera e per i servizi Agenda Tecnologia e core technology Processi core ed ausiliari Tecnologia e struttura organizzativa Tecnologia core manifatturiera
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
CAPACITÀ DI PROCESSO (PROCESS CAPABILITY)
 CICLO DI LEZIONI per Progetto e Gestione della Qualità Facoltà di Ingegneria CAPACITÀ DI PROCESSO (PROCESS CAPABILITY) Carlo Noè Università Carlo Cattaneo e-mail: cnoe@liuc.it 1 CAPACITÀ DI PROCESSO Il
CICLO DI LEZIONI per Progetto e Gestione della Qualità Facoltà di Ingegneria CAPACITÀ DI PROCESSO (PROCESS CAPABILITY) Carlo Noè Università Carlo Cattaneo e-mail: cnoe@liuc.it 1 CAPACITÀ DI PROCESSO Il
Scheduling della CPU. Sistemi multiprocessori e real time Metodi di valutazione Esempi: Solaris 2 Windows 2000 Linux
 Scheduling della CPU Sistemi multiprocessori e real time Metodi di valutazione Esempi: Solaris 2 Windows 2000 Linux Sistemi multiprocessori Fin qui si sono trattati i problemi di scheduling su singola
Scheduling della CPU Sistemi multiprocessori e real time Metodi di valutazione Esempi: Solaris 2 Windows 2000 Linux Sistemi multiprocessori Fin qui si sono trattati i problemi di scheduling su singola
Una sperimentazione. Probabilità. Una previsione. Calcolo delle probabilità. Nonostante ciò, è possibile dire qualcosa.
 Una sperimentazione Probabilità Si sta sperimentando l efficacia di un nuovo farmaco per il morbo di Parkinson. Duemila pazienti partecipano alla sperimentazione: metà di essi vengono trattati con il nuovo
Una sperimentazione Probabilità Si sta sperimentando l efficacia di un nuovo farmaco per il morbo di Parkinson. Duemila pazienti partecipano alla sperimentazione: metà di essi vengono trattati con il nuovo
Modellazione dinamica e strumenti per la valutazione sperimentale del comportamento della via EGFR-IGF1R per la linea cellulare H1650
 Università deglistudidiperugia FacoltàdiIngegneria Tesi di Laurea Magistrale in Ingegneria Informatica e dell Automazione Anno Accademico 2011/2012 Modellazione dinamica e strumenti per la valutazione
Università deglistudidiperugia FacoltàdiIngegneria Tesi di Laurea Magistrale in Ingegneria Informatica e dell Automazione Anno Accademico 2011/2012 Modellazione dinamica e strumenti per la valutazione
HISTO SPOT Sistema SSO
 HISTO SPOT Sistema SSO per la tipizzazione HLA Kit HISTO SPOT Strumento MR.SPOT Software HISTO MATCH Sistema completo certificato per uso IVD Le specifiche di HISTO SPOT Principio: blot SSO reverse miniaturizzato
HISTO SPOT Sistema SSO per la tipizzazione HLA Kit HISTO SPOT Strumento MR.SPOT Software HISTO MATCH Sistema completo certificato per uso IVD Le specifiche di HISTO SPOT Principio: blot SSO reverse miniaturizzato
MODELLO PER LO SVILUPPO DEL PRODOTTO
 MODELLO PER LO SVILUPPO DEL PRODOTTO 34 Funzioni aziendali coinvolte nello sviluppo prodotto LE PRINCIPALI FUNZIONI AZIENDALI PROTAGONISTE NELLO SVIPUPPO PRODOTTI SONO: Progettazione e sviluppo prodotto
MODELLO PER LO SVILUPPO DEL PRODOTTO 34 Funzioni aziendali coinvolte nello sviluppo prodotto LE PRINCIPALI FUNZIONI AZIENDALI PROTAGONISTE NELLO SVIPUPPO PRODOTTI SONO: Progettazione e sviluppo prodotto
Introduzione. Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD... 6
 Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
Appunti di Calcolatori Elettronici Esecuzione di istruzioni in parallelo Introduzione... 1 Classificazione di Flynn... 2 Macchine a pipeline... 3 Macchine vettoriali e Array Processor... 4 Macchine MIMD...
GLI APPARATI PER L INTERCONNESSIONE DI RETI LOCALI 1. Il Repeater 2. L Hub 2. Il Bridge 4. Lo Switch 4. Router 6
 GLI APPARATI PER L INTERCONNESSIONE DI RETI LOCALI 1 Il Repeater 2 L Hub 2 Il Bridge 4 Lo Switch 4 Router 6 Gli apparati per l interconnessione di reti locali Distinguiamo i seguenti tipi di apparati:
GLI APPARATI PER L INTERCONNESSIONE DI RETI LOCALI 1 Il Repeater 2 L Hub 2 Il Bridge 4 Lo Switch 4 Router 6 Gli apparati per l interconnessione di reti locali Distinguiamo i seguenti tipi di apparati:
gestione e modifica di immagini fotografiche digitali
 gestione e modifica di immagini fotografiche digitali il colore e le immagini la gestione delle immagini Il computer è in grado di gestire le immagini in formato digitale. Gestire vuol dire acquisirle,
gestione e modifica di immagini fotografiche digitali il colore e le immagini la gestione delle immagini Il computer è in grado di gestire le immagini in formato digitale. Gestire vuol dire acquisirle,
Dispositivi di rete. Ripetitori. Hub
 Ripetitori Dispositivi di rete I ripetitori aumentano la distanza che può essere ragginta dai dispositivi Ethernet per trasmettere dati l'uno rispetto all'altro. Le distanze coperte dai cavi sono limitate
Ripetitori Dispositivi di rete I ripetitori aumentano la distanza che può essere ragginta dai dispositivi Ethernet per trasmettere dati l'uno rispetto all'altro. Le distanze coperte dai cavi sono limitate
LA CORRENTE ELETTRICA CONTINUA
 LA CORRENTE ELETTRICA CONTINUA (Fenomeno, indipendente dal tempo, che si osserva nei corpi conduttori quando le cariche elettriche fluiscono in essi.) Un conduttore metallico è in equilibrio elettrostatico
LA CORRENTE ELETTRICA CONTINUA (Fenomeno, indipendente dal tempo, che si osserva nei corpi conduttori quando le cariche elettriche fluiscono in essi.) Un conduttore metallico è in equilibrio elettrostatico
La memoria centrale (RAM)
 La memoria centrale (RAM) Mantiene al proprio interno i dati e le istruzioni dei programmi in esecuzione Memoria ad accesso casuale Tecnologia elettronica: Veloce ma volatile e costosa Due eccezioni R.O.M.
La memoria centrale (RAM) Mantiene al proprio interno i dati e le istruzioni dei programmi in esecuzione Memoria ad accesso casuale Tecnologia elettronica: Veloce ma volatile e costosa Due eccezioni R.O.M.
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Tutorial 3DRoom. 3DRoom
 Il presente paragrafo tratta il rilievo di interni ed esterni eseguito con. L utilizzo del software è molto semplice ed immediato. Dopo aver fatto uno schizzo del vano si passa all inserimento delle diagonali
Il presente paragrafo tratta il rilievo di interni ed esterni eseguito con. L utilizzo del software è molto semplice ed immediato. Dopo aver fatto uno schizzo del vano si passa all inserimento delle diagonali
Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health. NGS: tutti i segreti del laboratorio
 Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health NGS: tutti i segreti del laboratorio Francesco Cerutti S.S. Genetica e Immunobiochimica IstitutoZooprofilatticoSperimentaledel
Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health NGS: tutti i segreti del laboratorio Francesco Cerutti S.S. Genetica e Immunobiochimica IstitutoZooprofilatticoSperimentaledel
(a cura di Francesca Godioli)
 lezione n. 12 (a cura di Francesca Godioli) Ad ogni categoria della variabile qualitativa si può assegnare un valore numerico che viene chiamato SCORE. Passare dalla variabile qualitativa X2 a dei valori
lezione n. 12 (a cura di Francesca Godioli) Ad ogni categoria della variabile qualitativa si può assegnare un valore numerico che viene chiamato SCORE. Passare dalla variabile qualitativa X2 a dei valori
MISURARE IL COSTO DELLA VITA
 Università degli Studi di Macerata Dipartimento di Scienze politiche, della Comunicazione e delle Relazioni internazionali ECONOMIA POLITICA MODULO TEORIA A.A. 2013/2014 MISURARE IL COSTO DELLA VITA Fabio
Università degli Studi di Macerata Dipartimento di Scienze politiche, della Comunicazione e delle Relazioni internazionali ECONOMIA POLITICA MODULO TEORIA A.A. 2013/2014 MISURARE IL COSTO DELLA VITA Fabio
Le obbligazioni: misure di rendimento Tassi d interesse, elementi di valutazione e rischio delle attività finanziarie
 Le obbligazioni: misure di rendimento Tassi d interesse, elementi di valutazione e rischio delle attività finanziarie Economia degli Intermediari Finanziari 29 aprile 2009 A.A. 2008-2009 Agenda 1. Il calcolo
Le obbligazioni: misure di rendimento Tassi d interesse, elementi di valutazione e rischio delle attività finanziarie Economia degli Intermediari Finanziari 29 aprile 2009 A.A. 2008-2009 Agenda 1. Il calcolo
Protocollo Crime Scene Investigation
 Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
Analisi del trivai point
 Analisi del trivai point Strategia di controllo: il valore trivai (punto di trivalenza) Questa strategia di controllo, basata sul valore trivai, è generalmente usata se è disponibile come generatore ausiliario
Analisi del trivai point Strategia di controllo: il valore trivai (punto di trivalenza) Questa strategia di controllo, basata sul valore trivai, è generalmente usata se è disponibile come generatore ausiliario
Sistema Informativo Geografico:
 Sistemi Informativi Geografici Sistema Informativo Geografico: È un sistema informativo che tratta informazioni spaziali georeferenziate, ne consente la gestione e l'analisi. Informazioni spaziali: dati
Sistemi Informativi Geografici Sistema Informativo Geografico: È un sistema informativo che tratta informazioni spaziali georeferenziate, ne consente la gestione e l'analisi. Informazioni spaziali: dati
OSSERVAZIONI TEORICHE Lezione n. 4
 OSSERVAZIONI TEORICHE Lezione n. 4 Finalità: Sistematizzare concetti e definizioni. Verificare l apprendimento. Metodo: Lettura delle OSSERVAZIONI e risoluzione della scheda di verifica delle conoscenze
OSSERVAZIONI TEORICHE Lezione n. 4 Finalità: Sistematizzare concetti e definizioni. Verificare l apprendimento. Metodo: Lettura delle OSSERVAZIONI e risoluzione della scheda di verifica delle conoscenze
Analisi della varianza (anova) a due vie
 Analisi della varianza (anova) a due vie Andrea Onofri 27 marzo 2014 Indice 1 Il concetto di interazione 1 2 Tipi di interazione 2 3 Descrizione del caso studio 3 4 Analisi dei dati 4 Sommario Scopo di
Analisi della varianza (anova) a due vie Andrea Onofri 27 marzo 2014 Indice 1 Il concetto di interazione 1 2 Tipi di interazione 2 3 Descrizione del caso studio 3 4 Analisi dei dati 4 Sommario Scopo di
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL RISCHIO D IMPRESA ED IL RISCHIO FINANZIARIO. LA RELAZIONE RISCHIO-RENDIMENTO ED IL COSTO DEL CAPITALE.
 IL RISCHIO D IMPRESA ED IL RISCHIO FINANZIARIO. LA RELAZIONE RISCHIO-RENDIMENTO ED IL COSTO DEL CAPITALE. Lezione 5 Castellanza, 17 Ottobre 2007 2 Summary Il costo del capitale La relazione rischio/rendimento
IL RISCHIO D IMPRESA ED IL RISCHIO FINANZIARIO. LA RELAZIONE RISCHIO-RENDIMENTO ED IL COSTO DEL CAPITALE. Lezione 5 Castellanza, 17 Ottobre 2007 2 Summary Il costo del capitale La relazione rischio/rendimento
Il SAC, sistema di controllo nella divisione cellulare
 Il SAC, sistema di controllo nella divisione cellulare IFOM per la scuola Lo Studente Ricercatore 2011 Villani Yuri Liceo Scientifico Tecnologico Chimico-Biologico IIS A. Maserati - Voghera(PV) Gruppo:
Il SAC, sistema di controllo nella divisione cellulare IFOM per la scuola Lo Studente Ricercatore 2011 Villani Yuri Liceo Scientifico Tecnologico Chimico-Biologico IIS A. Maserati - Voghera(PV) Gruppo:
Acidi e basi. HCl H + + Cl - (acido cloridrico) NaOH Na + + OH - (idrossido di sodio; soda caustica)
 Acidi e basi Per capire che cosa sono un acido e una base dal punto di vista chimico, bisogna riferirsi ad alcune proprietà chimiche dell'acqua. L'acqua, sia solida (ghiaccio), liquida o gassosa (vapore
Acidi e basi Per capire che cosa sono un acido e una base dal punto di vista chimico, bisogna riferirsi ad alcune proprietà chimiche dell'acqua. L'acqua, sia solida (ghiaccio), liquida o gassosa (vapore
Tirocinio per la Laurea Triennale
 Tirocinio per la Laurea Triennale Studente: Filippo Dattola Studio di un prototipo di infrastruttura virtuale di analisi parallela interattiva L'obiettivo del tirocinio è stato quello di studiare un prototipo
Tirocinio per la Laurea Triennale Studente: Filippo Dattola Studio di un prototipo di infrastruttura virtuale di analisi parallela interattiva L'obiettivo del tirocinio è stato quello di studiare un prototipo
Esame di INFORMATICA
 Università di L Aquila Facoltà di Biotecnologie Esame di INFORMATICA Lezione 4 MACCHINA DI VON NEUMANN Anni 40 i dati e i programmi che descrivono come elaborare i dati possono essere codificati nello
Università di L Aquila Facoltà di Biotecnologie Esame di INFORMATICA Lezione 4 MACCHINA DI VON NEUMANN Anni 40 i dati e i programmi che descrivono come elaborare i dati possono essere codificati nello
CAPITOLO 11 Innovazione cam i amen o
 CAPITOLO 11 Innovazione e cambiamento Agenda Ruolo strategico del cambiamento Cambiamento efficace Cambiamento tecnologico Cambiamento di prodotti e servizi i Cambiamento strategico e strutturale Cambiamento
CAPITOLO 11 Innovazione e cambiamento Agenda Ruolo strategico del cambiamento Cambiamento efficace Cambiamento tecnologico Cambiamento di prodotti e servizi i Cambiamento strategico e strutturale Cambiamento
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO DNA chiavi in mano. Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi
 PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
GestVetrine 1.1 versione novembre 2014
 GestVetrine 1.1 versione novembre 2014 0 GestVetrine 1.1 GestVetrine 1.0 Indice Indice pag.1 1. Finalità pag.2 2. Gestione screen pag.2 3. Come Accedere pag.3 4. Come Creare un negozio pag.4 5. Come Creare
GestVetrine 1.1 versione novembre 2014 0 GestVetrine 1.1 GestVetrine 1.0 Indice Indice pag.1 1. Finalità pag.2 2. Gestione screen pag.2 3. Come Accedere pag.3 4. Come Creare un negozio pag.4 5. Come Creare
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Analisi e diagramma di Pareto
 Analisi e diagramma di Pareto L'analisi di Pareto è una metodologia statistica utilizzata per individuare i problemi più rilevanti nella situazione in esame e quindi le priorità di intervento. L'obiettivo
Analisi e diagramma di Pareto L'analisi di Pareto è una metodologia statistica utilizzata per individuare i problemi più rilevanti nella situazione in esame e quindi le priorità di intervento. L'obiettivo
matematica probabilmente
 IS science centre immaginario scientifico Laboratorio dell'immaginario Scientifico - Trieste tel. 040224424 - fax 040224439 - e-mail: lis@lis.trieste.it - www.immaginarioscientifico.it indice Altezze e
IS science centre immaginario scientifico Laboratorio dell'immaginario Scientifico - Trieste tel. 040224424 - fax 040224439 - e-mail: lis@lis.trieste.it - www.immaginarioscientifico.it indice Altezze e
(Esercizi Tratti da Temi d esame degli ordinamenti precedenti)
 (Esercizi Tratti da Temi d esame degli ordinamenti precedenti) Esercizio 1 L'agenzia viaggi GV - Grandi Viaggi vi commissiona l'implementazione della funzione AssegnaVolo. Tale funzione riceve due liste
(Esercizi Tratti da Temi d esame degli ordinamenti precedenti) Esercizio 1 L'agenzia viaggi GV - Grandi Viaggi vi commissiona l'implementazione della funzione AssegnaVolo. Tale funzione riceve due liste
Attualmente esistono sul mercato videoendoscopi sempre più sofisticati con una buona risoluzione di immagine ed estremamente portatili.
 LA MISURA 3D, UN METODO AFFIDABILE PER VALUTARE LE INDICAZIONI IN UN ISPEZIONE VISIVA F. Vullo GE Measurement & Control Solutions, Via Cardano 2, 20864 Agrate Brianza (MB) Tel: +39 0396561403 Cell: +39
LA MISURA 3D, UN METODO AFFIDABILE PER VALUTARE LE INDICAZIONI IN UN ISPEZIONE VISIVA F. Vullo GE Measurement & Control Solutions, Via Cardano 2, 20864 Agrate Brianza (MB) Tel: +39 0396561403 Cell: +39
