Tecniche di biologia molecolare. Utilizzi delle endonucleasi e polimerasi
|
|
|
- Alfonso Deluca
- 6 anni fa
- Visualizzazioni
Transcript
1 Tecniche di biologia molecolare Utilizzi delle endonucleasi e polimerasi
2 4 PRINCIPALI TIPI DI ENZIMI AGISCONO SUL DNA: - NUCLEASI (idrolisi del legame fosfodiestere in oligonucleotidi) - LIGASI (formazione del legame fosfodiestere fra due oligonucleotidi) - TOPOISOMERASI (GIRASI, ELICASI) (variazione di superavvolgimenti) - POLIMERASI (polimerizzazione mediante la condensazione di nucleotidi)
3 Endonucleasi di restrizione batteriche Sito di taglio palindromico 5 GATATC 3 3 CTATAG 5 EcoRV (E. coli Restrict. Endonucl. V) Gli ER sono proteine omodimeriche, simmetriche che riconoscono un sequenza bersaglio simmetrica (sito palindromico), e tagliano entrambe i filamenti di DNA Inseriscono anse nel solco maggiore e scorrono lungo il doppio filamento di DNA fermandosi in corrispondenza al sito palindromico (riconoscono uno specifico pattern di basi). L interazione causa mutamenti conformazionali nell'enzima e nel DNA, che si piega - l'enzima richiede Mg ++, necessario per la sua attività; Ci sono tre tipi di ER: Tipo I Tipo II aspecifici; specifici per sequenze palindromiche Tipo III specifici e richiedono l idrolisi di ATP
4 le endonucleasi di restrizione tagliano entrambe i filamenti della molecola di DNA alcune tagliano in modo netto (BLUNT end) (es. Ecor V, SmaI) altre tagliano in modo sfilacciato (STICKY end) (es. BamHI, KpnI) Sito di taglio palindromico taglio netto (blunt) tagli sfilacciati (sticky)
5 Gli ER sono utilizzati per tagliare frammenti oligonucleotidici con precisione che poi possone essere inseriti in vettori per esempio i plasmidi trattati con lo stesso ER. Questi frammenti possono essere sequenze codificanti (es. geni senza introni) per farli amplificare e/o esprimere. Possono anche essere sequenziati Il plasmide (vettore) è trasfettato/trasformato in una cellula ospite per permettere il clonaggio molecolare dei frammenti (produzione multipla di copie identiche = amplificazione) Fragmentation: rottura controllata del filamento di DNA Ligation: inserimento mirato nel vettore Transfection: inserimento del vettore nella cellula Selection: selezione delle cellule correttamente trasformate
6 l inserimento del plasmide nel vettore è un processo stocastico può risultare in diversi prodotti, solo uno dei quali è utilizzabile è quindi necessario includere nella preparazione del plasmide caratteristiche che permettono di isolare il prodotto desiderato
7 + - Electroporation
8 Le DNA ligasi Enzimi che congiungono catene di DNA - catalizzano la formazione di un legame fosofodiestere fra un ossidrile al 3' di un frammento con il fosfato sul 5' di un altro. DNA1 3'OH O ligasi - + O-P-O 5' DNA2 O- ATP (NAD+) PPi (AMP) O DNA1 3' O-P-O 5' DNA2 O - Agiscono su dsdna, chiudendo rotture su una delle due catene. ATP è usato come attivatore dagli eucarioti, NAD+ dai batteri. Si forma un intermedio nel quale una unità di AMP si lega al fosfato 5' di un frammento e lo attiva per la formazione del legame con OH sul 3 dell altro frammento. ligasi + ATP NH 2 PPi O - ligasi + O-P-O 5' DNA2 O- NH AM- P fosfoammide DNA1 3' O-P-O 5' DNA2 O O - DNA1 3'OH AM- P O -P-O 5' DNA2 O -
9 le DNA topoisomerasi Tipo I - rilassano il DNA superavvolto. Tagliando un solo filamento, e permettendo lo srotolamento dell altro e poi lo ricongiungono. Riducono localmente lo strain in tratti di doppia elica sovra- o sottoavvolti Tipo II (girasi) tagliano entrambe i filamenti e permettono il passaggio di un altro tratto della doppia elica attraverso il taglio. Un processo che richiede l idrolisi di ATP. Per entrambe i tipi, durante il taglio si forma un legame fosfoesterico con una tirosina enzimatica, ed è liberato l OH 5, che poi attacca la fosfotirosina dopo lo srotolamento. Le topoisomerasi e girasi sono necessarie durante la replicazione o trascrizione del DNA, per evitare crisi topologiche a monte e/o a vale del DNA localmente disavvolto.
10 DNA polimerasi e RNA polimerasi le DNA polimerasi richiedono un FILAMENTO STAMPO, a catena singola di DNA, sul quale agire, e sintetizza una catena esattamente complementare allo stampo (appaiamento Watson-Crick corretti fra la base entrante e quella dello stampo dntp richiedono anche una catena PRIMER (che può essere DNA o RNA) con un terminale 3'-OH libero sulla quale innestare il primo dntp. Il primer si lega alla catena stampo al sito di origine della sintesi. Le NA polimerasi raramente inserisce basi male appaiate (dominio 5 3 polimerasi molto fedele) e controlla l'appaiamento eliminando residui con basi appaiate incorrettamente (dominio 3 5 esonucleasico). La funzione 5'3 esonucleasi serve per rimuovere la sequenza primer alla fine della sintesi. DNA-dependent DNA polimerase (replicazione) richiedono primer DNA-dependent RNA polymerase (trascrizione) non richiedono primer. Non così precise RNA-dependent DNA polimerase (trascrittasi inverse) richiedono primer
11 La Trascrittasi Inversa e le librerie di cdna Il cdna è una copia retro-trascritta di un mrna. In effetti rappresenta solo la pare codificane di un gene (privo di sequenze promotrici o di introni) Per produrla si sfrutta una Trascrittasi inversa e le caratteristiche del mrna La trascrittasi è un RNA-dependent DNA polymerase e quindi necessita di un primer Si sfrutta la coda di poly(a) al terminale 3, ibridizzando con un primer poly(t) Si produce un ibrido DNA/RNA, e l RNA e rimosso con RNAsi (una nucleasi selettiva per RNA) Adesso è possibile utilizzare una DNA-dependent DNA-polymerase e primer poly(da) per sintetizzare l altro filamento. Eventualmente si può amplificare il cdna così prodotto mediante la tecnica del PCR Siccome tutti gli mrna estratti da una cellula hanno la coda poly(a) si ottiene una LIBRERIA genica. Questa può essere subclonata in batteri e sequenziata per ottenere i cosiddetti «Expressed Sequence Tag» (EST)
12 Molecular cloning insieme di metodi utilizzati per assemblare DNA ricombinante e dirigere la sua replicazione in una cellula ospite, inserendo nella cellula un vettore molecolare (plasmide, virale) che contiene il DNA d interesse. L espansione clonale della cellula permette: 1) l amplificazione del DNA (che può essere recuperato) ; 2) l espressione della proteine codificata dal DNA. Metodo convenzionale: 1) - estrazione di DNA genomico - preparazione di cdna da RNA - sintesi di un gene artificiale 2) Frammentazione con endonucleasi sprecifiche selective amplification 3) Amplificazione selettiva (es. con PCR) fragment isolate cellular DNA
13 4) Scelta della combinazione vettore/cellula ospite. Il vettore deve contenere: - un origine di replicazione (Ori) - uno o più siti di restrizione per endonucleasi specifiche dove introdurre il frammento - un gene marker che permette di selezionare le cellule ospite (gene essenziale) Multiple restriction (cloning) sites (MCS) marker reporter - un gene reporter per lo screening delle cellule trasfettate (es. effetto cromogenico) choose vector Marker: Il gene ampr codifica per l enzima -latamasi che idrolizza antibiotici -lattamici (ampicillina). Fornische resistenza ed è essenziale per crescere in terreno con ampicillina Reporter: Il gene lacz codifica per l enzima -galattosidasi che scinde legami glicosidici, es. per idrolizzare il lattosio (e che è inattivato nel batterio ospite). Usando come substrato X- gal, produce colore.
14 5) Inserimento del frammento nel vettore (un plasmide o cromosoma artificiale), utilizzando apposite endonucleasi e ligasi per es. inserire all interno del gene lacz 6) Introduzione del vettore nella cellula ospite (spesso E. coli) resa competente Trasfezione (cellule animali); trasformazione (batteri, lievito); trasduzione (vettore virale) 7) Screening e selezione delle cellule ospiti correttamente trasformate (solo se trasformate sono resistenti a ampicillina, solo se ricombinanti non si colorano) 8) Espansione clonale della cellula ospite (amplificazione) ed utilizzo
15 Restriction mapping: l'utilizzo di ER con diverse specificità permette di mappare un frammento di DNA, cioè di generare una serie caratteristica di frammenti, che risulta in un pattern elettroforetico tipico. Nella medicina forense, il DNA proveniente da diversi individui, per la presenza o assenza di siti di taglio diversi, da luogo a mappe uniche e distinguibili. 1) Isolare un frammento di DNA usando l ER EcoRI (es. Frammento di 2 kilobasi) 2) Clonare in plasmide (es. pbluescript, 3.0 kbasi, MCS per EcoRI e BamH1). + scarr/rflp_animation.gif 3) Trattare il plasmide con l altro ER (BamH1) - ci sono due possibilità: - l inserto non contiene un sito BamH1 solo una banda in SDS PAGE (5 kb) - l inserto contiene un sito BamH1 due bande in SDS-PAGE (x e y KB)
16 4) Gel con inserto che contiene un sito BamH1: Un taglio avviene a livello del sito nel plasmide Bluescript, accanto a quello per EcoRI (terminale del frammento) e uno interno al frammento stesso. - si ottengono due tipi d informazione: a) che l inserto contiene un sito BamHI b) che il sito BamHI è a 1.4 kb dal terminale (posizione del sito) sito BamH1 mappato
17 Utilizzo del il restriction mapping nel sequenziamento di DNA L informazione nella mappa di restrizione è utile per molte tecniche che manipolano il DNA Per esempio permettere di tagliare un segmento di DNA grande in frammenti più piccoli, e poi selezionarli per il sequenziamento L ultima generazione di sequenziatori agisce infatti su frammenti piccoli ( basi) Il restriction mapping permette di confrontare i frammenti anche senza informazioni sulla sequenza. Per esempio, sapendo che due frammenti di 8 e 10 kb provengono dallo stesso pezzo di DNA cromosoma si vuole capire come si sovrappongono. Si determina quindi la mappa di restrizione con diversi ER Dalla distanza dei siti dai terminali noti si può dedurre la loro posizione e quindi dove i frammenti più grandi si sovrappongono. in questa maniera si possono scegliere i frammenti da sequenziare evitando costose duplicazioni
18 POLYMERASE CHAIN REACTION - PCR Una tecnica di biologia molecolare che consente la moltiplicazione (amplificazione) di frammenti di DNA o RNA dei quali si conoscano le sequenze nucleotidiche iniziali e terminali. RICHIEDE: DNA template con regione di DNA (target) da amplificare. Due primers che sono complementari ai terminali 3' dei filamenti senso and anti-senso. Taq polymerase o altra polimerasi con TM elevato e funzionamento ottimale a ca. 70 C. Soluzione tampone, che fornisce un ambiente chimico adatto a mantenerla stabile e attiva dntp Cationi divalenti, generalmente Mg 2+ o Mn 2+, e monovalente K +
19 POLYMERASE CHAIN REACTION - PCR
20 POLYMERASE CHAIN REACTION AMPLIFICAZIONE DI DNA GENOMICO o DI cdna Whole gene with introns Only coding DNA
21 POLYMERASE CHAIN REACTION PROTOCOLLO Doppio filamento di DNA denaturazione primer Sequenza desiderata annealing & polimerizzazione Sequenza neosintetizzata 4 denaturazione 5 6 annealing & polimerizzazione 7 1) Portare temp. a 95 C e denaturare il doppio filamento di DNA. La sequenza che si vuole amplificare è denotata in rosso, ed è affiancata da sequenze note (giallo) 2) La miscela è raffreddata a C (dipende da primer e stampo). I primer (verde) si legano alle sequenze complementari (giallo) in un processo di annealing) 3) Si aggiunge la polimerasi TAQ ( ) a 72 C e i desossinucleotidi-3p (, A,T,G,C) ed inizia il processo di sintesi polimerasica di DNA complementare allo stampo, sia della sequenza desiderata che di quella affiancante (celeste). 4) Si riporta a 95 C, entrambe i nuovi doppi filamenti si denaturano. Ne seguiamo solo uno (l altro è sbiadito) 5) Si ripete il ciclo di annealing come in (2) 6) Si ripete il ciclo di sintesi come in (3) sia sul filamento originale che quello nuovo 7) Il filamento indicato è identico a quello superiore del doppio filamento originale Rappresentazione ciclica del processo PCR (PCR.EXE)
22 DETERMINAZIONE DEI LIVELLI DI TRASCRIZIONE DI RNA METODO NORTHERN BLOT housekeeping gene (constitutively expressed) (e.g. G-3-P dehydrogenase) target gene
23 DETERMINAZIONE DEI LIVELLI DI TRASCRIZIONE DI RNA REAL TIME PCR Un metodo denominato anche PCR quantitativa che permette di amplificare e simultaneamente quantificare molecole di DNA o RNA. Dopo ogni ciclo di trascrizione (turno di amplificazione) l acido nucleico viene quantificato utilizzando sonde fluorescenti Può esser utilizzato per diagnostica o per determinare il livello di trascrizione di geni Uno dei metodi: TaqMan
24 sequenziamento del DNA sequenziatori di 1 generazione Metodo classico di SANGER: dideoxy terminator nucleotides dntp ddntp ddntp non può reagire polimerasi c dntp c c c c c c c c c c c c ddntp c c c c c c c c catena stampo primer catena trascritta
25 Metodo classico di SANGER: dideoxy terminator nucleotides Elettroforesi capillare VANTAGGI SVANTAGGI - Preciso - molto affidabile - Costo elevato - Efficiente, fino a1000 basi/corsa - laborioso (preparazione stampo difficile) - Versatile (Utile per altri tipi di analisi) - Permette di analizzare pochi campioni contemporaneamente (non è MP)
26 Shotgun sequencing Hierarchical shotgun kb cloned in BAC Whole genome shotgun 2,10,50,150 kb cloned in vector
27 sequenziamento del DNA sequenziatori high-throughput Next Generation Sequencing (NGS) Bead method (emulsion PCR) Bridge method (surface PCR) Massively Parallel Sequencing (MPS)
28 BRIDGE AMPLIFICATION: cluster method with bridge PCR amplication Preparation Bridge amplification Complementary DNA strands Cut and flush one of the strands Cap and sequence 1) Prepare lawn of complementary oligo pairs 2) Seed DNA strand to be sequenced from a DNA Library and anneal to complementary linked oligo primer (green) 3) Build complementary strand and then denature, leaving an anchored complementary strand 4) This strand bends to anneal the other end to the other complementary oligo (yellow) forming a bridge 5) PCR produces double a stranded bridge. This denatures to two anchored strands (one complementary the other original) 6) The two strands then anneal to form two bridges etc., leading to (7) bridge amplifiaction 8) At the end of amplifiactaion many strands of both complementary and original sequences are present, but only the original sequence is sequenced. 9) The complementary strand is cut and flushed away, 10) The original strand is capped and sequenced using appropriate primer 11) Note that conditions are chosen so that each strand to be sequenced binds distant to other strands 12) This means that aplified strands are clustered in different parts of the surface
29 Emulsion PCR and BEAD enrichment 1) Prepare a DNA fragment library with all strands ending with the same adapter sequences 2) Prepare beads (~600 nm) with complementary oligonucleotides to the adapter on their surface 3) Mix beads and DNA fragments in an aqueous suspension in oil 4) Choose concentrations so that statistically each water drop has only one bead and one DNA strand 5) Carry out PCR within each droplet. Each cycle a complementary strand grows from the anchored oligonucleotide 6) At the end of PCR amplification, denature to leave only single-stranded DNA linked to the bead 7) Remove beads from emulsion and then place each bead in the well of 1000 nm a multi-well sequencing platform (only one 600 nm bead can enter into each 1000 nm well) 6) Carry out «ion torrent» or «pyro» sequencing. ph variation Ion torrent h pyroseq
30 Metodo ion torrent
31 METODI FGS (First generation sequencing) e NGS (Next Generation Sequencing) Method DNA Amplification Sequencing Chemistry Detection TYPE Sanger PCR Dideoxy termination + Capillary electrophoresis + fluorescence detection LTS 454 Bead PCR Pyro sequencing + dntp PPi light Luminescence emission MPS Illumina Bridge PCR Fluorescence release + dntp + Fluorescence emission MPS Ion Torrent Bead PCR H + release + dntp H + ph ph variation MPS LTS = low throughput system (only few sequences processed simultaneously) MPS = massively parallel sequencing (very many sequences processed at the same time it is a high throughpt system) NB 454, Illumna and Ion Torrent all determine the sequence as the polymerase catalized synthesis of the strand is proceeding so are «sequence through synthesis» methods (STS)
32
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Lezioni di biotecnologie
 Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI
 LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
Quanto DNA è contenuto in una cellula?...
 Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Acidi Nucleici: DNA = acido deossiribonucleico
 Acidi Nucleici: DNA = acido deossiribonucleico depositario dell informazione genetica RNA: acido ribonucleico trascrizione e traduzione dell informazione genetica dogma centrale della biologia molecolare
Acidi Nucleici: DNA = acido deossiribonucleico depositario dell informazione genetica RNA: acido ribonucleico trascrizione e traduzione dell informazione genetica dogma centrale della biologia molecolare
ENZIMI DI RESTRIZIONE E DNA RICOMBINANTE
 ENZIMI DI RESTRIZIONE E DNA RICOMBINANTE GLI ENZIMI DI RESTRIZIONE Sono enzimi che tagliano la doppia elica del DNA in corrispondenza di specifiche sequenze (frammenti non casuali). La grandezza dei frammenti
ENZIMI DI RESTRIZIONE E DNA RICOMBINANTE GLI ENZIMI DI RESTRIZIONE Sono enzimi che tagliano la doppia elica del DNA in corrispondenza di specifiche sequenze (frammenti non casuali). La grandezza dei frammenti
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
estremità 5' DNA O - P O H 2 C O H H H H G N H O H 2 C P O H H T N ponte fosfodiestere O 3 P O estremità 3' etc.
 estremità 5' 5 3 - P N 5 2 C 3 ponte fosfodiestere P A 1 2 C 5 P 3 G N 1 2 C 5 3 P T N 1 5 2 C 3 etc. DNA C N 1 estremità 3' zucchero N 1 C 3 4 3 timina N adenina N N 1 6 N N 9 N zucchero N zucchero citosina
estremità 5' 5 3 - P N 5 2 C 3 ponte fosfodiestere P A 1 2 C 5 P 3 G N 1 2 C 5 3 P T N 1 5 2 C 3 etc. DNA C N 1 estremità 3' zucchero N 1 C 3 4 3 timina N adenina N N 1 6 N N 9 N zucchero N zucchero citosina
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Traduzione. Trascrizione
 Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
lezione martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Escherichia coli. Applicazioni biotecnologie con microrganismi procarioti. Una delle speci batteriche più biotecnologica è senza dubbio
 Applicazioni biotecnologie con microrganismi procarioti Escherichia coli, Bacillus subtilis con altri batteri appartenenti ai generi Pseudomonas, Rhizobium, Lactobacillus possono essere usati: - piani
Applicazioni biotecnologie con microrganismi procarioti Escherichia coli, Bacillus subtilis con altri batteri appartenenti ai generi Pseudomonas, Rhizobium, Lactobacillus possono essere usati: - piani
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Retrovirus e DNA ricombinante. Lezioni d'autore di Paola Vinesi
 Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
REPLICAZIONE DEL DNA 1
 REPLICAZIONE DEL DNA 1 La replicazione del DNA è semiconservativa: ciascuno dei due filamenti parentali serve da stampo per la sintesi di un nuovo filamento e le due nuove doppie eliche sono costituite
REPLICAZIONE DEL DNA 1 La replicazione del DNA è semiconservativa: ciascuno dei due filamenti parentali serve da stampo per la sintesi di un nuovo filamento e le due nuove doppie eliche sono costituite
Il DNA come molecola in grado di veicolare informazione ereditabile (genetica)
 Il DNA come molecola in grado di veicolare informazione ereditabile (genetica) Essenz. Alberts: cap 6 La trasmissione dell informazione replicazione trascrizione traduzione DNA RNA Proteina da, dg, dc,
Il DNA come molecola in grado di veicolare informazione ereditabile (genetica) Essenz. Alberts: cap 6 La trasmissione dell informazione replicazione trascrizione traduzione DNA RNA Proteina da, dg, dc,
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
Corso di Lingua Inglese
 Corso di Lingua Inglese Si ricorda a tutti gli studenti del 3 anno (immatricolati nell aa 2014/2015) che per potersi laureare dovranno superare la prova di idoneità in Lingua Inglese. Il Corso di Lingua
Corso di Lingua Inglese Si ricorda a tutti gli studenti del 3 anno (immatricolati nell aa 2014/2015) che per potersi laureare dovranno superare la prova di idoneità in Lingua Inglese. Il Corso di Lingua
TRASCRIZIONE DEL DNA. Formazione mrna
 TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
DNA e replicazione del DNA
 DNA e replicazione del DNA 1928: EXP di Griffith Scoperto il fattore trasformante Struttura elicoidale del DNA Struttura del DNA Subunità nucleotidiche Struttura del DNA Subunità nucleotidiche La replicazione
DNA e replicazione del DNA 1928: EXP di Griffith Scoperto il fattore trasformante Struttura elicoidale del DNA Struttura del DNA Subunità nucleotidiche Struttura del DNA Subunità nucleotidiche La replicazione
COSTRUZIONE DI GENOTECHE
 COSTRUZIONE DI GENOTECHE Definizione di genoteca Una genoteca è una collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Genoteche di DNA genomico Genoteche di DNA
COSTRUZIONE DI GENOTECHE Definizione di genoteca Una genoteca è una collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Genoteche di DNA genomico Genoteche di DNA
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
FORMAZIONE DEL LEGAME PEPTIDICO
 AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Polymerase Chain Reaction
 Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
MODALITA DI REPLICAZIONE
 MODALITA DI REPLICAZIONE In teoria è ipotizzabile che il DNA possa duplicarsi con modalità: 1) semi-conservativa se alla generazione successiva passano due doppie eliche entrambi costituite da un'elica
MODALITA DI REPLICAZIONE In teoria è ipotizzabile che il DNA possa duplicarsi con modalità: 1) semi-conservativa se alla generazione successiva passano due doppie eliche entrambi costituite da un'elica
Definizione di genoteca (o library) di DNA
 Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI
 CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
LA REPLICAZIONE DEL DNA
 www.fisiokinesiterapia.biz LA REPLICAZIONE DEL DNA REPLICAZIONE DEL DNA Durante il processo di replicazione la doppia elica del DNA si srotola e ciascuno dei due filamenti funziona da stampo per un nuovo
www.fisiokinesiterapia.biz LA REPLICAZIONE DEL DNA REPLICAZIONE DEL DNA Durante il processo di replicazione la doppia elica del DNA si srotola e ciascuno dei due filamenti funziona da stampo per un nuovo
Acidi Nucleici. Contenuto:
 Acidi Nucleici Contenuto: Il DNA e' l'unica molecola depositaria dell'informazione genetica, ossia del progetto nel quale sono immagazzinate istruzioni precise per tutte le caratteristiche ereditarie autoduplicazione
Acidi Nucleici Contenuto: Il DNA e' l'unica molecola depositaria dell'informazione genetica, ossia del progetto nel quale sono immagazzinate istruzioni precise per tutte le caratteristiche ereditarie autoduplicazione
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
La Terminazione della Trascrizione
 La Terminazione della Trascrizione I geni batterici possiedono dei terminatori della trascrizione. I Terminatori sono sequenze dell RNA che promuovono il distacco dell RNA polimerasi dal templato Ne esistono
La Terminazione della Trascrizione I geni batterici possiedono dei terminatori della trascrizione. I Terminatori sono sequenze dell RNA che promuovono il distacco dell RNA polimerasi dal templato Ne esistono
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Purificazione degli acidi nucleici
 A cosa serve? Purificazione degli acidi nucleici DNA genomico: per costruire librerie genomiche e/o usato nell analisi d ibridazione Southern. mrna: sintesi del cdna; analisi d ibridazione Northern, o
A cosa serve? Purificazione degli acidi nucleici DNA genomico: per costruire librerie genomiche e/o usato nell analisi d ibridazione Southern. mrna: sintesi del cdna; analisi d ibridazione Northern, o
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Biochimica e biochimica applicata Biologia molecolare CdL Farmacia
 Tecniche di biologia molecolare Introduzione alla biologia molecolare Struttura degli acidi nucleici Enzimi usati in biologia molecolare Gli enzimi di restrizione Ligasi Purificazione dell mrna e ottenimento
Tecniche di biologia molecolare Introduzione alla biologia molecolare Struttura degli acidi nucleici Enzimi usati in biologia molecolare Gli enzimi di restrizione Ligasi Purificazione dell mrna e ottenimento
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Struttura dei nucleotidi...6 Modello di Watson e Crick...10 Organizzazione strutturale superiore del DNA...13 DUPLICAZIONE DEL DNA...
 ACIDI NUCLEICI...2 FUNZIONI DEL DNA...5 FUNZIONI DELL RNA...5 I NUCLEOTIDI...6 Struttura dei nucleotidi...6 Modello di Watson e Crick...10 Organizzazione strutturale superiore del DNA...13 DUPLICAZIONE
ACIDI NUCLEICI...2 FUNZIONI DEL DNA...5 FUNZIONI DELL RNA...5 I NUCLEOTIDI...6 Struttura dei nucleotidi...6 Modello di Watson e Crick...10 Organizzazione strutturale superiore del DNA...13 DUPLICAZIONE
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
Mutagenesi sito-specifica
 Mutagenesi sito-specifica La mutagenesi sito-diretta può essere utilizzata anche per introdurre in una sequenza (anche una singola base o poche basi): sfruttando la capacità di questi oligonucleotidi di
Mutagenesi sito-specifica La mutagenesi sito-diretta può essere utilizzata anche per introdurre in una sequenza (anche una singola base o poche basi): sfruttando la capacità di questi oligonucleotidi di
MODELLO SCHEDA INSEGNAMENTO
 Corso di Laurea Denominazione insegnamento: Numero di Crediti: Anno: Semestre: Docente Titolare: MODELLO SCHEDA INSEGNAMENTO Triennale in Scienze Biologiche Biologia molecolare 9 CFU II II Lina Sabatino
Corso di Laurea Denominazione insegnamento: Numero di Crediti: Anno: Semestre: Docente Titolare: MODELLO SCHEDA INSEGNAMENTO Triennale in Scienze Biologiche Biologia molecolare 9 CFU II II Lina Sabatino
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Regolazione dell espressione genica
 Regolazione dell espressione genica definizioni Gene attivato quando viene trascritto in RNA e il suo messaggio tradotto in molecole proteiche specifiche Espressione genica processo complessivo con cui
Regolazione dell espressione genica definizioni Gene attivato quando viene trascritto in RNA e il suo messaggio tradotto in molecole proteiche specifiche Espressione genica processo complessivo con cui
Importanza della genetica dei microrganismi
 Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
REPLICAZIONE DEI GENOMI A DNA
 REPLICAZIONE DEI GENOMI A DNA LA REPLICAZIONE DEL DNA SERVE A PERPETUARE L INFORMAZIONE GENICA FIGURA 12.26 GENES VII FIGURA 13.1 GENOMI II FIGURA 13.2 GENOMI II NEL 1958 MESELSON E STAHL DIMOSTRANO CHE
REPLICAZIONE DEI GENOMI A DNA LA REPLICAZIONE DEL DNA SERVE A PERPETUARE L INFORMAZIONE GENICA FIGURA 12.26 GENES VII FIGURA 13.1 GENOMI II FIGURA 13.2 GENOMI II NEL 1958 MESELSON E STAHL DIMOSTRANO CHE
COME GENERARE UNA PIANTA TRANSGENICA UTILIZZANDO AGROBACTERIUM TUMEFACIENS
 COME GENERARE UNA PIANTA TRANSGENICA UTILIZZANDO AGROBACTERIUM TUMEFACIENS Agrobacterium è in grado di trasferire nel genoma vegetale qualsiasi sequenza si trovi delimitata dai right e left borders gene
COME GENERARE UNA PIANTA TRANSGENICA UTILIZZANDO AGROBACTERIUM TUMEFACIENS Agrobacterium è in grado di trasferire nel genoma vegetale qualsiasi sequenza si trovi delimitata dai right e left borders gene
IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE
 Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Plasmidi come vettori di clonaggio
 Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
SINTESI DELL RNA. Replicazione. Trascrizione. Traduzione
 SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
SINTESI DELL RNA Replicazione Trascrizione Traduzione L RNA ha origine da informazioni contenute nel DNA La TRASCRIZIONE permette la conversione di una porzione di DNA in una molecola di RNA con una sequenza
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo INGEGNERIA GENETICA
 CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
Legami idrogeno tra le coppie di basi
 Legami idrogeno tra le coppie di basi 4 3 6 1 4 3 2 6 1 2 Interazioni elettrostatiche deboli che si stabiliscono tra un atomo elettronegativo (es.ossigeno o azoto) e un atomo di idrogeno legato ad un secondo
Legami idrogeno tra le coppie di basi 4 3 6 1 4 3 2 6 1 2 Interazioni elettrostatiche deboli che si stabiliscono tra un atomo elettronegativo (es.ossigeno o azoto) e un atomo di idrogeno legato ad un secondo
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo REPLICAZIONE E COMPOSIZIONE DEL DNA
 CORSO DI GENETICA REPLICAZIONE E COMPOSIZIONE DEL DNA La struttura del DNA La replicazione del DNA Iduefilamenti della doppia elica parentale si srotolano generando ciascuno un filamento figlio secondo
CORSO DI GENETICA REPLICAZIONE E COMPOSIZIONE DEL DNA La struttura del DNA La replicazione del DNA Iduefilamenti della doppia elica parentale si srotolano generando ciascuno un filamento figlio secondo
10/30/16. non modificato CAP al 5 e poly-a al 3. RNA messaggero: soggetto a splicing
 procarioti eucarioti poli-cistronico mono-cistronico non modificato CAP al 5 e poly-a al 3 RNA messaggero: procarioti eucarioti policistronico monocistronico non modificato CAP al 5 e poly-a al 3 continuo
procarioti eucarioti poli-cistronico mono-cistronico non modificato CAP al 5 e poly-a al 3 RNA messaggero: procarioti eucarioti policistronico monocistronico non modificato CAP al 5 e poly-a al 3 continuo
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Enzimi di restrizione. di Vittorio Scornaienchi
 Enzimi di restrizione di Vittorio Scornaienchi Generalità Nucleasi Le nucleasi sono enzimi che tagliano i legami sui filamenti degli acidi nucleici: 5. Le esonucleasi aggrediscono una delle estremità libere
Enzimi di restrizione di Vittorio Scornaienchi Generalità Nucleasi Le nucleasi sono enzimi che tagliano i legami sui filamenti degli acidi nucleici: 5. Le esonucleasi aggrediscono una delle estremità libere
Francesca Ceroni. Biotecnologie tradizionali. Biologia Sintetica. F. Ceroni 16/09/2010. Bressanone GNB 2010 1. 1) DNA ricombinante 2) PCR
 XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Dal Genotipo al Fenotipo
 Dal Genotipo al Fenotipo Dal Fenotipo normale al Fenotipo patologico Regolazione dell espressione genica Figure 7-1 Molecular Biology of the Cell ( Garland Science 2008) Una cellula differenziata contiene
Dal Genotipo al Fenotipo Dal Fenotipo normale al Fenotipo patologico Regolazione dell espressione genica Figure 7-1 Molecular Biology of the Cell ( Garland Science 2008) Una cellula differenziata contiene
GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
La trascrizione è simile alla replicazione ma esistono alcune importanti differenze
 LA TRASCRIZIONE processo nel quale il DNA stampo viene copiato in una molecola di RNA La molecola di RNA è identica in sequenza all elica codificante e complementare a quella stampo. La trascrizione è
LA TRASCRIZIONE processo nel quale il DNA stampo viene copiato in una molecola di RNA La molecola di RNA è identica in sequenza all elica codificante e complementare a quella stampo. La trascrizione è
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
Percorso formativo di BIOLOGIA. I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico)
 Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
I VIRUS. Tutti i virus esistono in due stati: EXTRACELLULARE e INTRACELLULARE. Nel primo caso si parla comnemente di virioni o particelle virali.
 I VIRUS Un virus è definito come materiale nucleico (DNA o RNA) organizzato in una struttura di rivestimento proteico. Il materiale nucleico del virus contiene l informazione necessaria alla sua replicazione
I VIRUS Un virus è definito come materiale nucleico (DNA o RNA) organizzato in una struttura di rivestimento proteico. Il materiale nucleico del virus contiene l informazione necessaria alla sua replicazione
Tecniche di sequenziamento del DNA
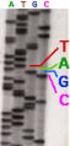 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Scientists with lab coats: Corso di laboratorio Chimico-Biologico. Dott.ssa Valeria Berton Università di Verona
 Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Il DNA Il DNA contiene l informazione genetica di un organismo Scritta in un codice chimico
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Applicazione della biologia molecolare nella valutazione del benessere del cavallo
 UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Dimensioni di Genomi
 Dimensioni di Genomi plasmids viruses bacteria fungi plants algae insects mollusks bony fish amphibians Il Genoma umano è costituito da circa 3 miliardi di bp e contiene un numero di geni pari a circa
Dimensioni di Genomi plasmids viruses bacteria fungi plants algae insects mollusks bony fish amphibians Il Genoma umano è costituito da circa 3 miliardi di bp e contiene un numero di geni pari a circa
Metabolismo, crescita e riproduzione batterica
 Metabolismo, crescita e riproduzione batterica 1 Ossigeno (presente o assente) Nutrienti (energia) Temperatura ottimale ph ottimale 2 OSSIGENO 1. Aerobi obbligati 2. Anaerobi obbligati 3. Aerobi/Anaerobi
Metabolismo, crescita e riproduzione batterica 1 Ossigeno (presente o assente) Nutrienti (energia) Temperatura ottimale ph ottimale 2 OSSIGENO 1. Aerobi obbligati 2. Anaerobi obbligati 3. Aerobi/Anaerobi
