Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS)
|
|
|
- Leona Gerardina Fabiani
- 6 anni fa
- Visualizzazioni
Transcript
1 Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS)
2 Per il sequenziamento di genomi la metodica principalmente utilizzata fino a pochi anni fa era basata su una strategia enzimatica, metodo della terminazione della catena sviluppato da Frederick Sanger (1977). Prevede uno step di clonaggio dei frammenti derivanti dalla diretta frammentazione del DNA genomico (o derivante dalla frammentazione di inserti di cloni BAC).
3
4 Più recentemente sono stati sviluppati nuovi metodi caratterizzati dalla capacità di sequenziare molti frammenti di DNA contemporaneamente (anche se con efficienza minore in termini di numero di basi sequenziate per frammento) aprendo una nuova era del sequenziamento. Queste metodiche di nuova concezione sono dette di NEXT GENERATION SEQUENCNG e prevedono un sequenziamento ad elevato parallelismo. Sanger 1000 bp X 384 reazioni contemporanee X 10 corse al giorno= (3,84 Mb) al giorno NGS bp X milioni di sequenze. Una corsa 0,5-10 Gb!!!
5 NEXT GENERATION SEQUENCNG - Non è più indispensabile il clonaggio in vettori batterici o virali. - Non è più necessaria la fase di migrazione dei frammenti su gel o in capillare per ottenere la sequenza. - La lettura della sequenza è effettuata step-by-step (sequencing by synthesis).
6 NEXT GENERATION SEQUENCNG - E necessaria la fase di amplificazione del DNA genomico prima del sequenziamento. Fra le tecnologie attualmente disponibili: 1) Roche/454 (pyrosequencing) dal ) Applied Biosystems SOLID (sequencing by ligation). Dal ) Illumina/Solexa (modified Sanger, reversible dyeterminators) dal 2006
7 Roche 454 GS-FLX System Using Titanium Chemistry Microbial whole genome sequencing BAC sequencing Metagenomics Mutation discovery mirna discovery cdna library screening ~500bp average read length milion reads/plate Obtain up to 500Mb per run (10 ore)
8 Anche la metodica del PYROSEQUENCING si basa sulla replicazione del DNA
9 1) PYROSEQUENCING (Ronaghi M, Ehleen M and Nyrén P (1998) A sequencing method based on real-time pyrophosphate. Science, 238, ).
10 PYROSEQUENCING: 4 passaggi ripetuti ciclicamente 1. La sequenza da analizzare, previa amplificazione con PCR, viene incubata come singola elica insieme a: -primers adeguati DNA polimerasi -ATP solforilasi e agli enzimi: - luciferasi apirasi -adenosinsolfofosfato (ASP) -luciferina 2. Un solo tipo di dntp alla volta viene aggiunto alla reazione. Per ogni tipo di dntp aggiunto si verificano due possibilità esclusive: in caso di non complementarietà allo stampo nella prima posizione dopo il primer, l'allungamento non avviene e il nucleotide mismatching viene degradato dalla apirasi; in caso di complementarietà, la DNA polimerasi ne catalizza l'aggiunta con liberazione di pirofosfato inorganico Ppi.
11 Metodi di Next Generation Sequencing (NGS) 3. Il PPi così prodotto viene quindi rivelato dalla produzione di un segnale luminoso: partendo dall' adenosinsolfofosfato (ASP) come substrato, la solforilasi catalizza la trasformazione del PPi in ATP, il quale fornisce energia per la conversione della luciferina ad ossiluciferina (ad opera della luciferasi). Il segnale luminoso così generato viene rilevato e registrato da una ccd. La presenza del segnale ci conferma l'appartenenza del nucleotide alla tal posizione della catena, mentre l'intensità del segnale sarà proporzionale al numero di ripetizioni della base lungo lo stesso filamento: un impulso doppio o triplo, per esempio, è indice dell'inglobamento nello stesso ciclo di 2 dntp (ripetizione della stessa base per 2 o 3 volte sul templato); viceversa un segnale nullo indica che il dntp aggiunto in quel ciclo non è complementare.
12 Metodi di Next Generation Sequencing (NGS) PYROSEQUENCING 4. L'enzima apirasi, degrada in continuazione l'eccesso di dntp che non è stato incorporato, e l'eccesso di ATP prodotto dalla solforilasi. Solo quando l'eccesso di ATP è stato eliminato completamente si può aggiungere un secondo dntp per far progredire la reazione di polimerizzazione (ritornando allo step 1). Si aggiungono quindi ciclicamente tutti e 4 i dntp fino al completamento della sequenza. (N.B. per l adenina non si può utilizzare ATP, ma l'adenosin-α-tiotrifosfato, che è un analogo riconosciuto dalla DNA polimerasi come se fosse ATP, ma non dalla luciferasi; l ATP produrrebbe infatti un segnale luminoso continuo che non deriverebbe dalla reazione di conversione del pirofosfato, ma dall'aggiunta tal quale di ATP, utilizzato direttamente dalla luciferasi).
13 Metodi di Next Generation Sequencing (NGS) PYROSEQUENCING Dall'analisi del pirogramma si risale alla sequenza completa. TCTCTGTAAAGTGG TCTCTATAAAGTGG Esempio di SNP rivelato dall analisi di un pirogramma
14 Metodi di Next Generation Sequencing (NGS) PYROSEQUENCING Filmato riassuntivo: pyrosequencing.flv
15 PYROSEQUENCING PER SEQUENZIARE INTERI GENOMI: L ELEVATO PARALLELISMO In realtà siamo già oltre 500 basi per corsa.
16 Preparazione della Library genomica: frammentazione e ligation di adattatori
17 Attacco dei frammenti di DNA a sferette con adattatori. In una sferetta si lega un solo frammento. Si emulsionano le sferette in una water-in-oil mixture contenente i reagenti per la amplificazione PCR. Amplificazione clonale ( emulsion PCR )
18 Le sferette sono inserite in supporti contenenti circa 1 milione di micro-pozzetti in cui avviene la reazione di sequenziamento per l aggiunta di sferette contenenti i reagenti per il sequenziamento
19
20
21 Sequencing by Ligation - SOLID TM system Il sistema SOLID prodotto da Applied Biosystem è una piattaforma per il sequenziamento in parallelo di segmenti di DNA amplificati in modo clonale e legati a sferette magnetiche. La metodologia di sequenziamento è basata sulla "ligazione sequenziale" di oligonucleotidi marcati con fluorocromi (SBL, sequencing by ligation). - fino a 10 Gbp per run -sequenze di lunghezza pari a nt con un'accuratezza superiore al 99% milioni di reads - Possibilità di caricare fino a 8 campioni in canali separati -Una corsa dura 3-6 giorni Attualmente il suo uso va diminuendo.
22 Preparazione della library con i frammenti legati a sferette magnetiche amplificati con Emulsion PCR e distribuiti su speciali vetrini 1-Enulsion PCR 2-Clonal amplification 3-Enrichment for DNApositive beads 4-Transfer on solid array
23 Si basa sulla ligazione sequenziale di oligonucleotidi marcati con fluorocromi differenti. La reazione è ciclica, ad ogni ciclo si aggiunge un oligonucleotide differente di 8 bp. La struttura degli oligonucleotidi fluorescenti è NNnnnZZZ* - NN è una delle 16 combinazioni dei 4 nucleotidi A, C, T, G. -nnn sono tre basi degenerate -ZZZ sono basi che legano il fluoroforo. -Tra le basi nnn e ZZZ c è un sito di taglio per una nucleasi.
24 Ogni ciclo è composto di vari step: annealing, ligation, lavaggio, visualizzazione e taglio dell estremità 5 del primer fluorescente. Al primo ciclo si aggiungono in sequenza i 16 oligonucleotidi possibili: il primo oligonucleotide si prova ad appaiare in 5 del primer universale di innesco lungo n attaccato all estremità 5 del filamento attaccato alla sferetta. Se non c è complementarietà si passa al secondo oligonucleotide possibile e così via. Se c è complementarietà la ligasi lega l oligo, e viene registrata la fluorescenza. Successivamente una nucleasi rimuove l estremità 5 dell oligo e si ricomincia ad aggiungere in sequenza i 16 oligonucleotidi possibili.
25
26
27 Al termine di 7-8 cicli il filamento neo sintetizzato a partire dal primer universale di innesco lungo n è rimosso e si fa ibridare un primer universale di innesco lungo n-1 all estremità 5 del filamento stampo.
28 Quindi si ripetono altri 7-8 cicli col primer lungo n-1 e successivamente con primer lungo n-2, n-3 e n-4.
29 n n-1 n-2 n-3 n-4 1/2 6/7 11/12 16/17 18/19 20/21 22/23 ATxxxACxxxAAxxxATxxxTCxxxAAxxxGCxxx CAxxxAAxxxTAxxxCAxxxGTxxxTAxxxCGxxx TAxxxGTxxxTCxxxCGxxxGTxxxGCxxx GTxxxCGxxxCTxxxGCxxxCGxxxGGxxx TGxxxCCxxxACxxxTGxxxCCxxxAGxxx seq CATGTAACCGTAACTCATGCGTCCGTAAGGCGC
30 Ho 4 fluorocromi (colori) ma devo distinguere 16 dinucleotidi L associazione colore dinucleotide è studiata in modo da facilitare la lettura della sequenza finale...
31 Conoscendo la Base Zero...
32 La base zero è data dall ultima base del primer universale lungo n n n-1 n-2 n-3 n-4 Primer univ 1/2 6/7 11/12 16/17 18/19 20/21 22/23 ATTTCGACGACC ATxxxACxxxAAxxxATxxxTCxxxAAxxxGCxxx.. ATTTCGACGAC CAxxxAAxxxTAxxxCAxxxGTxxxTAxxxCGxxx.. ATTTCGACGA TAxxxGTxxxTCxxxCGxxxGTxxxGCxxx.. ATTTCGACG GTxxxCGxxxCTxxxGCxxxCGxxxGGxxx.. ATTTCGAC TGxxxCCxxxACxxxTGxxxCCxxxAGxxx.. seq CATGTAACCGTAACTCATGCGTCCGTAAGGCGC..
33 Sapendo che il nucleotide zero è una A, c è solo una sequenza possibile A-T T-A C-G G-C G-A A-G T-C C-T A-C C-A G-T T-G A-A T-T C-C G-G
34 Sapendo che il nucleotide zero è una A, c è solo una sequenza possibile A-T T-A C-G G-C G-A A-G T-C C-T A-C C-A G-T T-G A-A T-T C-C G-G A T C A A
35 Sequencing by Synthesis - SOLEXA TM / ILLUMINA system Il sistema SOLEXA prodotto da Illumina è una piattaforma per il sequenziamento in parallelo di segmenti di DNA legati sulla superficie di un vetrino (flowcell) diviso in 8 lanes ed amplificati in modo clonale attraverso una Bridge PCR. La metodologia di sequenziamento è basata sulla sintesi sequenziale" di DNA attraverso l utilizzo di terminatori dideossi reversibili. Il sistema SOLEXA può generare circa un miliardo di bp per run (sequenze di lunghezza fino a 150 nt) con un'accuratezza superiore al 99%.
36 Solexa technology. Illumina Genome Analyzer Generating high quality readout of one billion bases per run at less than 1% of the cost of capillary-based methods, the Illumina Genome Analyzer is designed to enable researchers to dramatically improve the efficiency of current applications. Extremely high throughput: 2G sequences per run Scalable: Up to eight samples can be loaded onto the flow cell simultaneously High accuracy Cost effective Bioinformatics solution by professionals Genome resequencing BAC resequencing Expression profiling Small RNA identification Single ends sequencing (2-3 giorni) or Paired ends sequencing (4-6 giorni)
37 Illumina: la libreria di frammenti di DNA legati ad adattatori è attaccata al vetrino, niente sferette o micropozzetti. Il vetrino è cosparso di primer adattatori
38 Illumina: primo ciclo della Bridge PCR: l estremità libera di un frammento di DNA si piega a ponte e si ibrida ad un primer adattatore vicino con sequenza complementare
39 Dopo vari cicli di Bridge PCR si creano circa 10 milioni di gruppi (cluster) ciascuno con milioni di copie della stessa molecola di DNA, come fossero dei veri e propri cloni in una piasra
40 Inizia il sequenziamento: si aggiungono primer, enzima e terminatori dideossi reversibili marcati con fluorocromi. Si attacca la prima base a ciascun cluster, si eccitano i fluorocromi con luce laser e si cattura la prima immagine del vetrino
41 I nucleotidi dideossi reversibili possono consentire l aggiunta di nuovi nucleotidi in modo da non bloccare l allungamento della catena di DNA. Successivamente si lavano via tutti i reagenti e si aggiungono nuovi terminatori dideossi reversibili e si acquisisce l immagine della seconda base.
42 Col susseguirsi dei cicli di sequenza, in ciascun punto del vetrino si registrano immagini successive che il software tramuta in stringhe di sequenza (reads).
43 Illumina Emissione di fluorescenza sul vetrino. Ogni spot luminoso sul vetrino corrisponde a un frammento e il colore indica la base aggiunta.
44 Riassumendo il metodo Illumina
45 Riassumendo il metodo Illumina: video NGS.flv
46 NGS e sequenziamento dei genomi Per il sequenziamento di un genoma, a livello di assemblaggio, data la bassa taglia dei frammenti sequenziati, è importante utilizzare anche un altra informazione, la lunghezza dei frammenti stampo, in modo da tenere conto che fra le due estremità di un frammento (paired ends) c è un numero preciso di nucleotidi
47 Sequenza unica con marcatore molecolare Regione con sequenze ripetute, difficile da assemblare Frammento di 8000bp Frammento di 20000bp???????? Porzione di frammento sequenziato in 5 Porzione di frammento sequenziato in 3
48 Sequenziamento di un genoma usando NGS Illumina (ma anche 454): sono sequenze troppo corte Questo pone problemi, per esempio riguardo alle sequenze ripetute (metafora del puzzle)
49 grandezza dei pezzi = lunghezza delle sequenze Pezzi più grandi rendono il puzzle più facile = sequenze più lunghe rendono l assemblaggio del genoma più facile tuttavia ottenere sequenze lunghe con metodi di sequenziamento tradizionali (Sanger) è costoso tecniche di sequenziamento di next generation sequencing sono decisamente più economiche ma forniscono sequenze più corte rispetto ai metodi tradizionali Si cerca un compromesso, spesso attraverso soluzioni ibride tra metodi tradizionali e di next generation sequencing
50 Infatti NGS è utile per disporre a basso prezzo, di una grande copertura del genoma, con cui integrare dati ottenuti con Sanger Per avere contig più grandi, conviene partire da library BAC e sequenziare quelle (come se ogni BAC fosse un piccolo genoma) mediante NGS; i contig verranno poi più facilmente assemblati Per l assemblaggio inoltre è molto utile disporre di mappe genetiche sempre più sature in modo da poter ancorare i contig
51 Sequenziamento di un genoma usando NGS NGS molto utile per il risequenziamento, dove si dispone di un genoma già sequenziato che fa da riferimento. Tuttavia, mutazioni tipo inserzioni o delezioni di frammenti di DNA possono essere sottostimate. Per questo c è necessità di algoritmi sempre più efficienti che tengano conto della lunghezza del frammento compreso fra le due paired ends sequenziate. Genoma A completamente sequenziato (di riferimento) Genoma B risequenziato con (NGS) con inserzione rispetto ad A Complessivamente, si rivela comunque utile disporre di una library BAC della specie da sequenziare.
52 Annotazione di un genoma e NGS -Geni: uso di software predittivi e di allineamento con library EST; allineamento con db pubblici (BLASTx e BLASTn)- RepeatMasker -Sequenze ripetute: J dotter; uso di software predittivi (LTR finder); allineamento con db pubblici (BLASTx, BLASTn, RepBase); Allineamento con sequenze Illumina (mapping)
53 Mapping delle reads Regioni genomiche ripetute Contig da NGS Reads illumina che mappano col contig Le regioni a cui si allineano numerose reads contengono sequenze ripetute
54 Mapping delle reads Annotazione e mappaggio con reads Illumina di tre cloni BAC di girasole (DES, DHN, LTP) Sequenze di trasposoni sono indicate seguendo la nomenclatura di Wicker et al. (2007). I picchi ci danno un idea della ripetitività di una sequenza La mappatura è utile per trovare sequenze ripetute ignote (v. contig 61)
55 Genoma dollari? 2003 genoma umano è stato "sostanzialmente completo" costo di 3 miliardi di dollari o 300 milioni di dollari (Celera Genomics). 2005, il consorzio HapMap individua le ubicazioni di un milione di SNPs nel genoma umano Entrambe queste pietre miliari hanno aperto la porta a numerose opportunità di ricerca. Una di queste è un genoma dollari. Il genoma dollari servirà a comprendere la diversità del genoma umano e scoprire le sue variazioni, per determinare le caratteristiche che possono essere eliminate per garantire una migliore salute delle persone. Attualmente, gran parte della ricerca su questi aspetti non è stata fatta, perché ogni sequenza del genoma compiuta con metodi tradizionali costa un milione di $ e circa un anno di lavoro. Il genoma $ è realistico? Quanto tempo sarà l'attesa? Attualmente ci sono diverse piattaforme commerciali, come Illumina e Affymetrix, che hanno dimostrato la loro capacità di valutare rapidamente SNPs conosciuti a costi assai contenuti. Tuttavia, la tecnologia attuale non è ancora capace di scoprire nuovi SNPs o altri polimorfismi. Ma la tecnologia è in continua e rapida evoluzione.
56 Il progetto genoma $ è iniziato nel 2004, con il National Human Genome Research Institute (NHGRI). Il primo obiettivo, che mirava a ridurre il costo del sequenziamento a $, è stato raggiunto in cinque anni. Con le tecnologie attuali il costo del sequenziamento di un genoma umano è circa $ e sette giorni di lavoro, quindi, il traguardo non è lontano. Tuttavia, con sorpresa di tutti, Pacific Biosciences, una società di biotecnologie che produce una piattaforma per il sequenziamento di singole molecole di DNA, ha annunciato che sarà in grado di mappare un genoma in 15 minuti per meno di $ entro il La Life Technologies nel gennaio di quest anno ha annunciato Proton Ion per il sequenziamento del genoma umano in 24h per 1000 dollari. Illumina ha annunciato Hiseq2500 Realisticamente, prima che queste tecnologie possano essere utilizzate su larga scala, si può supporre un periodo di attesa di 3-5 anni. La riduzione dei costi può influenzare la qualità delle Reads? La qualità delle reads è sempre stata la prima priorità per coloro che lavorano al progetto genoma di dollari. L'obiettivo è di avere una sequenza de-novo assemblata con non più di un errore per nucleotidi e non lasciare spazi vuoti. Per anni è stato utilizzato il metodo Sanger, con adeguata lunghezza e qualità delle sequenze. Nel corso degli anni il metodo ha ridotto i costi a $ 0,6 per basi, ma per un genoma umano occorrerebbe quasi un anno di lavoro e 1 milione di dollari!
57 Tecniche di NGS e nuovi algoritmi per l assemblaggio sono in grado di offrire risultati migliori, meno costosi e in tempi più rapidi, anche se forniscono sequenze più brevi ed hanno bisogno di una copertura più elevata per essere assemblate. Roche 454, per esempio, necessita di una copertura di 20X per assemblare contig di dimensioni utili. Il 454 è in grado di sequenziare un piccolo genoma al costo di 0.07$/ kb, Solexa $/kb, Solid 0.004$/kb. Un genoma umano ha un costo < $ Occorrerà attendere tecniche di next next generation sequencing
58 La tecnologia sarà disponibile per applicazioni mediche? Il campo della medicina non sarà in grado di accedere al genoma dollari subito. Al costo del sequenziamento occorre aggiungere l analisi computazionale delle sequenze di DNA, inoltre occorrerà tempo e denaro per fornirsi di attrezzature mediche e per la preparazione di medici in grado di interpretare le sequenze. Inoltre le sequenze che si potranno ottenere probabilmente non saranno molto precise da essere utilizzare per uso medico. La precisione necessaria aumenterebbe i costi di 10 volte, un prezzo che non molte strutture possono essere in grado di pagare. Quando anche i costi per l accuratezza si ridurranno il sistema sanitario sarà in grado di utilizzare le nuove tecnologie di sequenziamento del DNA per ridurre le malattie genetiche e contribuire a ridurre le spese mediche ad esse correlate. Il genoma personale porterà a problemi etici, legali e medici come la discriminazione a causa di un genoma sbagliato? I prossimi anni saranno cruciali per il genoma di dollari.
59 Sui grafici del National Human Genome Research Institute (INGRI) è riportato il costo del sequenziamento (per Megabasi e per genoma umano) e anche l andamento della legge di Moore, che indica la velocità secondo la quale aumentano le prestazioni dei processori. Fa abbastanza impressione notare come la nostra capacità di produrre sequenze di DNA stia diventando molto ma molto superiore alla capacità di analizzarle!!
60 Dal gennaio 2008 si è passati dal Sanger al NGS
61 Dal gennaio 2008 si è passati dal Sanger al NGS
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
NEXT- GENERATION DNA SEQUENCING
 NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
Tecniche di sequenziamento del DNA
 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Biodiversità Molecolare: aspetti di base e nuove tecnologie
 Biodiversità Molecolare: aspetti di base e nuove tecnologie Graziano PESOLE Università di Bari & IBBE-CNR, Bari, Italy Seminari sulla Biodiversità Bari, 18 Febbraio 2011 La Biodiversità Per biodiversità
Biodiversità Molecolare: aspetti di base e nuove tecnologie Graziano PESOLE Università di Bari & IBBE-CNR, Bari, Italy Seminari sulla Biodiversità Bari, 18 Febbraio 2011 La Biodiversità Per biodiversità
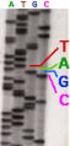
Lettura del sequenziamento: il cromatogramma
 CLONAGGIO E SEQUENZIAMENTO Per sequenziare il DNA è importante clonare i frammenti di sequenza ignota in appositi vettori. I frammenti sono ottenuti mediante frammentazione del genoma o, nel caso di produzione
CLONAGGIO E SEQUENZIAMENTO Per sequenziare il DNA è importante clonare i frammenti di sequenza ignota in appositi vettori. I frammenti sono ottenuti mediante frammentazione del genoma o, nel caso di produzione
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Biologia Molecolare. CDLM in CTF La Replicazione del DNA
 Biologia Molecolare CDLM in CTF 2010-2011 La Replicazione del DNA Prospettiva Storica La replicazione semidiscontinua Differenze tra procarioti ed eucarioti E fondamentale che ad ogni divisione cellulare
Biologia Molecolare CDLM in CTF 2010-2011 La Replicazione del DNA Prospettiva Storica La replicazione semidiscontinua Differenze tra procarioti ed eucarioti E fondamentale che ad ogni divisione cellulare
Sequenziamento del DNA. Preparazione di librerie. Library di cdna e di DNA genomico. Analisi di librerie. Sequenziamento del DNA
 Sequenziamento del DNA Preparazione di librerie Library di cdna e di DNA genomico Analisi di librerie Sequenziamento del DNA 1) Metodo di Maxam&Gilbert (taglio chimico): il DNA viene marcato ad un estremità
Sequenziamento del DNA Preparazione di librerie Library di cdna e di DNA genomico Analisi di librerie Sequenziamento del DNA 1) Metodo di Maxam&Gilbert (taglio chimico): il DNA viene marcato ad un estremità
Corso di Elementi di Bionformatica
 Corso di Elementi di Bionformatica Laurea Triennale in Informatica Il formato FASTQ per la qualità delle sequenze Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 La qualità delle sequenze
Corso di Elementi di Bionformatica Laurea Triennale in Informatica Il formato FASTQ per la qualità delle sequenze Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 La qualità delle sequenze
Materiale del corso: http://bioinformatica.uniroma2.it/gc2014/
 Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Summer School 2015. Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi
 Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
FORMAZIONE DEL LEGAME PEPTIDICO
 AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Avanzamento dei sistemi di sequenziamento
 Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Elementi ripetuti del Genoma Umano. Milioni di anni
 Elementi ripetuti del Genoma Umano Patologia Milioni di anni Evoluzione Un gene è duplicato Un gene è riarrangiato Genoma Il Genoma è l intero DNA contenuto in una Cellula Geni Sequenze Intergeniche Sequenze
Elementi ripetuti del Genoma Umano Patologia Milioni di anni Evoluzione Un gene è duplicato Un gene è riarrangiato Genoma Il Genoma è l intero DNA contenuto in una Cellula Geni Sequenze Intergeniche Sequenze
Retrovirus e DNA ricombinante. Lezioni d'autore di Paola Vinesi
 Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
MICROARRAY Esempio generico
 MICROARRAY MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione
MICROARRAY MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Traduzione. Trascrizione
 Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Duplicazione del DNA IL DNA è LA MOLECOLA DEPOSITARIA DELL INFORMAZIONE GENICA CHE DEVE ESSERE EREDITATA DI GENERAZIONE IN GENERAZIONE. PERTANTO LA STRUTTURA DEL DNA DEVE ESSERE TALE DA SODDISFARE AL MEGLIO
Genomica, proteomica, genomica strutturale, banche dati.
 Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Malattie genetiche e progetto genoma: a che punto siamo arrivati?
 Malattie genetiche e progetto genoma: a che punto siamo arrivati? dott. Elena Belloni Gruppo di ricerca IEO: Meccanismi molecolari del cancro e dell invecchiamento (responsabile prof. Pier Giuseppe Pelicci)
Malattie genetiche e progetto genoma: a che punto siamo arrivati? dott. Elena Belloni Gruppo di ricerca IEO: Meccanismi molecolari del cancro e dell invecchiamento (responsabile prof. Pier Giuseppe Pelicci)
Il Genoma Umano e La Bioinformatica. Marin Vargas, Sergio Paul
 Il Genoma Umano e La Bioinformatica Marin Vargas, Sergio Paul Gennaio 2014 L'acido desossiribonucleico o deossiribonucleico (DNA), che si trova nel nucleo della cellula, è un acido nucleico che contiene
Il Genoma Umano e La Bioinformatica Marin Vargas, Sergio Paul Gennaio 2014 L'acido desossiribonucleico o deossiribonucleico (DNA), che si trova nel nucleo della cellula, è un acido nucleico che contiene
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Next-generation sequencing : dalla ricerca di base alla diagnostica*
 Next-generation sequencing : dalla ricerca di base alla diagnostica* Karl V. Voelkerding 1,2, Shale A. Dames 1, Jacob D. Durtschi 1 1ARUP Institute for Experimental and Clinical Pathology, Salt Lake City,
Next-generation sequencing : dalla ricerca di base alla diagnostica* Karl V. Voelkerding 1,2, Shale A. Dames 1, Jacob D. Durtschi 1 1ARUP Institute for Experimental and Clinical Pathology, Salt Lake City,
Lezione IX-X martedì 25-X-2011
 Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Genomica Servizio Sequenziamento DNA
 Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
CORSO INTEGRATO DI GENETICA a.a. 2011-2012. Genetica molecolare in medicina: Analisi di Mutazioni. Cristina Bombieri 7 dicembre 2011
 CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
Next Generation Sequencers: from the bacterial culture to raw data. Valeria Michelacci NGS course, June 2015
 Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
La farmacogeneticanella gestione del paziente con tumore colorettale, l esperienza traslazionaleal CRO di Aviano
 SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Lezione 5. Next Generation Sequencing
 Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola)
 Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
Plasmidi come vettori di clonaggio
 Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona
 Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
GENOMICA. = conoscenza del DNA. Creazione banche banchedati datidel del DNA. Conoscenza di difenomeni biologici
 GENOMICA = conoscenza del DNA Conoscenza di difenomeni biologici Creazione banche banchedati datidel del DNA DNA Prevenzione e cura curadi dimalattie (screening del (screening DNA del per DNA malattie
GENOMICA = conoscenza del DNA Conoscenza di difenomeni biologici Creazione banche banchedati datidel del DNA DNA Prevenzione e cura curadi dimalattie (screening del (screening DNA del per DNA malattie
Analisi di sequenziamento degli acidi nucleici
 Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
La struttura covalente delle proteine (la sequenza amminoacidica)
 La struttura covalente delle proteine (la sequenza amminoacidica) Sequenza amminoacidica dell ormone insulina bovino (Frederick Sanger, 1953) Il primo passo per determinare la sequenza di un peptide è
La struttura covalente delle proteine (la sequenza amminoacidica) Sequenza amminoacidica dell ormone insulina bovino (Frederick Sanger, 1953) Il primo passo per determinare la sequenza di un peptide è
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica
 SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica Prof. Francesco Michelotti SAPIENZA Università di Roma Facoltà di Ingegneria
SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica Prof. Francesco Michelotti SAPIENZA Università di Roma Facoltà di Ingegneria
Genomics Session. Lezione 2 Assemblaggio del genoma
 Genomics Session Assemblaggio del genoma Genomica Computazionale, Laurea Magistrale AA 2010/2011 Genome assembly software: Celera Whole-genome Assembler Maschera sequenze ripetute Identifica regioni sovrapposte
Genomics Session Assemblaggio del genoma Genomica Computazionale, Laurea Magistrale AA 2010/2011 Genome assembly software: Celera Whole-genome Assembler Maschera sequenze ripetute Identifica regioni sovrapposte
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI
 LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
La trascrizione. La trascrizione è la sintesi delle molecole di RNA sulla base di un filamento stampo di DNA
 LA TRASCRIZIONE La trascrizione La trascrizione è la sintesi delle molecole di RNA sulla base di un filamento stampo di DNA Le caratteristiche dell RNA La costituzione a singolo filamento permette alle
LA TRASCRIZIONE La trascrizione La trascrizione è la sintesi delle molecole di RNA sulla base di un filamento stampo di DNA Le caratteristiche dell RNA La costituzione a singolo filamento permette alle
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
La mappatura dei geni umani. SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione
 La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
Leggere il Dna. Innovazione. Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica.
 Innovazione Leggere il Dna Rosario Iacono Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica. Nel 1977 fu pubblicato il metodo per il sequenziamento delle
Innovazione Leggere il Dna Rosario Iacono Dal metodo Sanger al Next generation sequencing: il genoma a disposizione della ricerca scientifica. Nel 1977 fu pubblicato il metodo per il sequenziamento delle
SERVIZIO DI SEQUENZIAMENTO AUTOMATICO
 REPARTO GENOMICA LABORATORIO ANALISI GENOMICHE SERVIZIO DI SEQUENZIAMENTO AUTOMATICO DOCUMENTO ILLUSTRATIVO DEL SERVIZIO DI SEQUENZIAMENTO ACIDI NUCLEICI ISTITUTO ZOOPROFILATTICO SPERIMENTALE DELLA LOMBARDIA
REPARTO GENOMICA LABORATORIO ANALISI GENOMICHE SERVIZIO DI SEQUENZIAMENTO AUTOMATICO DOCUMENTO ILLUSTRATIVO DEL SERVIZIO DI SEQUENZIAMENTO ACIDI NUCLEICI ISTITUTO ZOOPROFILATTICO SPERIMENTALE DELLA LOMBARDIA
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Tecniche biofisiche basate su nanopori
 Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI
 VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
CORSO BIOLOGIA MOLECOLARE I. Testi consigliati
 CORSO BIOLOGIA MOLECOLARE I Dott. Massimo Pancione e.mail massimo.pancione@unisannio.it Obiettivo del corso: Comprendere i meccanismi molecolari dei processi biologici fondamentali, descrivere le tecniche
CORSO BIOLOGIA MOLECOLARE I Dott. Massimo Pancione e.mail massimo.pancione@unisannio.it Obiettivo del corso: Comprendere i meccanismi molecolari dei processi biologici fondamentali, descrivere le tecniche
Francesca Ceroni. Biotecnologie tradizionali. Biologia Sintetica. F. Ceroni 16/09/2010. Bressanone GNB 2010 1. 1) DNA ricombinante 2) PCR
 XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
XXIX Scuola Annuale di Bioingegneria. Bressanone, 13-17 settembre 2010 Francesca Ceroni Biotecnologie tradizionali 1) DNA ricombinante 2) PCR 3) Sequenziamento automatizzato Biologia Sintetica 4) Approccio
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo INGEGNERIA GENETICA
 CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
Percorso formativo di BIOLOGIA. I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico)
 Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
Diagnosi genetiche molecolari
 Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Il sequenziamento del DNA
 Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
Analisi MLPA & Troubleshooting
 Analisi MLPA & Troubleshooting - Fondata nel 1985 - Focalizzata in precedenza sulla produzione di endonucleasi e marker molecolari - Dal 2002 tecnica MLPA : - ~400 probemix - Usate in tutto il mondo per
Analisi MLPA & Troubleshooting - Fondata nel 1985 - Focalizzata in precedenza sulla produzione di endonucleasi e marker molecolari - Dal 2002 tecnica MLPA : - ~400 probemix - Usate in tutto il mondo per
David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis. Biologia La scienza della vita
 1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione Il linguaggio della vita 3 Il materiale genetico
1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione Il linguaggio della vita 3 Il materiale genetico
Lezione 1-2 martedì 9 Marzo corso Biologia applicata BU, Ingegneria genetica BCM
 Lezione 1-2 martedì 9 Marzo 2010 corso Biologia applicata BU, Ingegneria genetica BCM libro di testo un libro di testo con parte degli argomenti del corso è: Biotecnologie molecolari Principi e tecniche
Lezione 1-2 martedì 9 Marzo 2010 corso Biologia applicata BU, Ingegneria genetica BCM libro di testo un libro di testo con parte degli argomenti del corso è: Biotecnologie molecolari Principi e tecniche
Espressione ed utilizzo della informazione genetica II Trascrizione e Traduzione
 Espressione ed utilizzo della informazione genetica II Trascrizione e Traduzione CdL Tecnici di Lab Biomedico AA. 2011-12 - Prof.ssa Frabetti Come si esprime l informazione? Per i geni classici vedremo:
Espressione ed utilizzo della informazione genetica II Trascrizione e Traduzione CdL Tecnici di Lab Biomedico AA. 2011-12 - Prof.ssa Frabetti Come si esprime l informazione? Per i geni classici vedremo:
Paraplegie Spastiche Ereditarie - PSE
 LE PARAPLEGIE SPASTICHE EREDITARIE: Una malattia, molti geni e molti ancora da scoprire E. Storti A. Tessa F. M. Santorelli Paraplegie Spastiche Ereditarie - PSE Cos è una paraparesi spastica ereditaria
LE PARAPLEGIE SPASTICHE EREDITARIE: Una malattia, molti geni e molti ancora da scoprire E. Storti A. Tessa F. M. Santorelli Paraplegie Spastiche Ereditarie - PSE Cos è una paraparesi spastica ereditaria
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Il benessere passa dalla tua tavola
 Il benessere passa dalla tua tavola Test Genetico per il metabolismo dell omocisteina. Introduzione. Oltre 30.000 geni compongono il DNA di una persona, una sorta di istruzioni per l uso per l organismo.
Il benessere passa dalla tua tavola Test Genetico per il metabolismo dell omocisteina. Introduzione. Oltre 30.000 geni compongono il DNA di una persona, una sorta di istruzioni per l uso per l organismo.
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
Replicazione Lewin, IL GENE VIII, Zanichelli editore S.p.A. Copyright 2006
 Replicazione Lewin, IL GENE VIII, Zanichelli editore S.p.A. Copyright 2006 Replicazione Ciclo cellulare Lewin, IL GENE VIII, Zanichelli editore S.p.A. Copyright 2006 Arthur Kornberg, 1970 Replicazione
Replicazione Lewin, IL GENE VIII, Zanichelli editore S.p.A. Copyright 2006 Replicazione Ciclo cellulare Lewin, IL GENE VIII, Zanichelli editore S.p.A. Copyright 2006 Arthur Kornberg, 1970 Replicazione
Nel 1997 un gruppo di ricercatori dell Università di Monaco, guidato. Genomi e genomica. DOMANDE CHIAVE Come si ottengono
 Genomi e genomica 14 DOMANDE CHIAVE Come si ottengono le sequenze del DNA genomico? Come viene decifrata l informazione contenuta nel genoma? Che cosa può rivelare la genomica comparata sulla struttura
Genomi e genomica 14 DOMANDE CHIAVE Come si ottengono le sequenze del DNA genomico? Come viene decifrata l informazione contenuta nel genoma? Che cosa può rivelare la genomica comparata sulla struttura
