Il sequenziamento del DNA
|
|
|
- Rachele Palmieri
- 8 anni fa
- Visualizzazioni
Transcript
1 Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile di praticamente qualunque tecnica di manipolazione genica Determinare la sequenza di una particolare regione di DNA può rappresentare un fine già di per sé (ad esempio se si voglia studiare una mutazione ereditaria) Ancora più importante è il fatto che le informazioni ottenute tramite il sequenziamento sono la base indispensabile per la messa in atto di qualsiasi procedura di manipolazione del DNA La conoscenza delle proprietà chimico-fisiche degli acidi nucleici ha reso possibile la messa a punto di due tecniche per la determinazione della sequenza del DNA L obiettivo principale della maggior parte dei progetti del genoma èdi determinare la sequenza del DNA dell intero genoma o quella di un numero elevato di trascritti Progetti di sequenziamento completo del genoma Microrganismi organuli >600 virus, >10 archaea, >50 batteri e diversi viroidi, plasmidi, e 24 genomi di organuli (completi) S. cerevisiae (completo) Animali Piante superiori Choenorhabditis elegans (in corso) Drosophyla melanogaster (in corso) Mus musculus (completo) Homo sapiens (completo) Arabidopsis thaliana (completo) Oryza sativa (completo) Medicago truncatula (in corso) 1
2 Le prime procedura per il sequenziamento del DNA vennero messe a punto da Maxam e Gilbert nel 1977 Basato sul trattamento con reagenti chimici capaci di degradare una molecola di DNA in corrispondenza di specifici nucelotidi Nel 1981 Sanger e collaboratori: Metodo Sanger o metodo dideossi Basato sulla sintesi enzimatica di molecole di DNA che terminano in corrispondenza di specifici nucelotidi Per entrambi i metodi la visualizzazione della sequenza avviene per elettroforesi in gel ad altissima risoluzione (si devono separare frammenti che differiscono per un singolo nucleotide) L idea che sta alla base del metodo Sanger è di produrre tutte le possibili molecole di DNA a filamento singolo, complementari a una sequenza stampo, che inizia con una base e che si estende fino a una di stanza di 1 kb in direzione 3. Reazione di polimerizzazione che si arresta specificamente dopo ogni G oppure dopo ogni A o T o C. L arresto specifico si ottiene aggiungendo alla reazione di polimerizzazione dei dideossinucleotidi trifosfati (ddntp) 2
3 In pratica si parte da una popolazione numerosa di molecole di DNA identiche ottenute per amplificazione enzimatica o batterica (E. coli) Prevede l uso di un primer complementare ad una sequenza terminale del frammento prodotto per PCR o ad una sequenza del DNA plasmidico adiacente al frammento clonato da sequenziare La reazione è catalizzata dalla DNA polimersai in direzione 5-3 a partire dall ossidrile contenuto nel primer e richiede i 4 nucleotidi (datp, dttp, dctp, dgtp) come precursori Il sequenziamento necessita anche di uno stampo di DNA e 4 differenti reazioni di sintesi in ciascuna delle quali c è un diverso ddntp Siccome la DNA polimerasi non è in grado di discriminare tra il ddntp e il corrispondente dntp ognuno dei possibili nucleotidi può essere inserito nella catena di DNA in allungamento 3
4 La separazione delle molecole in base alle loro dimensioni, ottenuta tramite elettroforesi, origina una serie di bande, ciascuna delle quali corrisponde a una classe di molecole che differisce per un nucleotide in più rispetto alla banda successiva Perché non ci sono due o più frammenti di una determinata lunghezza? Perchè il primer utilizzato è complementare soltanto alla sequenza che ci interessa (è progettato in modo da essere specifico) Direzione elettroforesi Direzione lettura A T C G 4
5 Risultato della corsa elettroforetica di un sequenziamento Evoluzione della tecnica Primers marcati con fluorocromi differenti ddatp ddctp ddgtp A AC ACG ddttp ACGT Le 4 reazioni possono correre insieme 5
6 Terminatori marcati ddatp ddctp ddgtp A AC ACG ddttp ACGT PCR Incorporazione dei terminatori marcati denaturazione appaiamento Miscela di reazione A C G T A C G T Prodotti marcati A C G T A estensione A C G T G A C G T 6
7 CCD Camera Laser Scanner Plates with acrylamide gel Elettroforesi capillare capillare Laser Camera CCD Buffer + _ 7
8 CCD (Chip Colour Detection) camera Lettura dei tracciati La lettura dei tracciati grezzi (non elaborati) viene anche detta identificazione delle basi (base-calling) è oggi effettuata tramite software che leggono automaticamente le basi, allinea le sequenze simili e fornisce una base intuitiva per la correzione di bozze (editing) 8
9 Mentre leggono i tracciati, i software assegnano punteggi di probabilità all accuratezza di identificazione di ciascuna base e le informazioni ottenute vengono usate nei passaggi successivi della procedura di allineamento errori Gli errori di sequenziamento possono essere provocati da problemi molto frequenti: Le prime 50 basi di una lettura sono indistinguibili dal rumore di fondo a causa della migrazione anomala di brevi frammenti di DNA che contengono agglomerati di coloranti 9
10 errori Inoltre i tracciati diventano progressivamente meno uniformi con l avanzare della corsa: aumentano gli effetti della diffusione molecolare e contemporaneamente diminuiscono le differenze relative di massa tra frammenti successivi Assemblaggio dei contig Lo stadio di rifinitura nel sequenziamento di un tratto di DNA più lungo di un singolo clone comprende l allineamento, il controllo e la correzione degli errori Questi passaggi sono in genere eseguiti con software di controllo delle sequenze (esempio il pacchetto Vector NTI) Le caratteristiche fondamentali sono: 1 - La capacità di visualizzare i tracciati di sequenze comparate tra di loro, di navigare tra di essi e segnalare con precisione le ambiguità (Conting express) 10
11 2 una visibilità facile e immediata del filamento complementare 3 gli strumenti per correggere manualmente la sequenza, come ad es. la possibilità di inserire o eliminare basi senza alterare in modo significativo i file dei tracciati originali 4 la capacità di individuare eventuali siti polimorfici 11
12 Il software produce, quindi, un interfaccia grafica che permette di richiamare e correggere in maniera interattiva le singole letture di contig Il compito finale di un programma di controllo delle sequenze è di aiutare a risolvere le lacune e le ambiguità 12
13 We sequenced 6 colonies for each RACE experiments We aligned the sequences by using the software VECTOR NTI The coding regions were mostly similar But..the 5 and 3 UTRs of some colonies were different This let us believe that for some genes we obtained more alleles at the same time 13
14 Also intriguing was the fact that the few differences were at the restriction sites of fragments... Mse-I Restriction site Sequenziamento del genoma Le sequenze dei cromosomi interi vengono ricostruite a partire dalle sequenze di centinaia di migliaia di frammenti di DNA, normalmente di lunghezza compresa fra 500 e 800 pb Si usano 2 strategie generali per la frammentazione e la ricostruzione: Il sequenziamento gerarchico Il sequenziamento a rosa di pallini (shotgun) Le due strategie sono reciprocamente complementari 14
15 Differenze: Nel sequenziamento gerarchico il prmo passaggio è quello di costruire un sentiero principale (tiling path) che serva ad assicurarsi che la sequenza sia ottenuta in grandi pezzi ordinati tra di loro Mentre nel metodo shotgun frammenta semplicemente il genoma in piccole unità sequenziabili e si affida ad algoritmi del computer per ricostruire l ordine dei frammenti Sequenziamento gerarchico (genoma umano) Le strategie gerarchiche conosciute anche come dall alto in basso (topdown), basate su mappe o clone per clone sono state ideate alla fine degli anni 80 quando i reagenti erano molto costosi e i computer non erano ancora abbastanza potenti per elaborare sequenze intere ottenute con lo shotgun Metodologia: frammentare il genoma in unità sempre più piccole le cui posizioni relative sono conosciute prima di iniziare il sequenziamento Vantaggi: favorisce la ricostruzione di mappe fisiche e genetiche ad alta risuluzione e permette a gruppi di lavoro di tutto il mondo di formare consorzi e lavorare insieme senza rischiare di ripetere le stesse ricerce (un gruppo = un cromosoma) 15
16 Il primo passaggio è quello di ottenere cloni del genoma umano di dimensioni comprese tra 50 e 200 kb I vettori in cui è possibile inserire tratti genomici di grandi dimensioni sono i BAC i PAC (derivati dal fago P1) Si costruiscono librerie di DNA tramite digestione parziale o frammentazione del DNA genomico tramite sonicazione In genere si desidera un certo grado di ridondanza Questo perché i cloni in teoria dovrebbero avere estremità diverse e questo dovrebbe rendere possibile selezionare una impalcatura di cloni che formino una sequenza contigua comprendente l intero cromosoma un tiling path 16
17 È possibile ricostruire un tiling path mediante una combinazione di 3 metodi: Ibridazione: è possibile identificare rapidamente in una libraria tutti i cloni che contengono una particolare sequenza, ibridando una sonda di piccole dimensioni, marcata con isotopi radioattivi o molecole fluorescenti, e contenente la sequenza, a un filtro su cui è fissata una combinazione di decine di migliaia di cloni In seguito si può usare l estremità di questi cloni come sonda per individuare i cloni adiacenti in quello che viene denominato chromosome walking 17
18 Fingerprinting: il modo meno costoso e più efficace per ricostruire l ordine dei contig di grandi cloni inseriti nei vettori è quello di confrontarli tra loro e allinearli in base ai profili di digestione con enzimi di restrizione Se si usa un enzima esacutter, questo opererà un taglio ogni 4 kb: se sono usati per tagliare un BAC di 100 kb, producono a 20 a 30 frammenti. Questi possono essere separati tramite elettroforesi e, quindi, assegnati a gruppi dimensionali, dopo una adeguata normalizzazione statistica 18
19 End-sequencing=sequenziare le estremità della raccolta di cloni BAC. Modo comune per identificare i cloni che si sovrappongono alle lacune rimanenti dopo il fingerprinting Una volta raggiunta una soglia critica di ricostruzione della sequenza, vi è una probabilità elevata che almeno un BAC si trovi all interno di una regione già ricostruita: di conseguenza l altra estremità del clone si estenderà nella lacuna o si collegherà ad un contig adiacente Il sequenziamento a una estremità è inoltre un componente importante delle tecniche usate per verificare la correttezza della ricostruzione delle sequenze. Tornando al sequenziamento gerarchico. Una volta scelto il tiling path, i singoli cloni BAC sono suddivisi in piccoli frammenti, che sono clonati a loro volta per il sequenziamento automatico La frammentazione viene fatta con ultrasuoni e questo assicura che ogni frammento abbia estremità diverse dagli altri 19
20 Sequenziamento shotgun Nel sequenziamento shotgun si usano algoritmi del computer per ricostruire la sequenza dei contig derivati da migliaia di cloni sovrapposti. I contig sono prodotti da una libreria plasmidica costruita a partire da un unico genoma intero Come nel sequenziamento gerarchico lo scopo è quello di ottenere una ridondanza di ciascun frammento del genoma da 5 a 10 volte L allineamento serve per ottenere l assemblaggio delle sequenze contigue di cloni, ma anche per migliorare l accuratezza della sequenza, tramite la produzione di una sequenza consenso Ad esempio nel genoma umano la Celera ha utilizzato 5 algoritmi con l obiettivo di riunire insieme il più possibile le parti dell unica sequenza sovrapponendo tra loro le sequenze parziali Così facendo le uniche lacune che restano sono quelle dovute a DNA ripetitivo o a sequenze non rappresentate nella libreria 20
21 Sequenziamento di interi genomi Frammento cromosomico sequenziato DNA genomico Frammentazione Assemblaggio dei frammenti sequenziati contigui (Software appositi) Clonaggio in vettori Sequenziamento 21
22 Verifica delle sequenze La veridicità di qualunque sequenza di un intero genoma deve essere valutata a tre livelli: la completezza, l accuratezza della sequenza di basi e la validità della ricostruzione. Completezza: in genere i microrganismi (così pure l uomo, il riso, arabidopsis e il topo) sono stati sequenziati per intero Possono contenere comunque lacune difficili da colmare (di circa 1 kb). Questo può essere dovuto a sequenze ripetute difficili da colmare o al fatto che il sequenziamento è stato effettuato partendo da individui diversi per cui potrebbero esserci polimorfismo dovuti a indel o a duplicazioni Accuratezza: l accuratezza è stabilita in punteggi di probabilità Validità della ricostruzione: non è facilmente determinabile: è possibile averne un idea approssimativa misurando al coerenza interna della sequenza, oppure paragonando la ricostruzione con mappe genetiche o fisiche preesisteti o fatte all uopo. 22
23 Chromosome painting con cloni BAC 12.7 Mb CEN 14.4 Mb Condizioni di Painting 1. Chromosoma 1 di Arabidopsis thaliana 2. Un totale di 183 cloni BAC del chr-1 3. BAC dal braccio superiore (12.7 MB) marcati con biotin-dutp (rosso) 4. BACs dal braccio inferiore (14.4 MB) marcati con digoxygenin-dutp (verde) FISH painting del cromosoma 4 con 131 BAC 23
24 WS (knob)? C24 (knobless) * F9H3 F4C21 24
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Database. Si ringrazia Marco Bertini per le slides
 Database Si ringrazia Marco Bertini per le slides Obiettivo Concetti base dati e informazioni cos è un database terminologia Modelli organizzativi flat file database relazionali Principi e linee guida
Database Si ringrazia Marco Bertini per le slides Obiettivo Concetti base dati e informazioni cos è un database terminologia Modelli organizzativi flat file database relazionali Principi e linee guida
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Immagine di una sequenza ben riuscita. Sequences troubleshooting
 Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
MODULO 5 Appunti ACCESS - Basi di dati
 MODULO 5 Appunti ACCESS - Basi di dati Lezione 1 www.mondopcnet.com Modulo 5 basi di dati Richiede che il candidato dimostri di possedere la conoscenza relativa ad alcuni concetti fondamentali sui database.
MODULO 5 Appunti ACCESS - Basi di dati Lezione 1 www.mondopcnet.com Modulo 5 basi di dati Richiede che il candidato dimostri di possedere la conoscenza relativa ad alcuni concetti fondamentali sui database.
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Lezioni di biotecnologie
 Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Tecniche di sequenziamento del DNA
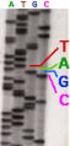 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
1- OBIETTIVI DEL DOCUMENTO 2- INTRODUZIONE
 1- OBIETTIVI DEL DOCUMENTO... 1 2- INTRODUZIONE... 1 3- ACCESSO ALLA PROCEDURA... 2 4- COMPILAZIONE ON-LINE... 4 5- SCELTA DELLA REGIONE O PROVINCIA AUTONOMA... 5 6- DATI ANAGRAFICI... 6 7- DATI ANAGRAFICI
1- OBIETTIVI DEL DOCUMENTO... 1 2- INTRODUZIONE... 1 3- ACCESSO ALLA PROCEDURA... 2 4- COMPILAZIONE ON-LINE... 4 5- SCELTA DELLA REGIONE O PROVINCIA AUTONOMA... 5 6- DATI ANAGRAFICI... 6 7- DATI ANAGRAFICI
Sistema di diagnosi CAR TEST
 Data: 30/09/09 1 di 7 Sistema di diagnosi CAR TEST Il sistema di diagnosi CAR TEST venne convenientemente utilizzato per: - verificare che la scocca di un veicolo sia dimensionalmente conforme ai disegni
Data: 30/09/09 1 di 7 Sistema di diagnosi CAR TEST Il sistema di diagnosi CAR TEST venne convenientemente utilizzato per: - verificare che la scocca di un veicolo sia dimensionalmente conforme ai disegni
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
Parte I. Prima Parte
 Parte I Prima Parte Capitolo 1 Introduzione generale 1.1 Il problema dell assegnazione Corsi-Borsisti Il problema dell assegnazione delle borse dei corsi ai vari studenti può essere riassunto nei punti
Parte I Prima Parte Capitolo 1 Introduzione generale 1.1 Il problema dell assegnazione Corsi-Borsisti Il problema dell assegnazione delle borse dei corsi ai vari studenti può essere riassunto nei punti
Excel avanzato. I nomi. Gli indirizzi e le formule possono essere sostituiti da nomi. Si creano tramite Inserisci Nome Definisci
 Excel avanzato I nomi marco.falda@unipd.it Gli indirizzi e le formule possono essere sostituiti da nomi documentazione astrazione Si creano tramite Inserisci Nome Definisci Vengono raccolti nell area riferimento
Excel avanzato I nomi marco.falda@unipd.it Gli indirizzi e le formule possono essere sostituiti da nomi documentazione astrazione Si creano tramite Inserisci Nome Definisci Vengono raccolti nell area riferimento
Agenda telematica delle manifestazioni pubbliche
 Prefettura Ufficio territoriale del Governo di Campobasso Università degli Studi del Molise Agenda telematica delle manifestazioni pubbliche Manuale Utente : Personale Ente Organizzatore Sommario 1. Introduzione
Prefettura Ufficio territoriale del Governo di Campobasso Università degli Studi del Molise Agenda telematica delle manifestazioni pubbliche Manuale Utente : Personale Ente Organizzatore Sommario 1. Introduzione
Sistema operativo. Sommario. Sistema operativo...1 Browser...1. Convenzioni adottate
 MODULO BASE Quanto segue deve essere rispettato se si vuole che le immagini presentate nei vari moduli corrispondano, con buona probabilità, a quanto apparirà nello schermo del proprio computer nel momento
MODULO BASE Quanto segue deve essere rispettato se si vuole che le immagini presentate nei vari moduli corrispondano, con buona probabilità, a quanto apparirà nello schermo del proprio computer nel momento
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20
 SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
CMS ERMES INFORMATICA
 01/07/2014 Guida di riferimento alla pubblicazione di contenuti sul portale Il presente documento costituisce una guida di riferimento all aggiornamento e alla pubblicazione di contenuti sui portali web
01/07/2014 Guida di riferimento alla pubblicazione di contenuti sul portale Il presente documento costituisce una guida di riferimento all aggiornamento e alla pubblicazione di contenuti sui portali web
CORSO ACCESS PARTE II. Esistono diversi tipi di aiuto forniti con Access, generalmente accessibili tramite la barra dei menu (?)
 Ambiente Access La Guida di Access Esistono diversi tipi di aiuto forniti con Access, generalmente accessibili tramite la barra dei menu (?) Guida in linea Guida rapida Assistente di Office indicazioni
Ambiente Access La Guida di Access Esistono diversi tipi di aiuto forniti con Access, generalmente accessibili tramite la barra dei menu (?) Guida in linea Guida rapida Assistente di Office indicazioni
GHPPEditor è un software realizzato per produrre in modo rapido e guidato un part program per controlli numerici Heidenhain.
 *+33(GLWRU GHPPEditor è un software realizzato per produrre in modo rapido e guidato un part program per controlli numerici Heidenhain. Il programma si basa su un architettura di tasti funzionali presenti
*+33(GLWRU GHPPEditor è un software realizzato per produrre in modo rapido e guidato un part program per controlli numerici Heidenhain. Il programma si basa su un architettura di tasti funzionali presenti
Aris TimeSheet. che guardano oltre. enti e aziende. Soluzioni per
 Aris TimeSheet Soluzioni per enti e aziende che guardano oltre L applicativo ARIS TIMESHEET è stato progettato e sviluppato per supportare i project manager nel monitoraggio dello stato di avanzamento
Aris TimeSheet Soluzioni per enti e aziende che guardano oltre L applicativo ARIS TIMESHEET è stato progettato e sviluppato per supportare i project manager nel monitoraggio dello stato di avanzamento
Manuale Utente Albo Pretorio GA
 Manuale Utente Albo Pretorio GA IDENTIFICATIVO DOCUMENTO MU_ALBOPRETORIO-GA_1.4 Versione 1.4 Data edizione 04.04.2013 1 TABELLA DELLE VERSIONI Versione Data Paragrafo Descrizione delle modifiche apportate
Manuale Utente Albo Pretorio GA IDENTIFICATIVO DOCUMENTO MU_ALBOPRETORIO-GA_1.4 Versione 1.4 Data edizione 04.04.2013 1 TABELLA DELLE VERSIONI Versione Data Paragrafo Descrizione delle modifiche apportate
Biotecnologie ed OGM. Prima parte: DNA ricombinante e microorganismi geneticamente modificati.
 Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
ShellExcel. Una domanda contiene i riferimenti (#A, #B, #C) alle celle che contengono i dati numerici del
 Progetto Software to Fit - ShellExcel Pagina 1 Manuale d'uso ShellExcel ShellExcel è una interfaccia per disabili che permette ad un alunno con difficoltà di apprendimento di esercitarsi ripetitivamente
Progetto Software to Fit - ShellExcel Pagina 1 Manuale d'uso ShellExcel ShellExcel è una interfaccia per disabili che permette ad un alunno con difficoltà di apprendimento di esercitarsi ripetitivamente
Come modificare la propria Home Page e gli elementi correlati
 Come modificare la propria Home Page e gli elementi correlati Versione del documento: 3.0 Ultimo aggiornamento: 2006-09-15 Riferimento: webmaster (webmaster.economia@unimi.it) La modifica delle informazioni
Come modificare la propria Home Page e gli elementi correlati Versione del documento: 3.0 Ultimo aggiornamento: 2006-09-15 Riferimento: webmaster (webmaster.economia@unimi.it) La modifica delle informazioni
Elementi di Psicometria con Laboratorio di SPSS 1
 Elementi di Psicometria con Laboratorio di SPSS 1 12-Il t-test per campioni appaiati vers. 1.2 (7 novembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
Elementi di Psicometria con Laboratorio di SPSS 1 12-Il t-test per campioni appaiati vers. 1.2 (7 novembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
per immagini guida avanzata Organizzazione e controllo dei dati Geometra Luigi Amato Guida Avanzata per immagini excel 2000 1
 Organizzazione e controllo dei dati Geometra Luigi Amato Guida Avanzata per immagini excel 2000 1 Il raggruppamento e la struttura dei dati sono due funzioni di gestione dati di Excel, molto simili tra
Organizzazione e controllo dei dati Geometra Luigi Amato Guida Avanzata per immagini excel 2000 1 Il raggruppamento e la struttura dei dati sono due funzioni di gestione dati di Excel, molto simili tra
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona
 Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Approccio stratificato
 Approccio stratificato Il sistema operativo è suddiviso in strati (livelli), ciascuno costruito sopra quelli inferiori. Il livello più basso (strato 0) è l hardware, il più alto (strato N) è l interfaccia
Approccio stratificato Il sistema operativo è suddiviso in strati (livelli), ciascuno costruito sopra quelli inferiori. Il livello più basso (strato 0) è l hardware, il più alto (strato N) è l interfaccia
Manuale Operativo per l utilizzo della piattaforma E-Learning@AQ. Versione 1.1
 Manuale Operativo per l utilizzo della piattaforma E-Learning@AQ Versione 1.1 Autore Antonio Barbieri, antonio.barbieri@gmail.com Data inizio compilazione 11 maggio 2009 Data revisione 14 maggio 2009 Sommario
Manuale Operativo per l utilizzo della piattaforma E-Learning@AQ Versione 1.1 Autore Antonio Barbieri, antonio.barbieri@gmail.com Data inizio compilazione 11 maggio 2009 Data revisione 14 maggio 2009 Sommario
Funzioni in C. Violetta Lonati
 Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Università degli studi di Milano Dipartimento di Scienze dell Informazione Laboratorio di algoritmi e strutture dati Corso di laurea in Informatica Funzioni - in breve: Funzioni Definizione di funzioni
Gestione delle informazioni necessarie all attività di validazione degli studi di settore. Trasmissione degli esempi da valutare.
 Gestione delle informazioni necessarie all attività di validazione degli studi di settore. Trasmissione degli esempi da valutare. E stato previsto l utilizzo di uno specifico prodotto informatico (denominato
Gestione delle informazioni necessarie all attività di validazione degli studi di settore. Trasmissione degli esempi da valutare. E stato previsto l utilizzo di uno specifico prodotto informatico (denominato
Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16
 Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16 L'immunoistochimica e' una tecnica ampiamente utilizzata per l'identificazione e la localizzazione di costituenti cellulari
Laboratorio di Tecniche Microscopiche AA 2007-2008 Lezione 12 Marzo 2008 Ore 15-16 L'immunoistochimica e' una tecnica ampiamente utilizzata per l'identificazione e la localizzazione di costituenti cellulari
Sequenziamento e analisi di genomi completi
 Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Avanzamento dei sistemi di sequenziamento
 Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
PULSANTI E PAGINE Sommario PULSANTI E PAGINE...1
 Pagina 1 Sommario...1 Apertura...2 Visualizzazioni...2 Elenco...2 Testo sul pulsante e altre informazioni...3 Comandi...3 Informazioni...4 Flow chart...5 Comandi...6 Pulsanti Principali e Pulsanti Dipendenti...6
Pagina 1 Sommario...1 Apertura...2 Visualizzazioni...2 Elenco...2 Testo sul pulsante e altre informazioni...3 Comandi...3 Informazioni...4 Flow chart...5 Comandi...6 Pulsanti Principali e Pulsanti Dipendenti...6
Il sofware è inoltre completato da una funzione di calendario che consente di impostare in modo semplice ed intuitivo i vari appuntamenti.
 SH.MedicalStudio Presentazione SH.MedicalStudio è un software per la gestione degli studi medici. Consente di gestire un archivio Pazienti, con tutti i documenti necessari ad avere un quadro clinico completo
SH.MedicalStudio Presentazione SH.MedicalStudio è un software per la gestione degli studi medici. Consente di gestire un archivio Pazienti, con tutti i documenti necessari ad avere un quadro clinico completo
Automazione Industriale (scheduling+mms) scheduling+mms. adacher@dia.uniroma3.it
 Automazione Industriale (scheduling+mms) scheduling+mms adacher@dia.uniroma3.it Introduzione Sistemi e Modelli Lo studio e l analisi di sistemi tramite una rappresentazione astratta o una sua formalizzazione
Automazione Industriale (scheduling+mms) scheduling+mms adacher@dia.uniroma3.it Introduzione Sistemi e Modelli Lo studio e l analisi di sistemi tramite una rappresentazione astratta o una sua formalizzazione
SOFTWARE PER LA RILEVAZIONE DEI TEMPI PER CENTRI DI COSTO
 SOFTWARE PER LA RILEVAZIONE DEI TEMPI PER CENTRI DI COSTO Descrizione Nell ambito della rilevazione dei costi, Solari con l ambiente Start propone Time&Cost, una applicazione che contribuisce a fornire
SOFTWARE PER LA RILEVAZIONE DEI TEMPI PER CENTRI DI COSTO Descrizione Nell ambito della rilevazione dei costi, Solari con l ambiente Start propone Time&Cost, una applicazione che contribuisce a fornire
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
MANUALE MOODLE STUDENTI. Accesso al Materiale Didattico
 MANUALE MOODLE STUDENTI Accesso al Materiale Didattico 1 INDICE 1. INTRODUZIONE ALLA PIATTAFORMA MOODLE... 3 1.1. Corso Moodle... 4 2. ACCESSO ALLA PIATTAFORMA... 7 2.1. Accesso diretto alla piattaforma...
MANUALE MOODLE STUDENTI Accesso al Materiale Didattico 1 INDICE 1. INTRODUZIONE ALLA PIATTAFORMA MOODLE... 3 1.1. Corso Moodle... 4 2. ACCESSO ALLA PIATTAFORMA... 7 2.1. Accesso diretto alla piattaforma...
PIANO BIENNALE PER I DIRITTI DELLE PERSONE CON DISABILITÀ
 PIANO BIENNALE PER I DIRITTI DELLE PERSONE CON DISABILITÀ 15 novembre 2014 Daniela Sangiovanni Comunicazione e Ufficio stampa Policlinico S,Orsola ACCESSIBILITÀ Gli Stati dovrebbero riconoscere l importanza
PIANO BIENNALE PER I DIRITTI DELLE PERSONE CON DISABILITÀ 15 novembre 2014 Daniela Sangiovanni Comunicazione e Ufficio stampa Policlinico S,Orsola ACCESSIBILITÀ Gli Stati dovrebbero riconoscere l importanza
2.0 Gli archivi. 2.1 Inserire gli archivi. 2.2 Archivio Clienti, Fornitori, Materiali, Noleggi ed Altri Costi. Impresa Edile Guida all uso
 2.0 Gli archivi All interno della sezione archivi sono inserite le anagrafiche. In pratica si stratta di tutti quei dati che ricorreranno costantemente all interno dei documenti. 2.1 Inserire gli archivi
2.0 Gli archivi All interno della sezione archivi sono inserite le anagrafiche. In pratica si stratta di tutti quei dati che ricorreranno costantemente all interno dei documenti. 2.1 Inserire gli archivi
Amministrazione gruppi (Comunità)
 Amministrazione gruppi (Comunità) Guida breve per il docente che amministra il gruppo Premessa Di regola i gruppi sono creati all interno della Scuola. Nel caso in cui vi fosse la necessità di aprire un
Amministrazione gruppi (Comunità) Guida breve per il docente che amministra il gruppo Premessa Di regola i gruppi sono creati all interno della Scuola. Nel caso in cui vi fosse la necessità di aprire un
Corso di. Dott.ssa Donatella Cocca
 Corso di Statistica medica e applicata Dott.ssa Donatella Cocca 1 a Lezione Cos'è la statistica? Come in tutta la ricerca scientifica sperimentale, anche nelle scienze mediche e biologiche è indispensabile
Corso di Statistica medica e applicata Dott.ssa Donatella Cocca 1 a Lezione Cos'è la statistica? Come in tutta la ricerca scientifica sperimentale, anche nelle scienze mediche e biologiche è indispensabile
LA REVISIONE LEGALE DEI CONTI La comprensione
 LA REVISIONE LEGALE DEI CONTI La comprensione dell impresa e del suo contesto e la valutazione dei rischi di errori significativi Ottobre 2013 Indice 1. La comprensione dell impresa e del suo contesto
LA REVISIONE LEGALE DEI CONTI La comprensione dell impresa e del suo contesto e la valutazione dei rischi di errori significativi Ottobre 2013 Indice 1. La comprensione dell impresa e del suo contesto
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
lo 2 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000
 Capittol lo 2 Visualizzazione 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000 Nel primo capitolo sono state analizzate le diverse componenti della finestra di Word 2000: barra del titolo, barra dei menu,
Capittol lo 2 Visualizzazione 2-1 - PERSONALIZZARE LA FINESTRA DI WORD 2000 Nel primo capitolo sono state analizzate le diverse componenti della finestra di Word 2000: barra del titolo, barra dei menu,
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
MANUALE PARCELLA FACILE PLUS INDICE
 MANUALE PARCELLA FACILE PLUS INDICE Gestione Archivi 2 Configurazioni iniziali 3 Anagrafiche 4 Creazione prestazioni e distinta base 7 Documenti 9 Agenda lavori 12 Statistiche 13 GESTIONE ARCHIVI Nella
MANUALE PARCELLA FACILE PLUS INDICE Gestione Archivi 2 Configurazioni iniziali 3 Anagrafiche 4 Creazione prestazioni e distinta base 7 Documenti 9 Agenda lavori 12 Statistiche 13 GESTIONE ARCHIVI Nella
Circuiti amplificatori
 Circuiti amplificatori G. Traversi Strumentazione e Misure Elettroniche Corso Integrato di Elettrotecnica e Strumentazione e Misure Elettroniche 1 Amplificatori 2 Amplificatori Se A V è negativo, l amplificatore
Circuiti amplificatori G. Traversi Strumentazione e Misure Elettroniche Corso Integrato di Elettrotecnica e Strumentazione e Misure Elettroniche 1 Amplificatori 2 Amplificatori Se A V è negativo, l amplificatore
Progetto SINTESI - Dominio Provinciale
 - Dominio Provinciale Tecnologia dei Processi R.T.I. per Pag. 2 di 19 Indice 1 INTRODUZIONE... 3 2 LETTURA DEL FILE... 4 3 IMPORT DEI FILE... 9 4 VERIFICA DELLE BOZZE E LORO INVIO... 12 5 COMUNICAZIONI
- Dominio Provinciale Tecnologia dei Processi R.T.I. per Pag. 2 di 19 Indice 1 INTRODUZIONE... 3 2 LETTURA DEL FILE... 4 3 IMPORT DEI FILE... 9 4 VERIFICA DELLE BOZZE E LORO INVIO... 12 5 COMUNICAZIONI
Altri metodi di indicizzazione
 Organizzazione a indici su più livelli Altri metodi di indicizzazione Al crescere della dimensione del file l organizzazione sequenziale a indice diventa inefficiente: in lettura a causa del crescere del
Organizzazione a indici su più livelli Altri metodi di indicizzazione Al crescere della dimensione del file l organizzazione sequenziale a indice diventa inefficiente: in lettura a causa del crescere del
Gestione ed analisi di base dati nell epidemiologia. delle malattie infettive
 Università degli Studi di Torino - Facoltà di Medicina Veterinaria Laboratorio di epidemiologia delle malattie infettive Scuola Specializzazione in Sanità Animale, Allevamento e Produzioni Zootecniche
Università degli Studi di Torino - Facoltà di Medicina Veterinaria Laboratorio di epidemiologia delle malattie infettive Scuola Specializzazione in Sanità Animale, Allevamento e Produzioni Zootecniche
Soluzione dell esercizio del 2 Febbraio 2004
 Soluzione dell esercizio del 2 Febbraio 2004 1. Casi d uso I casi d uso sono riportati in Figura 1. Figura 1: Diagramma dei casi d uso. E evidenziato un sotto caso di uso. 2. Modello concettuale Osserviamo
Soluzione dell esercizio del 2 Febbraio 2004 1. Casi d uso I casi d uso sono riportati in Figura 1. Figura 1: Diagramma dei casi d uso. E evidenziato un sotto caso di uso. 2. Modello concettuale Osserviamo
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Archivi e database. Prof. Michele Batocchi A.S. 2013/2014
 Archivi e database Prof. Michele Batocchi A.S. 2013/2014 Introduzione L esigenza di archiviare (conservare documenti, immagini, ricordi, ecc.) è un attività senza tempo che è insita nell animo umano Primi
Archivi e database Prof. Michele Batocchi A.S. 2013/2014 Introduzione L esigenza di archiviare (conservare documenti, immagini, ricordi, ecc.) è un attività senza tempo che è insita nell animo umano Primi
MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013]
![MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013] MOCA. Modulo Candidatura. http://www.federscacchi.it/moca. moca@federscacchi.it. [Manuale versione 1.0 marzo 2013]](/thumbs/25/5308947.jpg) MOCA Modulo Candidatura http://www.federscacchi.it/moca moca@federscacchi.it [Manuale versione 1.0 marzo 2013] 1/12 MOCA in breve MOCA è una funzionalità del sito web della FSI che permette di inserire
MOCA Modulo Candidatura http://www.federscacchi.it/moca moca@federscacchi.it [Manuale versione 1.0 marzo 2013] 1/12 MOCA in breve MOCA è una funzionalità del sito web della FSI che permette di inserire
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
SISTEMI DI NUMERAZIONE E CODICI
 SISTEMI DI NUMERAZIONE E CODICI Il Sistema di Numerazione Decimale Il sistema decimale o sistema di numerazione a base dieci usa dieci cifre, dette cifre decimali, da O a 9. Il sistema decimale è un sistema
SISTEMI DI NUMERAZIONE E CODICI Il Sistema di Numerazione Decimale Il sistema decimale o sistema di numerazione a base dieci usa dieci cifre, dette cifre decimali, da O a 9. Il sistema decimale è un sistema
Sostituto abilitato Entratel con più sedi: ricezione diretta e incarico ad intermediario abilitato
 FAQ Flusso telematico dei modelli 730-4 D.M. 31 maggio 1999, n. 164 Comunicazione dei sostituti d imposta per la ricezione telematica, tramite l Agenzia delle entrate, dei dati dei 730-4 relativi ai mod.
FAQ Flusso telematico dei modelli 730-4 D.M. 31 maggio 1999, n. 164 Comunicazione dei sostituti d imposta per la ricezione telematica, tramite l Agenzia delle entrate, dei dati dei 730-4 relativi ai mod.
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
Metodologie di programmazione in Fortran 90
 Metodologie di programmazione in Fortran 90 Ing. Luca De Santis DIS - Dipartimento di informatica e sistemistica Anno accademico 2007/2008 Fortran 90: Metodologie di programmazione DIS - Dipartimento di
Metodologie di programmazione in Fortran 90 Ing. Luca De Santis DIS - Dipartimento di informatica e sistemistica Anno accademico 2007/2008 Fortran 90: Metodologie di programmazione DIS - Dipartimento di
Sistema GEFO. Guida all utilizzo Presentazione delle domande
 Sistema GEFO Guida all utilizzo Presentazione delle domande INDICE 1 LA PRESENTAZIONE DELLA DOMANDA DI CONTRIBUTO...3 1.1 L AREA BANDI: INSERIMENTO DI UN NUOVO PROGETTO...3 1.1.1 Il modulo progetto...12
Sistema GEFO Guida all utilizzo Presentazione delle domande INDICE 1 LA PRESENTAZIONE DELLA DOMANDA DI CONTRIBUTO...3 1.1 L AREA BANDI: INSERIMENTO DI UN NUOVO PROGETTO...3 1.1.1 Il modulo progetto...12
Progettazione : Design Pattern Creazionali
 Progettazione : Design Pattern Creazionali Alessandro Martinelli alessandro.martinelli@unipv.it 30 Novembre 2010 Progettazione : Design Pattern Creazionali Aspetti generali dei Design Pattern Creazionali
Progettazione : Design Pattern Creazionali Alessandro Martinelli alessandro.martinelli@unipv.it 30 Novembre 2010 Progettazione : Design Pattern Creazionali Aspetti generali dei Design Pattern Creazionali
MODELLO UNICO DI DOMANDA
 Fascicolo aziendale MODELLO UNICO DI DOMANDA Le imprese agricole iscritte all anagrafe delle imprese dispongono di un fascicolo aziendale, costituito da un insieme di informazioni generali relative all
Fascicolo aziendale MODELLO UNICO DI DOMANDA Le imprese agricole iscritte all anagrafe delle imprese dispongono di un fascicolo aziendale, costituito da un insieme di informazioni generali relative all
DNA - RNA. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi.
 DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
Librerie digitali. Video. Gestione di video. Caratteristiche dei video. Video. Metadati associati ai video. Metadati associati ai video
 Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
MANUALE UTENTE Fiscali Free
 MANUALE UTENTE Fiscali Free Le informazioni contenute in questa pubblicazione sono soggette a modifiche da parte della ComputerNetRimini. Il software descritto in questa pubblicazione viene rilasciato
MANUALE UTENTE Fiscali Free Le informazioni contenute in questa pubblicazione sono soggette a modifiche da parte della ComputerNetRimini. Il software descritto in questa pubblicazione viene rilasciato
LE MOLECOLE INFORMAZIONALI. Lezioni d'autore Treccani
 LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
Come archiviare i dati per le scienze sociali
 Come archiviare i dati per le scienze sociali ADPSS-SOCIODATA Archivio Dati e Programmi per le Scienze Sociali www.sociologiadip.unimib.it/sociodata E-mail: adpss.sociologia@unimib.it Tel.: 02 64487513
Come archiviare i dati per le scienze sociali ADPSS-SOCIODATA Archivio Dati e Programmi per le Scienze Sociali www.sociologiadip.unimib.it/sociodata E-mail: adpss.sociologia@unimib.it Tel.: 02 64487513
Elementi di Psicometria con Laboratorio di SPSS 1
 Elementi di Psicometria con Laboratorio di SPSS 1 29-Analisi della potenza statistica vers. 1.0 (12 dicembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
Elementi di Psicometria con Laboratorio di SPSS 1 29-Analisi della potenza statistica vers. 1.0 (12 dicembre 2014) Germano Rossi 1 germano.rossi@unimib.it 1 Dipartimento di Psicologia, Università di Milano-Bicocca
I MODULI Q.A.T. PANORAMICA. La soluzione modulare di gestione del Sistema Qualità Aziendale
 La soluzione modulare di gestione del Sistema Qualità Aziendale I MODULI Q.A.T. - Gestione clienti / fornitori - Gestione strumenti di misura - Gestione verifiche ispettive - Gestione documentazione del
La soluzione modulare di gestione del Sistema Qualità Aziendale I MODULI Q.A.T. - Gestione clienti / fornitori - Gestione strumenti di misura - Gestione verifiche ispettive - Gestione documentazione del
LA MISURAZIONE DEL CARATTERE
 TPO PROGETTAZIONE UD 03 GESTIONE DEL CARATTERE IL TIPOMETRO LA MISURAZIONE DEL CARATTERE A.F. 2011/2012 MASSIMO FRANCESCHINI - SILVIA CAVARZERE 1 IL TIPOMETRO: PARTI FONDAMENTALI Il tipometro è uno strumento
TPO PROGETTAZIONE UD 03 GESTIONE DEL CARATTERE IL TIPOMETRO LA MISURAZIONE DEL CARATTERE A.F. 2011/2012 MASSIMO FRANCESCHINI - SILVIA CAVARZERE 1 IL TIPOMETRO: PARTI FONDAMENTALI Il tipometro è uno strumento
GESTIONE FATTURE (VELINE)
 GESTIONE FATTURE (VELINE) Sommario Importazione Veline da Excel... 2 Generazione di una Velina da uno o più Ordini... 3 Creazione di una Velina prelevando i prodotti da più Ordini... 4 Gestione scadenze
GESTIONE FATTURE (VELINE) Sommario Importazione Veline da Excel... 2 Generazione di una Velina da uno o più Ordini... 3 Creazione di una Velina prelevando i prodotti da più Ordini... 4 Gestione scadenze
Guida alla registrazione on-line di un DataLogger
 NovaProject s.r.l. Guida alla registrazione on-line di un DataLogger Revisione 3.0 3/08/2010 Partita IVA / Codice Fiscale: 03034090542 pag. 1 di 17 Contenuti Il presente documento è una guida all accesso
NovaProject s.r.l. Guida alla registrazione on-line di un DataLogger Revisione 3.0 3/08/2010 Partita IVA / Codice Fiscale: 03034090542 pag. 1 di 17 Contenuti Il presente documento è una guida all accesso
Riconoscibilità dei siti pubblici: i domini della Pa e le regole di.gov.it
 Riconoscibilità dei siti pubblici: i domini della Pa e le regole di.gov.it Gabriella Calderisi - DigitPA 2 dicembre 2010 Dicembre 2010 Dominio.gov.it Cos è un dominio? Se Internet è una grande città, i
Riconoscibilità dei siti pubblici: i domini della Pa e le regole di.gov.it Gabriella Calderisi - DigitPA 2 dicembre 2010 Dicembre 2010 Dominio.gov.it Cos è un dominio? Se Internet è una grande città, i
Organizzazione degli archivi
 COSA E UN DATA-BASE (DB)? è l insieme di dati relativo ad un sistema informativo COSA CARATTERIZZA UN DB? la struttura dei dati le relazioni fra i dati I REQUISITI DI UN DB SONO: la ridondanza minima i
COSA E UN DATA-BASE (DB)? è l insieme di dati relativo ad un sistema informativo COSA CARATTERIZZA UN DB? la struttura dei dati le relazioni fra i dati I REQUISITI DI UN DB SONO: la ridondanza minima i
Uso di base delle funzioni in Microsoft Excel
 Uso di base delle funzioni in Microsoft Excel Le funzioni Una funzione è un operatore che applicato a uno o più argomenti (valori, siano essi numeri con virgola, numeri interi, stringhe di caratteri) restituisce
Uso di base delle funzioni in Microsoft Excel Le funzioni Una funzione è un operatore che applicato a uno o più argomenti (valori, siano essi numeri con virgola, numeri interi, stringhe di caratteri) restituisce
Procedura Import tracciato ministeriale
 Progetto SINTESI Dominio Provinciale Modulo Applicativo:COB Procedura Import tracciato ministeriale 1 INDICE 1 INTRODUZIONE... 3 2 LETTURA DEL FILE... 4 3 IMPORT DEI FILE... 10 4 VERIFICA DELLE BOZZE E
Progetto SINTESI Dominio Provinciale Modulo Applicativo:COB Procedura Import tracciato ministeriale 1 INDICE 1 INTRODUZIONE... 3 2 LETTURA DEL FILE... 4 3 IMPORT DEI FILE... 10 4 VERIFICA DELLE BOZZE E
8-06-2010. BLOTTING degli acidi nucleici: RT-PCR SOUTHERN BLOTTING NORTHERN BLOTTING. BLOTTING delle proteine: NORTHERN BLOTTING SOUTHERN BLOTTING
 8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
UNIEMENS Manuale consultazione DM10 Virtuale e DM10VIG nel sito www.inps.it
 UNIEMENS Manuale consultazione DM10 Virtuale e DM10VIG nel sito www.inps.it Dalla home page del sito www.inps.it l utente deve accedere alla voce UniEMens (cerchiata nell immagine) Figura 1 Home page www.inps.it
UNIEMENS Manuale consultazione DM10 Virtuale e DM10VIG nel sito www.inps.it Dalla home page del sito www.inps.it l utente deve accedere alla voce UniEMens (cerchiata nell immagine) Figura 1 Home page www.inps.it
ColorSplitter. La separazione automatica dei colori di Colibri.. Perché ColorSplitter? Come opera ColorSplitter?
 ColorSplitter La separazione automatica dei colori di Colibri.. ColorSplitter è una nuova funzionalità aggiunta a Colibri, che permette di elaborare un immagine trasformandola in una separata in canali
ColorSplitter La separazione automatica dei colori di Colibri.. ColorSplitter è una nuova funzionalità aggiunta a Colibri, che permette di elaborare un immagine trasformandola in una separata in canali
NUOVA PROCEDURA COPIA ED INCOLLA PER L INSERIMENTO DELLE CLASSIFICHE NEL SISTEMA INFORMATICO KSPORT.
 NUOVA PROCEDURA COPIA ED INCOLLA PER L INSERIMENTO DELLE CLASSIFICHE NEL SISTEMA INFORMATICO KSPORT. Con l utilizzo delle procedure di iscrizione on line la società organizzatrice ha a disposizione tutti
NUOVA PROCEDURA COPIA ED INCOLLA PER L INSERIMENTO DELLE CLASSIFICHE NEL SISTEMA INFORMATICO KSPORT. Con l utilizzo delle procedure di iscrizione on line la società organizzatrice ha a disposizione tutti
Capitolo 4 Pianificazione e Sviluppo di Web Part
 Capitolo 4 Pianificazione e Sviluppo di Web Part Questo capitolo mostra come usare Microsoft Office XP Developer per personalizzare Microsoft SharePoint Portal Server 2001. Spiega come creare, aggiungere,
Capitolo 4 Pianificazione e Sviluppo di Web Part Questo capitolo mostra come usare Microsoft Office XP Developer per personalizzare Microsoft SharePoint Portal Server 2001. Spiega come creare, aggiungere,
Gestire immagini e grafica con Word 2010
 Gestire immagini e grafica con Word 2010 Con Word 2010 è possibile impaginare documenti in modo semi-professionale ottenendo risultati graficamente gradevoli. Inserire un immagine e gestire il testo Prima
Gestire immagini e grafica con Word 2010 Con Word 2010 è possibile impaginare documenti in modo semi-professionale ottenendo risultati graficamente gradevoli. Inserire un immagine e gestire il testo Prima
