La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria
|
|
|
- Arnoldo Randazzo
- 6 anni fa
- Visualizzazioni
Transcript
1 La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria Il 15% del DNA associato ai centromeri, costituito da sequenze altamente ripetute (DNA satellite e STR) Il 82% sequenze non codificanti per proteine (codificanti trna, rrna, microrna e sequenze regolatrici; pseudogeni, e regioni di DNA la cui funzione rimane ignota (junk DNA) solo 1,5-3% del DNA da origine a proteine
2 Il genoma umano 3,2 miliardi di paia di basi di DNA geni 23 coppie di cromosomi
3 Definizione di gene un gene è una parte di DNA che porta tutte le informazioni necessarie per lo sviluppo di un carattere (segmento di DNA che codifica per una proteina) occupa sul cromosoma una determinata posizione (locus) viene trasmesso alle generazioni successive come un carattere ereditario È costituito da regioni codificanti le proteine (esoni) e regioni non codificanti (introni) fiancheggianti gli esoni
4 Dalla sequenza codificata di un gene alla sua funzione
5 Funzioni dei geni umani Gene ontology (GO) PANTHER (Protein ANalysis THrough Evolutionary Relationships) classification system biological database
6 Genome Sequencing TG..GT TC..CC AC..GC CG..CA TT..TC AC..GC GA..GC TG..AC CT..TG GT..GC AC..GC AC..GC AA..GC AT..AT TT..CC Genome Short fragments of DNA Short DNA sequences ACGTGGTAA CGTATACAC TAGGCCATA GTAATGGCG CACCCTTAG TGGCGTATA CATA ACGTGGTAATGGCGTATACACCCTTAGGCCATA ACGTGACCGGTACTGGTAACGTACA CCTACGTGACCGGTACTGGTAACGT ACGCCTACGTGACCGGTACTGGTAA CGTATACACGTGACCGGTACTGGTA ACGTACACCTACGTGACCGGTACTG GTAACGTACGCCTACGTGACCGGTA CTGGTAACGTATACCTCT... Sequenced genome 6
7 DNA Sequencing Basic DNA sequencing o Sanger method (chain termination) o Maxam Gilbert method (chemical termination cleavage) Advanced DNA sequencing o Shotgun Next Generation Sequencing o Solid Sequencing o Illumina Sequencing o Pyrosequencing
8 DNA Sequencing: the chain termination method (Sanger's method)
9 DNA Sequencing: the chain termination method (Sanger's method) A B C D
10 DNA Sequencing: the chain termination method (Sanger's method) deoxy NTPs have a 3 hydroxyl group so chain elongation can occur dideoxy NTPs don t have a 3 hydroxyl group so when this NTPs is added, chain elongation terminates
11 DNA Sequencing: the chain termination method (Sanger's method)
12 DNA Sequencing: the chain termination method (Sanger's method) ddatp ddttp ddgtp ddctp ddatp ddttp ddgtp ddctp Denaturing PAGE gels for DNA sequencing: gel is 40 cm long with a uniform thickness of 0.4 mm 25-40% 40 % acrylamide/bisacrylamide, mixing ratio 29:1 6-8 M urea as their denaturant TBE as their buffer system After a hour run, a sequencing gel will give bases of readable sequence starting at or close to the end of the primer. The radioactive label exposes a sheet of X-ray film by autoradiography
13 DNA Sequencing: the chain termination method (Sanger's method) dsdna 5 -ATGGCAGTTATCGATCGAT-3 3 -TACCGTCAATAGCTAGCTA-5 ssdna - extention ATCGATCGAT P-ATGGCAGTT- 3 -TACCGTCAATAGCTAGCTA-5 denaturation 5 -ATGGCAGTTATCGATCGAT-3 3 -TACCGTCAATAGCTAGCTA-5 annealing 5-32P-ATGGCAGTT TACCGTCAATAGCTAGCTA-5
14 Reading: to identify sequence A G C G T C G C A G
15 DNA Sequencing: chemical termination cleavage (Maxam and Gilbert) A single stranded DNA fragment is obtained by PCR using 5 -end-labeled primer The oligonucleotide employed as primer is end-labeled by treatment with alkaline phosphatase to remove the 5 -phosphate (if it s present). Then it s followed the reaction with P 32 -labeled ATP in the presence of polynucleotide kinase, this enzyme attaches P 32 -labeled to the 5 -terminal.
16 DNA Sequencing: Sanger method vs. Maxam Gilbert method Sanger Sequencing Maxam Gilbert Sequencing
17 DNA Sequencing: chemical termination cleavage (Maxam and Gilbert) The labeled DNA fragment is then divided into 4 aliquots, each treated with a chemical agent, which modifies a specific base: 1. Aliquot A + dimethyl sulphate, which methylated GUANINE residue 2. Aliquot B + formic acid, which modifies ADENINE and GUANINE residues 3. Aliquot C + hydrazine, which modifies THYMINE and CYTOSINE residues 4. Aliquot D + hydrazine+nacl 5 mol/l, which makes the reaction specific for CYTOSINE residues The 4 aliquots are incubated with piperidine, which cleaves the sugar phosphate backbone of DNA next to the residue that has been modified
18 Dimethyl sulphate to methylate GUANINE residue GUANINE DMS methylates the N7 position of guanine, which then leads to facile depurination piperidine will cleave the phosphate bond at the 3 carbon. dimethyl sulphate, formic acid, hydrazine, hydrazine+ NaCl 5 mol/l attack the purine or pyrimidine rings respectively Piperidine will cleave the phosphate bond at the 3 carbon
19 DNA Sequencing: chemical termination cleavage (Maxam and Gilbert)
20 Reading: to identify sequence Radioactive fragments Unlabelled fragments
21 DNA Sequencing: Sanger method vs. Maxam Gilbert method Sanger method DNA sequence is annealed to an endlabeled oligonucleotide with [ 32 P], which is then extended by DNA polymerase using a mixture of dntp DNA sequence is stopped using ddntp as chain terminator sequence DNA fragment up to 500 nucleotides in length (250 using forward radiolabelled primer and 250 using reverse radiolabelled primer) Maxam Gilbert method DNA sequence is annealed to an endlabeled oligonucleotide with [ 32 P], as primer, which is then extended by DNA polymerase using a mixture of dntp DNA sequence is chemically cleaved to leave a signature pattern of bands sequence DNA fragment up to 500 nucleotides in length (250 using forward radiolabelled primer and 250 using reverse radiolabelled primer)
22 Shotgun for genome sequencing CGATTAGGCGTACGTACGGTCGAATGCAACGTAGGCCTACGAATGCG
23 BAC cloning Shotgun sequencing
24 Shotgun sequencing 1. Amplificazione del DNA mediante clonaggio in vettori BAC (Bacterial Artificial Chromosome) per studiare: geni di grandi dimensioni diversi geni in una sola volta interi genomi virali 2. La sequenza dei frammenti è ottenuta utilizzando il metodo Sanger
25 Shotgun sequencing
26
27 Fluorescent DNA sequencing
28 Fluorescent DNA sequencing
29 Fluorescent DNA sequencing
30 Capillary Electrophoresis in DNA sequencing Sequencing a lot of samples at the same time
31 Sequenziamento automatizzato usando ddntp legati a molecole cromogeniche
32 Sequenziamento automatizzato usando ddntp legati a molecole cromogeniche Vantaggi Lunghe letture di frammenti (~ 900 bps) Adatto per piccoli progetti di sequenziamento Svantaggi Bassa produttività Costoso
33 Shotgun sequencing Avantages Much more economical and faster Labour saving because the sequencing reactions are virtually fully automated and the sequences being assemblated by computer programs Disvantages It requires a great skill to allign the overlapped fragments and to deduce the sequence Complex process
34 Minisequencing single base extension
35 Minisequencing single base extension for SNPs analysis The minisequencing reaction uses a DNA polymerase to extend detection primers that anneal immediately adjacent to the sites of the SNPs. The primers are extended with fluorescently labeled, terminating nucleotide analogues that are complementary to the nucleotide at the SNP site.
36 Minisequencing single base extension
37
38 Obiettivi principali dell Human Genome Project determinare la sequenza delle coppie di basi azotate che formano il DNA identificare e mappare (localizzare) i geni Il genoma di qualsiasi individuo (tranne quello dei gemelli monozigoti e degli organismi clonati) è unico; mappare il genoma umano significa quindi fare il sequenziamento di tutte le variazioni di un gene, per questo si è lavorato sul DNA di donatori selezionati con criteri di rappresentatività statistica comprendere la funzione dei geni determinare la sequenza anche di altri organismi per studi applicati alle Scienze Evoluzionistiche creazione banche dati integrate ad un interfaccia web per la ricerca ed analisi dei dati
39 Principali scoperte del Progetto Genoma Gli esseri umani hanno circa geni, più o meno lo stesso numero di quelli dei topi e da confrontarsi con i circa di una pianta e i di un verme, questa differenza non sembra abbastanza marcata per spiegare la complessità dell'organismo umano rispetto a forme di vita più semplici. Tutte le razze umane sono uguali al 99%. Indicando una discendenza evolutiva da un'unica madre. La maggior parte delle mutazioni genetiche avviene nel maschio della specie, il maggiore responsabile nella trasmissione delle variazioni genetiche. La comprensione dell'espressione di questi geni potrà fornire degli indizi sulle cause di alcune malattie.
40 Log 10 (price) Sequencing the Human Genome 2001: Human Genome Project 2.7G$, 11 years : Celera 100M$, 3 years 2005 Year 2007: 454 1M$, 3 months 2008: ABI SOLiD 60K$, 2 weeks 2009: Illumina, Helicos 40-50K$ : 5K$, a few days? 2012: 100$, <24 hrs?
41
42 Next Generation Sequencing
43 Next Generation Sequencing
44 1. NGS: sample preparation
45 Frammentazione del DNA e in vitro legame degli «adattatori» In generale, gli step di base nella preparazione di RNA o DNA per analisi NGS sono: (i) frammentazione e/o riduzione delle sequenze bersaglio ad una lunghezza desiderata, (ii) conversione del substrato (RNA o DNA) in DNA a doppia elica, (iii) fissare oligonucleotidi adattatori alle estremità dei frammenti di DNA bersaglio (iv) arricchimento mediante PCR dei prodotti per il sequenziamento della «library». Le dimensioni dei frammenti di DNA bersaglio nella biblioteca finale è un parametro fondamentale per NGS costruzione della «library». Il Next Generation Sequencing, vi darà un sacco di letture di sequenze, ma di tratterà di brevi frammenti di lettura. con Illumina e Ion Torrent le letture sono attualmente della lunghezza di 600 basi. con Roche 454 legge meno di 1kb e PacBio meno di 9 kb di lunghezza. (i) (ii) (iii) (iv) Questo rende molto importante la definizione delle dimensione dei frammenti di DNA da produrre prima della costruzione biblioteca.
46 Metodi per la frammentazione del DNA 1. Fisico 2. Chimico 3. Enzimatico
47 1. Frammentazione fisica del DNA a) Rottura acustica taglio acustico e ad ultrasuoni sono i principali metodi fisici utilizzati per rompere il DNA. Lo strumento Covaris (Woburn, MA) è un dispositivo acustico per la rottura del DNA in frammenti di circa kb. Covaris produce anche tubi (gtubes) per processare campioni di 6-20 kb. b) Sonicazione Il Bioruptor (Denville, NJ) è un dispositivo che utilizza ultrasuoni per la frattura della cromatina, del DNA e dei tessuti. Piccole quantità di DNA possono essere tagliate in kb di lunghezza. c) Taglio idrodinamico Hydroshear, una sorta di forbici idrauliche della Digilab (Marlborough, MA) utilizzano forze idrodinamiche per tagliare il DNA. Nebulizzatori (Vita Tech, Grand Island, NY) possono anche essere usati per frammentare il DNA in pezzi da kb in pochi secondi. Mentre la nebulizzazione ha un basso costo e non richiede l'acquisto di uno strumento, non è tuttavia consigliabile se avete poco materiale di partenza: si può perdere fino al 30% del vostro DNA con questo metodo. Gli altri dispositivi sonicazione e il taglio acustico sono progettate per volumi più piccoli e non causano perdite del DNA di partenza. 2. Frammentazione chimica del DNA Calore e cationi di metalli bivalenti Il taglio chimico è generalmente limitato alla rottura di lunghi frammenti di RNA. Viene tipicamente eseguito tramite la digestione di RNA col calore e cationi di metallo bivalenti (magnesio o zinco). La lunghezza del RNA ( bp) può essere regolata aumentando o diminuendo il tempo di incubazione. 3. Frammentazione del DNA con metodi enzimatici a) DNasi I o alter endonucleasi di restrizione, nuclease non-specifiche I metodi enzimatici includono la digestione con DNasi I o Fragmentasi, o una miscela dei due. La Fragmentasi sembra essere la più efficace. Tuttavia, la Fragmentase produce un numero maggiore di artefatti rispetto alla frammentazione con metodi fisici. a) Transposasi Un metodo enzimatico alternativo è rappresentato dalla tecnologia tagmentation NextEra (Illumina, San Diego, CA), in cui un enzima la Trasposasi frammenta e contemporaneamente inserisce sequenze dell adattatore in dsdna. Questo metodo presenta diversi vantaggi, tra cui ridotta manipolazione del campione e brevi tempo di preparazione.
48 Transposase tagmentation reaction La transposasi frammenta e simultaneamente inserisce adattatori dentro un dsdna Sono state sequenziate library contenenti frammenti di lunghezza superior alle 1500 basi
49 Sonication vs. Transposase fragmentation Scott Brouilette et all. DEVELOPMENTAL DYNAMICS 241: , 2012
50 Legame dell adattatore
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecniche di sequenziamento del DNA
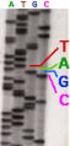 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei
 ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Replicazione del DNA
 Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
Replicazione del DNA la replicazione del DNA viene effettuata da ENZIMI: DNA-polimerasi (catalizza la formazione del legame fosfodiestere) ogni filamento fa da stampo (enzima diretto dallo stampo) le DNA-polimerasi
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI
 LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
Il sequenziamento del DNA
 Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
Laboratorio di Elementi di Bioinformatica
 Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2016/2017 I dati in Bioinformatica Docente del laboratorio: Raffaella Rizzi 1 Il DNA (oggetto biologico)
Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2016/2017 I dati in Bioinformatica Docente del laboratorio: Raffaella Rizzi 1 Il DNA (oggetto biologico)
TRASCRIZIONE DEL DNA. Formazione mrna
 TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
Relatrice: dott.ssa Ilaria Pegoretti
 Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
GENE. Fattore trasformante? Riproduzione del ceppo S. Preparazione del fattore trasformante. Trasformazione del ceppo R. ceppo S. deceduto.
 ceppo S 1 Griffith and Avery 1930 deceduto ceppo R 2 vivo ceppo S ucciso al calore 3 ceppo R 4 vivo ceppo S ucciso al calore deceduto Riproduzione del ceppo S Preparazione del fattore trasformante lisi
ceppo S 1 Griffith and Avery 1930 deceduto ceppo R 2 vivo ceppo S ucciso al calore 3 ceppo R 4 vivo ceppo S ucciso al calore deceduto Riproduzione del ceppo S Preparazione del fattore trasformante lisi
Genotipizzazione virale
 Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp ( Mbp=150Gbp) di una
 Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp (150.000Mbp=150Gbp) di una Liliacea. Tra le graminacee il frumento ha un genoma
Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp (150.000Mbp=150Gbp) di una Liliacea. Tra le graminacee il frumento ha un genoma
Polymerase Chain Reaction
 Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
Polymerase Chain Reaction La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona
 Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale
 ? what is ChIP?? È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale E uno strumento potente per ottenere immagini ad alta risoluzione di proteine nel contesto
? what is ChIP?? È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale E uno strumento potente per ottenere immagini ad alta risoluzione di proteine nel contesto
David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis. Biologia La scienza della vita
 1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione La regolazione genica negli eucarioti 3 I genomi
1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione La regolazione genica negli eucarioti 3 I genomi
Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata Via Belmeloro, 8 Bologna
 GENETICA GENERALE - 1 CFU Modulo Biologia Applicata e Genetica generale CORSO INTEGRATO: SCIENZE BIOLOGICHE - 7 CFU Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata
GENETICA GENERALE - 1 CFU Modulo Biologia Applicata e Genetica generale CORSO INTEGRATO: SCIENZE BIOLOGICHE - 7 CFU Dott.ssa Raffaella Casadei Dipartimento di Istologia Embriologia e Biologia Applicata
Corso di Genetica -Lezione 8- Cenci
 Corso di Genetica -Lezione 8- Cenci Mappatura mediante ricombinazione corpo nero; fenotipo dominante N(ero)/N(ero) Su quale cromosoma? Y; N/N X w/w; Cy/Sco; Sb/Ser Tutti maschi occhio bianco sono normali
Corso di Genetica -Lezione 8- Cenci Mappatura mediante ricombinazione corpo nero; fenotipo dominante N(ero)/N(ero) Su quale cromosoma? Y; N/N X w/w; Cy/Sco; Sb/Ser Tutti maschi occhio bianco sono normali
Introduzione alla Genomica
 Laboratorio di Bioinformatica I Introduzione alla Genomica Dott. Sergio Marin Vargas (2014 / 2015) Il Genoma umano Gene codificanti proteine Gene non codificanti proteine Geni codificanti proteine 3 Il
Laboratorio di Bioinformatica I Introduzione alla Genomica Dott. Sergio Marin Vargas (2014 / 2015) Il Genoma umano Gene codificanti proteine Gene non codificanti proteine Geni codificanti proteine 3 Il
TRE PAROLE CHIAVE DELLA GENETICA
 TRE PAROLE CHIAVE DELLA GENETICA Questo documento è pubblicato sotto licenza Creative Commons Attribuzione Non commerciale Condividi allo stesso modo http://creativecommons.org/licenses/by-nc-sa/2.5/deed.it
TRE PAROLE CHIAVE DELLA GENETICA Questo documento è pubblicato sotto licenza Creative Commons Attribuzione Non commerciale Condividi allo stesso modo http://creativecommons.org/licenses/by-nc-sa/2.5/deed.it
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Università degli Studi di Ferrara. UTILIZZO DI U1-snRNA PER LA CORREZIONE DEL DIFETTO SEVERO DI FATTORE VII DELLA COAGULAZIONE
 Università degli Studi di Ferrara UTILIZZO DI U1-snRNA PER LA CORREZIONE DEL DIFETTO SEVERO DI FATTORE VII DELLA COAGULAZIONE FVII TF FVIIa FX PT FXa FVa TH FVII TF FVIIa FX PT FXa FVa TH Deficienza congenita
Università degli Studi di Ferrara UTILIZZO DI U1-snRNA PER LA CORREZIONE DEL DIFETTO SEVERO DI FATTORE VII DELLA COAGULAZIONE FVII TF FVIIa FX PT FXa FVa TH FVII TF FVIIa FX PT FXa FVa TH Deficienza congenita
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
particolare sequenza di purine e pirimidine elica localmente sinistrorsa andamento a zig-zag (da cui il nome)
 particolare sequenza di purine e pirimidine elica localmente sinistrorsa andamento a zig-zag (da cui il nome) i due solchi, maggiore e minore, dell'elica B sono qui sostituiti da un singolo (profondo)
particolare sequenza di purine e pirimidine elica localmente sinistrorsa andamento a zig-zag (da cui il nome) i due solchi, maggiore e minore, dell'elica B sono qui sostituiti da un singolo (profondo)
Malattie genetiche. Dott. Giovanni LONGO
 Malattie genetiche Dott. Giovanni LONGO Il ruolo della ricerca scientifica Esistono una quantità quasi infinita di rimedi o cure contro un'altrettanta quantità di malattie. La ricerca scientifica si occupa
Malattie genetiche Dott. Giovanni LONGO Il ruolo della ricerca scientifica Esistono una quantità quasi infinita di rimedi o cure contro un'altrettanta quantità di malattie. La ricerca scientifica si occupa
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
IL GENOMA DELLA CELLULA VEGETALE
 IL GENOMA DELLA CELLULA VEGETALE I GENOMI DELLE CELLULE VEGETALI Genoma nucleare Geni per il funzionamento globale della cellula vegetale Condivisi o specifici per la cellula vegetale Genoma plastidiale
IL GENOMA DELLA CELLULA VEGETALE I GENOMI DELLE CELLULE VEGETALI Genoma nucleare Geni per il funzionamento globale della cellula vegetale Condivisi o specifici per la cellula vegetale Genoma plastidiale
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
ACIDI NUCLEICI ESPERIMENTI
 ACIDI NUCLEICI ESPERIMENTI 1869 FRIEDRICK MIESCHER Isolò per la prima volta una sostanza zuccherina leggermente acida contenente fosforo Questa sostanza venne chiamata: acido nucleico perché scoperta nel
ACIDI NUCLEICI ESPERIMENTI 1869 FRIEDRICK MIESCHER Isolò per la prima volta una sostanza zuccherina leggermente acida contenente fosforo Questa sostanza venne chiamata: acido nucleico perché scoperta nel
Il genoma umano. Cosa significa genoma? Ditelo con parole vostre
 Il genoma umano Cosa significa genoma? Ditelo con parole vostre Cos è il genoma umano? 22 coppie di autosomi + una coppia di cromosomi sessuali (XX o XY) http://www.molecularstation.com/molecular-biology-images/data/502/human-genome-gene.png
Il genoma umano Cosa significa genoma? Ditelo con parole vostre Cos è il genoma umano? 22 coppie di autosomi + una coppia di cromosomi sessuali (XX o XY) http://www.molecularstation.com/molecular-biology-images/data/502/human-genome-gene.png
PRINCIPALI APPLICAZIONI DELLA PCR. PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b) PRINCIPALI TIPI DI PCR a)
 EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
Principali classi di mutazioni
 Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La genetica molecolare
 La genetica molecolare 1 Il materiale genetico Varia di quantità da specie a specie. Regola lo sviluppo della cellula. Ha la capacità di duplicarsi. Nome comune Numero di coppie di cromosomi zanzara 3
La genetica molecolare 1 Il materiale genetico Varia di quantità da specie a specie. Regola lo sviluppo della cellula. Ha la capacità di duplicarsi. Nome comune Numero di coppie di cromosomi zanzara 3
Il DNA mobile rappresenta una componente importante del genoma eucariotico e procariotico
 Il DNA mobile rappresenta una componente importante del genoma eucariotico e procariotico In Drosophila meta delle mutazioni sono generate da elementi genetici mobili Generalmente le componenti mobili
Il DNA mobile rappresenta una componente importante del genoma eucariotico e procariotico In Drosophila meta delle mutazioni sono generate da elementi genetici mobili Generalmente le componenti mobili
PROGETTO DNA chiavi in mano. Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi
 PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
PROGETTO DNA chiavi in mano Le Biotecnologie in classe e in laboratorio 11/03/2015 Prof.ssa Paola Cazzani I.T.E. A. Bassi - Lodi PROGETTO DNA chiavi in mano PROGETTO DNA chiavi in mano IFOM PROGETTO DNA
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Sequenziamento del DNA: strutture dei nucleotidi
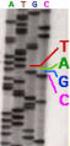 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Genomica, proteomica, genomica strutturale, banche dati.
 Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3. Criteri di identificazione
 Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Test con punteggio 0.5
 Cognome e Nome Associazione Onlus Lesina (FG) Venerdi 12 Ottobre 2007 Test con punteggio 0.5 1. Un gene è 6. La coppia di cromosomi XY potrebbe essere quella di chi? una proteina il piano di costruzione
Cognome e Nome Associazione Onlus Lesina (FG) Venerdi 12 Ottobre 2007 Test con punteggio 0.5 1. Un gene è 6. La coppia di cromosomi XY potrebbe essere quella di chi? una proteina il piano di costruzione
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Importanza della genetica dei microrganismi
 Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
Il benessere passa dalla tua tavola
 Il benessere passa dalla tua tavola Test Genetico per il metabolismo dell omocisteina. Introduzione. Oltre 30.000 geni compongono il DNA di una persona, una sorta di istruzioni per l uso per l organismo.
Il benessere passa dalla tua tavola Test Genetico per il metabolismo dell omocisteina. Introduzione. Oltre 30.000 geni compongono il DNA di una persona, una sorta di istruzioni per l uso per l organismo.
MODELLO SCHEDA INSEGNAMENTO
 Corso di Laurea Denominazione insegnamento: Numero di Crediti: Anno: Semestre: Docente Titolare: MODELLO SCHEDA INSEGNAMENTO Triennale in Scienze Biologiche Biologia molecolare 9 CFU II II Lina Sabatino
Corso di Laurea Denominazione insegnamento: Numero di Crediti: Anno: Semestre: Docente Titolare: MODELLO SCHEDA INSEGNAMENTO Triennale in Scienze Biologiche Biologia molecolare 9 CFU II II Lina Sabatino
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
lezione giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
Quando mi collego ad alcuni servizi hosting ricevo un messaggio relativo al certificato di protezione del sito SSL, come mai?
 IT FAQ-SSL Quando mi collego ad alcuni servizi hosting ricevo un messaggio relativo al certificato di protezione del sito SSL, come mai? Il certificato SSL relativo ai servizi hosting è stato rinnovato
IT FAQ-SSL Quando mi collego ad alcuni servizi hosting ricevo un messaggio relativo al certificato di protezione del sito SSL, come mai? Il certificato SSL relativo ai servizi hosting è stato rinnovato
GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Lezione 5. Next Generation Sequencing
 Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Studio della regolazione dell espressione genica
 Studio della regolazione dell espressione genica Un gene soggetto a regolazione possiede una o più sequenze regolatorie posizionate in maniera diversa rispetto al gene. I regolatori possono essere positivi
Studio della regolazione dell espressione genica Un gene soggetto a regolazione possiede una o più sequenze regolatorie posizionate in maniera diversa rispetto al gene. I regolatori possono essere positivi
I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più.
 Piante transgeniche prive di geni marker I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più. Possibili problemi una volta in
Piante transgeniche prive di geni marker I geni marker sono necessari per l'isolamento di piante transgeniche (efficienza di trasf. non ottimale), ma poi non servono più. Possibili problemi una volta in
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
Tecniche biofisiche basate su nanopori
 Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
IL GIOVANE HOLDEN FRANNY E ZOOEY NOVE RACCONTI ALZATE LARCHITRAVE CARPENTIERI E SEYMOUR INTRODUZIONE BY JD SALINGER
 IL GIOVANE HOLDEN FRANNY E ZOOEY NOVE RACCONTI ALZATE LARCHITRAVE CARPENTIERI E SEYMOUR INTRODUZIONE BY JD SALINGER READ ONLINE AND DOWNLOAD EBOOK : IL GIOVANE HOLDEN FRANNY E ZOOEY NOVE RACCONTI ALZATE
IL GIOVANE HOLDEN FRANNY E ZOOEY NOVE RACCONTI ALZATE LARCHITRAVE CARPENTIERI E SEYMOUR INTRODUZIONE BY JD SALINGER READ ONLINE AND DOWNLOAD EBOOK : IL GIOVANE HOLDEN FRANNY E ZOOEY NOVE RACCONTI ALZATE
CORSO BIOLOGIA MOLECOLARE I. Testi consigliati
 CORSO BIOLOGIA MOLECOLARE I Dott. Massimo Pancione e.mail massimo.pancione@unisannio.it Obiettivo del corso: Comprendere i meccanismi molecolari dei processi biologici fondamentali, descrivere le tecniche
CORSO BIOLOGIA MOLECOLARE I Dott. Massimo Pancione e.mail massimo.pancione@unisannio.it Obiettivo del corso: Comprendere i meccanismi molecolari dei processi biologici fondamentali, descrivere le tecniche
Info per il futuro. Erasmus a Parigi: Master giornalismo: RIS check giornata su
 Info per il futuro Erasmus a Parigi: www.saggiolab.it Master giornalismo: www.mastersgp.it RIS check giornata su www.mastersgp.it Organizzazione corso Lezioni Esercitazioni bonus (17/12, 19/12, 7/1, 9/1,
Info per il futuro Erasmus a Parigi: www.saggiolab.it Master giornalismo: www.mastersgp.it RIS check giornata su www.mastersgp.it Organizzazione corso Lezioni Esercitazioni bonus (17/12, 19/12, 7/1, 9/1,
Definizione di genoteca (o library) di DNA
 Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
IBRIDAZIONE DNA:DNA. Tassonomia molecolare CHEMOTAXONOMY
 IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
Corso di Elementi di Bioinformatica
 Corso di Elementi di Bioinformatica Laurea Triennale in Informatica I dati e le banche dati in Bioinformatica Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 Il DNA (oggetto biologico)
Corso di Elementi di Bioinformatica Laurea Triennale in Informatica I dati e le banche dati in Bioinformatica Anno Accademico 2015-2016 Docente del laboratorio: Raffaella Rizzi 1 Il DNA (oggetto biologico)
Nuovi ruoli dei telomeri e della telomerasi
 Nuovi ruoli dei telomeri e della telomerasi Marco Santagostino Tutor: Elena Giulotto Dipartimento di Genetica e Microbiologia, Università degli Studi di Pavia Argomenti trattati 1. I telomeri e la telomerasi
Nuovi ruoli dei telomeri e della telomerasi Marco Santagostino Tutor: Elena Giulotto Dipartimento di Genetica e Microbiologia, Università degli Studi di Pavia Argomenti trattati 1. I telomeri e la telomerasi
