COME CALCOLARE IL PUNTEGGIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi
|
|
|
- Aloisia Di Giacomo
- 7 anni fa
- Visualizzazioni
Transcript
1 COME CALCOLARE IL PUNTEGGIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al lato pratico. 1) edit-distance o distanza di Levenshtein: in questo caso il punteggio dell allineamento è calcolato basandosi sul minimo numero di sostituzioni, inserzioni e delezioni che si devono fare per passare dalla sequenza A alla sequenza B (concetto dell evoluzione). Ad esempio ai match si attribuisce un valore di 0 ed ai mismatch un valore positivo tipo 1. 2) Punteggio di similarità: il punteggio dell allineamento è frutto del calcolo della similarità tra le due sequenze allineate in questione. Ovvero si deve trovare l allineamento con il punteggio più alto. Ai match si attribuisce un valore positivo. Processo addittivo. Nella maggior parte dei casi le due quantità sono correlate ma ci sono delle differenze concettuali. L edit-distance è adatta nel caso degli allineamenti globali. Per l editdistance deve valere la disuguaglianza triangolare. Se ho x, y e z e l evento di mutazione da x a y ha un costo > dell evento x->z + z->y allora ogni volta che devo sostituire x con y devo effettuare due sostituzioni prima con z e poi con y perché al fine del punteggio finale costano meno. L editdistance, infatti, prevede che si devono minimizzare i costi di eventi di mutazioni per passare da una sequenza all altra. Prevale il concetto che l evoluzione deve risparmiare.
2 Specie A Specie B Specie C A W T V A S A V R T S I A Y T V A A A V R T S I A Y T V A A A V L T S I Edit-distance Da A si passa a B (1 mutazione) si passa a C (una nuova mutazione). Quindi da A a C ci sono state due mutazioni (edit-distance = 2). Ovvero per passare da A a C non si passa direttamente ma si passa attraverso lo stato intermedio di B poiché B è meno mutata rispetto ad A di quanto non lo sia C (A->B->C percorso = 2) e quindi non è possibile pensare un rapporto evolutivo che prevede di partire da A poi si arriva a C e da C si arriva a B (A->C->B percorso = 3) devo minimizzare il percorso!!!! Similarità A e B hanno 11 AA in comune A e C ne hanno 10 quindi il percorso anche in questo caso sarà A->B->C devo massimizzare il percorso (A->B = 11 A->C sarebbe 10. Preferisco passare prima attraverso B
3 EDIT-DISTANCE Problema di base: misurare la differenza o distanza tra due sequenze Trasformare una sequenza in un altra per mezzo di una serie di operazioni di editing su singoli caratteri Operazioni di editing: inserzione, delezione, sostituzione, match (non operazione) Edit distance: è definita come il numero minimo di operazioni di editing (inserzione, delezione e sostituzione) necessarie per trasformare la prima sequenza nella seconda RIMDMDMMI v intner wri t ers Edit distance: indica gli eventi di mutazione che hanno differenziato due sequenze (Processo) Allineamento: indica la relazione che intercorre tra due sequenze (Prodotto)
4 SIMILARITA Date due sequenze A e B, costituite ognuna da una serie di residui, rispettivamente (a 1,a 2,a 3,..., a i ) e (b 1,b 2,b 3,..., b i ), il punteggio (score) di un qualsiasi allineamento tra tutti quelli possibili tra le due sequenze può essere calcolato con la formula sottostante. Lo score totale dell'allineamento è dato dalla somma degli scores relativi alle singole coppie di residui appaiati s (a i,, b i ) per tutti i valori di i compresi tra 1 e la lunghezza (L) dell'allineamento, a cui deve essere sottratta la somma delle penalità dovute ai gap; quindi per ciascuno dei G gap deve essere calcolata una penalità γ per I'apertura del gap più una penalità δ per ogni sua singola estensione. Pur non essendo I'unico modo possibile di assegnare uno score di similarità il criterio riportato sopra è utilizzato dai principali programmi di allineamento. II migliore allineamento tra tutti quelli possibili tra due sequenze è quello che produce il massimo score, per cui generalmente si adotta lo score del migliore allineamento come score di similarità delle due sequenze. sequenza 1 M - N A L S D R T sequenza 2 M G S D R T T E T punteggio = -5
5 ALLINEAMENTO A PARTIRE DA UNA MATRICE Congiungendo con una linea continua le due estremità il alto a sinistra e in basso a destra possono essere rappresentati diversi percorsi, cui corrispondono altrettanti allineamenti possibili. Costruendo delle matrici di similarità capaci di assegnare valori numerici ad ogni possibile accoppiamento amminoacidico e sviluppando algoritmi capaci di identificare i percorsi con gli score più alti è possibile trovare l allineamento ottimale di due sequenze. LAMIASEQUENZAALLINEASEMPREPERCHE GQGPTCGLAMIASIGGTD GQGPTCGLAMIASIGGTDPREPGKN PREPGKN LAMIASEQUENZ-AALLINEASEMPREPERCHE
6 Le matrici di similarità A differenza degli acidi nucleici in cui gli appaiamenti tra basi complementari hanno tutti lo stesso valore di tipo tutto o niente: appaiamento o non appaiamento nel caso delle proteine abbiamo 20 amminoacidi e le singole sostituzioni amminoacidiche non hanno lo stesso peso. E intuitivo capire, per esempio che la sostituzione di una serina (S) con una treonina (T) oppure di un acido glutammico (E) con un acido aspartico (D) sono ben tollerate dalle proteine perché i corrispondenti amminoacidi sono molto simili tra loro. Su questi presupposti sono state costruite delle matrici di similarità costituite da tabelle in cui a ciascun tipo di sostituzione amminoacidica è assegnato un valore che ne indica il grado di similarità Sebbene queste matrici possano essere basate sulle proprietà chimico-fisiche dei singoli amminoacidi, le matrici più usate sono state sviluppate con metodi statistici che indicano la frequenza con cui un ammino-acido si sostituisce ad un altro in famiglie di proteine omologhe. Allineando famiglie di proteine omologhe, infatti, è possibile calcolare la frequenza con cui un certo amminoacido viene sostituito con un altro, per esempio A V calcolato come numero di allineamenti A-V diviso il numero di coppie allineate e si indica come f A V. Analogamente si calcolano la frequenza complessiva di A e di V, come f A e f V. Infine, da questi valori di frequenze si calcolano i valori delle matrici come log (f A V /(f A x f V ) ) Il prodotto delle frequenze indica la probabilità che l allineamento tra V e A avvenga casualmente (l atteso per eventi indipendenti) mentre il logaritmo per avere quantità trattabili (numeri con molti decimali)
7 Probabilità di AA non correlati (modello random) per tutto l allineamento lo score si formalizza come segue: P ( a, b R ) = C ( a, b ) = q q ai bi i Nel caso invece di Probabilità di AA correlati (modello match) ovvero dovuti all osservazione che a e b in realtà sono AA che derivano da uno stesso antenato c sono cioè evolutivamente correlati il punteggio P ( a, b M ) = M ( a, b) = pai, bi i Il rapporto di queste entità o likelihoods è noto come come odds ratio ed è il rapporto di un evento osservato con quello atteso: M ( a, b) C( a, b) = Per rendere trattabile queste quantità e non perdere decimali nelle moltiplicazioni da effettuare si trasformano in quantità additive con il logaritmo (log odds ratio): i p q ai,bi ai q bi s( a, b) = log( p q ai, bi ai q bi ) S = i s( a, b)
8 Le matrici PAM M. Dayhoff (1978) Il problema fondamentale da affrontare per costruire una matrice di similarità è quello di convenire su una famiglia di proteine omologhe sulle quali basare i calcoli della matrice. Le matrici PAM ( Point accepted mutation) sono costruite su sequenze omologhe che presentano solo l 1% di mutazioni accettate, dove per accettate si intende mutazioni che non alterano la funzione della proteina. Due sequenze sono dette a 1 PAM di distanza se per convertirle l una nell altra si è verificata, in media, una mutazione ogni 100 amminoacidi. Da questi dati vengono inferiti tutti gli altri. Sapendo che la probabilità di due eventi indipendenti è uguale al prodotto delle probabilità possiamo ricavarci valori corrispondenti a proteine molto più divergenti moltiplicando i valori tra loro. Per esempio per ricavarci i valori PAM 2, corrispondenti a due proteine con 2 amminoacidi diversi/100 amminoacidi moltiplichiamo tra loro i valori PAM1 x PAM1. Naturalmente man mano che le sequenze divergono aumenta la probabilità che singole mutazioni revertano da cui deriva che i valori non coincidono più; per esempio una matrice PAM 80 non identifica proteine che divergono per l 80% dei loro residui ma solo del 50%. Per la PAM 250 in cui sono stati calcolati 250 passi evolutivi, ad esempio, il risultato è che le sequenze mantengono ancora un 20% di identità.
9 Calcolo di matrici PAM Basato su 1572 mutazioni in 71 gruppi di sequenze simili almeno all 85% per evitare più di una mutazione nella stessa posizione Le mutazioni non alterano significativamente la funzione delle proteine (mutazioni accettate) Le sequenze simili vengono organizzate in alberi filogenetici dai quali vengono desunte le mutazioni
10 Calcolo di matrici PAM La comprensione delle matrici PAM è complicata dal fatto che con il termine PAM si possono intendere due cose diverse: 1) le matrici PAM di probabilità di sostituzione, 2) le matrici PAM di punteggi (scoring matrix) come la PAM 240, che sono utilizzate dai programmi di allineamento. Queste ultime sono calcolate a partire dalle matrici di probabilità, applicando la seguente formula: dove s (a, b) è il punteggio (score) da attribute all'appaiamento tra i due aminoacidi a e b, mentre M (a, b) e C (a, b) sono rispettivamente la probabilità di sostituzione espressa nella matrice PAM di cui al punto 1) e la probabilità di appaiamento casuale dei due amminoacidi.
11 Calcolo di matrici PAM La probabilità M (a, b) di sostituzione dell'aminoacido a in amminoacido b è calcolata a partire dalla matrice di probabilità PAM 1 ed equivale alla probabilità che, a una definita distanza PAM, i due amminoacidi siano correlati filogeneticamente, cioè siano omologhi. La probabilità di omologia definita in M(a,b) è divisa per la probabilità C (a, b) di trovare casualmente I'appaiamento degli stessi amminoacidi, calcolata in base alla frequenza media di ogni amminoacido, assumendo che tutti gli amminoacidi si possano appaiare senza alcuna preferenza. Per esempio, I'appaiamento tra due amminoacidi con frequenze di 0,1 e 0,05 avverrà con una probabilità di 0,005 dovuta al caso. Procedendo sistematicamente per tutti i valori della matrice, sono calcolati i rapporti di probabilità (definiti odds) che in pratica rappresentano quante volte la probabilità di omologia sia maggiore della probabilità casuale.
12 Calcolo di matrici PAM I rapporti di probabilità calcolati sono convertiti nei loro logaritmi (log odds) in modo che nel calcolo globale del punteggio di un allineamento possano essere sommati piuttosto che moltiplicati tra loro, rendendo piu semplici i calcoli. Valori pari a 0 significano che la probabilità di omologia è uguale alla probabilità di appaiamento casuale, mentre valori positivi e negativi indicano rispettivamente una maggiore o minore probabilità di omologia o di appaiamento casuale. Generalmente i log odds sono moltiplicati per una costante e arrotondati a numero intero per costituire le matrici PAM comunemente usate dai programmi di allineamento. Più le sequenze sono distanti e più le PAM che devono essere usate avranno un numero alto.
13
14 Matrice BLOSUM (Henikoff & Henikoff, 1992) Blocks Amino Acid Substitution Matrices = BLOSUM Basata sulle sostituzioni amminoacidiche osservate in ~2000 blocchi conservati di sequenze. Questi blocchi sono stati estratti da una banca dati di 500 famiglie di proteine Sono contati gli scambi amminoacidici osservati in ciascuna colonna
15
16 Calcolo di matrici BLOSUM Il calcolo della matrice è simile a quello delle PAM ma il termine M(a,b) è relativo alla probabilità di sostituzione negli allineamenti delle famiglie proteiche del database BLOCKS. Il database BLOCKS ha allineamenti di sequenze che sono simili tra loro per una data soglia P che varia in genere da 35% a 95% di identità. Una famiglia di BLOCKS 50 ha sequenze allineate con >= 50% identità tra loro.
17 DIFFERENZE TRA PAM E BLOSUM 1) PAM minori servono per allineare sequenze strettamente correlate PAM maggiori servono per allineare sequenze tra loro distanti viceversa per le BLOSUM 2) Troppo poche sequenze utilizzate per ricavare la matrice di Dayhoff e propagazione dell errore dalla PAM1 alla PAM250 3) Per le matrici BLOSUM non si fa alcuna assunzione di omologia visto che derivano da blocchi conservati di sequenza. È una osservazione diretta 4) Le matrici PAM tendono a dare un peso maggiore alle sostituzioni aminoacidiche che derivano dalla mutazione di una singola base (tirosina/fenilalanina) penalizzando quelle più complesse. 5) Le matrici BLOSUM per tali ragione sono forse più adatte per la valutazione degli allineamenti T C A G T TTT Phe (F) TTC " TTA Leu (L) TTG " TCT Ser (S) TCC " TCA " TCG " TAT Tyr (Y) TAC TAA Ter TAG Ter TGT Cys (C) TGC TGA Ter TGG Trp (W) Il codice genetico C CTT Leu (L) CTC " CTA " CTG " CCT Pro (P) CCC " CCA " CCG " CAT His (H) CAC " CAA Gln (Q) CAG " CGT Arg (R) CGC " CGA " CGG " A ATT Ile (I) ATC " ATA " ATG Met (M) ACT Thr (T) ACC " ACA " ACG " AAT Asn (N) AAC " AAA Lys (K) AAG " AGT Ser (S) AGC " AGA Arg (R) AGG " G GTT Val (V) GTC " GTA " GTG " GCT Ala (A) GCC " GCA " GCG " GAT Asp (D) GAC " GAA Glu (E) GAG " GGT Gly (G) GGC " GGA " GGG "
18 DIFFERENZE TRA PAM E BLOSUM PAM è basato su un modello evolutivo BLOSUM è basato su famiglie proteiche. PAM è basato su allineamento globale. BLOSUM è basato su allineamento locale.
A W T V A S A V R T S I A Y T V A A A V R T S I A Y T V A A A V L T S I
 COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
COME CALCOLARE IL PUNTEIO DI UN ALLINEAMENTO? Il problema del calcolo del punteggio di un allineamento può essere considerato in due modi diversi che, però, sono le due facce di una stessa medaglia al
Classificazione. I complessi. Le pietre miliari della tassonomia. Tassonomia del genere Mycobacterium. Pietre miliari nella tassonomia dei micobatteri
 Le pietre miliari della tassonomia Tassonomia del genere Mycobacterium Enrico Tortoli Centro Regionale di Riferimento per i Micobatteri Firenze Adamo è autorizzato da Dio a dare un nome a tutti gli esseri
Le pietre miliari della tassonomia Tassonomia del genere Mycobacterium Enrico Tortoli Centro Regionale di Riferimento per i Micobatteri Firenze Adamo è autorizzato da Dio a dare un nome a tutti gli esseri
Proficiency test soia Roundup Ready (GTS 40-3-2)
 ISTITUTO ZOOPROFILATTICO SPERIMENTALE DELLE REGIONI LAZIO E TOSCANA (D.L.vo 30.06.1993 n. 270) SEDE CENTRALE - 00178 Roma/Capannelle- Via Appia Nuova, 1411 Tel. (06) 79099.1 (centralino) - Fax (06) 79340724
ISTITUTO ZOOPROFILATTICO SPERIMENTALE DELLE REGIONI LAZIO E TOSCANA (D.L.vo 30.06.1993 n. 270) SEDE CENTRALE - 00178 Roma/Capannelle- Via Appia Nuova, 1411 Tel. (06) 79099.1 (centralino) - Fax (06) 79340724
Come si sceglie l algoritmo di allineamento? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione?
 Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
Come si sceglie l algoritmo di allineamento? Domande: le due proteine hanno domini simili? hanno pezzi di struttura simili? appartengono alla stessa famiglia? svolgono la stessa funzione? hanno un antenato
PROBLEMI CONNESSI ALL USO DI PESTICIDI IN AGRICOLTURA
 PROBLEMI CONNESSI ALL USO DI PESTICIDI IN AGRICOLTURA COSTI DI PRODUZIONE SVILUPPO DI POPOLAZIONI DI INSETTI RESISTENTI NON SPECIFICITÀ DEGRADO AMBIENTALE DANNI ALLA SALUTE DELL UOMO PROTEASI Enzimi che
PROBLEMI CONNESSI ALL USO DI PESTICIDI IN AGRICOLTURA COSTI DI PRODUZIONE SVILUPPO DI POPOLAZIONI DI INSETTI RESISTENTI NON SPECIFICITÀ DEGRADO AMBIENTALE DANNI ALLA SALUTE DELL UOMO PROTEASI Enzimi che
A cosa serve la mutagenesi del DNA?
 MUTAGENESI DEL DNA A cosa serve la mutagenesi del DNA? 1. Studio delle sequenze nucleotidiche, delle funzioni dei geni e di particolari aminoacidi nelle proteine. 2. Ingegnerizzazione di proteine in modo
MUTAGENESI DEL DNA A cosa serve la mutagenesi del DNA? 1. Studio delle sequenze nucleotidiche, delle funzioni dei geni e di particolari aminoacidi nelle proteine. 2. Ingegnerizzazione di proteine in modo
La sintesi delle proteine
 La sintesi delle proteine Struttura del trna In che modo l informazione contenuta sotto forma di sequenze nucleotidiche nel DNA e nell RNA si traduce nella sequenza amminoacidica delle proteine? Esperimenti
La sintesi delle proteine Struttura del trna In che modo l informazione contenuta sotto forma di sequenze nucleotidiche nel DNA e nell RNA si traduce nella sequenza amminoacidica delle proteine? Esperimenti
q xi Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p i >0;
 Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
Modelli probabilis-ci Lanciando un dado abbiamo sei parametri p1 p6 p i >0; 6! i=1 p i =1 Sequenza di dna/proteine x con probabilita q x Probabilita dell intera sequenza n " i!1 q xi Massima verosimiglianza
COREA DI HUNTINGTON. Dott.ssa Silvia Battista Scuola di Specializzazione in Psichiatria
 COREA DI HUNTINGTON Dott.ssa Silvia Battista Scuola di Specializzazione in Psichiatria George Huntington (1850-1916) DESCRIZIONE Disordine genetico neurodegenerativo progressivo A trasmissione autosomica
COREA DI HUNTINGTON Dott.ssa Silvia Battista Scuola di Specializzazione in Psichiatria George Huntington (1850-1916) DESCRIZIONE Disordine genetico neurodegenerativo progressivo A trasmissione autosomica
Le L z e io i ne n 6 Co C n o f n ro r n o t n i i fra r a se s q e u q e u n e z n e z : e di d s i t s a t nz n e z, e allineamenti
 Lezione 6 Confronti fra sequenze: distanze, Confronti fra sequenze: distanze, allineamenti Distanze fra sequenze Per N siti ed n differenze: grado di divergenza = n/n AATGAAAGAA 10 siti; 3 differenze ACTGGAGGAA
Lezione 6 Confronti fra sequenze: distanze, Confronti fra sequenze: distanze, allineamenti Distanze fra sequenze Per N siti ed n differenze: grado di divergenza = n/n AATGAAAGAA 10 siti; 3 differenze ACTGGAGGAA
Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica. Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA.
 Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA. In genere si ottengono trattando il DNA con agenti chimici (es.
Mutagenesi: introduzione di alterazioni in una sequenza nucleotidica Mutagenesi random: le mutazioni avvengono a caso su un tratto di DNA. In genere si ottengono trattando il DNA con agenti chimici (es.
Edit distance. v intner RIMDMDMMI wri t ers
 L'allineamento Edit distance Le operazioni permesse sono: I: insert (inserimento, inserzione) D: delete (cancellazione, delezione, rimozione) R: replacement (substition, sostituzione) M: match (corrispondenza,
L'allineamento Edit distance Le operazioni permesse sono: I: insert (inserimento, inserzione) D: delete (cancellazione, delezione, rimozione) R: replacement (substition, sostituzione) M: match (corrispondenza,
- PROGETTO DI LABORATORIO - PCR
 - PROGETTO DI LABORATORIO - PCR 1 DIFFERENZIAMENTO DEI SUBSET CELLULARI Th1, Th2, Th17 e Treg Controllo/eradicazione di tumori e patogeni intracellulari 2 CITOCHINE DELLA FAMIGLIA DI IL-12 3 4 5 PROGETTO
- PROGETTO DI LABORATORIO - PCR 1 DIFFERENZIAMENTO DEI SUBSET CELLULARI Th1, Th2, Th17 e Treg Controllo/eradicazione di tumori e patogeni intracellulari 2 CITOCHINE DELLA FAMIGLIA DI IL-12 3 4 5 PROGETTO
Omologia di sequenze: allineamento e ricerca
 Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
Omologia di sequenze: allineamento e ricerca Genomi (organismi) e geni hanno un evoluzione divergente Sequenze imparentate per evoluzione divergente sono omologhe Le sequenze sono confrontabili tramite
Allineamento e similarità di sequenze
 Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Allineamento e similarità di sequenze Allineamento di Sequenze L allineamento tra due o più sequenza può aiutare a trovare regioni simili per le quali si può supporre svolgano la stessa funzione; La similarità
Prof. Giorgio Sartor. Sintesi proteica. Trasmissione dell informazione
 rof. Giorgio Sartor Sintesi proteica Copyright 2001-2013 by Giorgio Sartor. All rights reserved. B15 -Versione 1.0 nov2013 Trasmissione dell informazione L informazione è contenuta nel DA ed è trasferita
rof. Giorgio Sartor Sintesi proteica Copyright 2001-2013 by Giorgio Sartor. All rights reserved. B15 -Versione 1.0 nov2013 Trasmissione dell informazione L informazione è contenuta nel DA ed è trasferita
UNIVERSITÁ POLITECNICA DELLE MARCHE FACOLTÁ DI MEDICINA E CHIRURGIA
 UNIVERSITÁ POLITECNICA DELLE MARCHE FACOLTÁ DI MEDICINA E CHIRURGIA Dottorato di Ricerca X ciclo- Nuova serie Curriculum Oncologia MUTAZIONI GERMINALI DI GENI CORRELATI ALLA SINDROME DI LYNCH Tesi di Dottorato
UNIVERSITÁ POLITECNICA DELLE MARCHE FACOLTÁ DI MEDICINA E CHIRURGIA Dottorato di Ricerca X ciclo- Nuova serie Curriculum Oncologia MUTAZIONI GERMINALI DI GENI CORRELATI ALLA SINDROME DI LYNCH Tesi di Dottorato
ALLINEAMENTO DI SEQUENZE
 ALLINEAMENTO DI SEQUENZE 1 DATABASE DI SEQUENZE RICERCA TESTUALE Ricerca dei record i cui campi soddisfano determinati criteri (hanno certi valori) Abbiamo già visto nelle lezioni precedenti SIMILARITA
ALLINEAMENTO DI SEQUENZE 1 DATABASE DI SEQUENZE RICERCA TESTUALE Ricerca dei record i cui campi soddisfano determinati criteri (hanno certi valori) Abbiamo già visto nelle lezioni precedenti SIMILARITA
3.3.2. ALIMENTI DI ORIGINE VEGETALE. 3.3.2.1. Verdure
 3.3.2. ALIMENTI DI ORIGINE VEGETALE 3.3.2.1. Verdure Nella Tabella 9 sono riportati i valori degli aminoacidi liberi presenti in alcune fra le più diffuse verdure in commercio. In un passato recente era
3.3.2. ALIMENTI DI ORIGINE VEGETALE 3.3.2.1. Verdure Nella Tabella 9 sono riportati i valori degli aminoacidi liberi presenti in alcune fra le più diffuse verdure in commercio. In un passato recente era
Caratteristiche generali
 AMMINOACIDI Gli amminoacidi sono le unità costruttive (building blocks) delle proteine. Come dice il termine, gli amminoacidi naturali sono costituiti da un gruppo amminico (-NH 2 ) e da un gruppo carbossilico
AMMINOACIDI Gli amminoacidi sono le unità costruttive (building blocks) delle proteine. Come dice il termine, gli amminoacidi naturali sono costituiti da un gruppo amminico (-NH 2 ) e da un gruppo carbossilico
POLITECNICO DI BARI Corso di Laurea Magistrale in Ingegneria Elettronica BIOINFORMATICA DNA COMPUTING Docente Prof. Giuseppe Mastronardi
 POLITECNICO DI BARI Corso di Laurea Magistrale in Ingegneria Elettronica BIOINFORMATICA DNA COMPUTING Docente Prof. Giuseppe Mastronardi Sommario Introduzione Cenni di biologia Modello di Adleman Modello
POLITECNICO DI BARI Corso di Laurea Magistrale in Ingegneria Elettronica BIOINFORMATICA DNA COMPUTING Docente Prof. Giuseppe Mastronardi Sommario Introduzione Cenni di biologia Modello di Adleman Modello
Probabilità congiunta
 Una vera matrice di sostituzione F K M N P Q 4 5 5 4 3 3 5 6 5 4 3 5 4 4 3 6 4 3 4 5 3 F 8 5 5 4 5 5 4 5 3 4 3 3 6 3 5 K 5 3 3 6 4 3 3 3 M 6 N P 6 Q 4 6 V Y 3 3 3 3 V 4 4 6 8 6 6 6 3 5 3 4 4 6 5 5 6 7
Una vera matrice di sostituzione F K M N P Q 4 5 5 4 3 3 5 6 5 4 3 5 4 4 3 6 4 3 4 5 3 F 8 5 5 4 5 5 4 5 3 4 3 3 6 3 5 K 5 3 3 6 4 3 3 3 M 6 N P 6 Q 4 6 V Y 3 3 3 3 V 4 4 6 8 6 6 6 3 5 3 4 4 6 5 5 6 7
MUTAGENESI DEL DNA. A cosa serve la mutagenesi del DNA?
 A cosa serve la mutagenesi del DNA? MUTAGENESI DEL DNA 1. Studio delle sequenze nucleotidiche, delle funzioni dei geni e di particolari aminoacidi nelle proteine. 2. Ingegnerizzazione di proteine in modo
A cosa serve la mutagenesi del DNA? MUTAGENESI DEL DNA 1. Studio delle sequenze nucleotidiche, delle funzioni dei geni e di particolari aminoacidi nelle proteine. 2. Ingegnerizzazione di proteine in modo
ISTITUTO SUPERIORE DI SANITÀ
 ISTITUTO SUPERIORE DI SANITÀ Metodi microbiologici tradizionali e metodi molecolari per l analisi degli integratori alimentari a base di o con probiotici per uso umano Paolo Aureli, Alfonsina Fiore, Concetta
ISTITUTO SUPERIORE DI SANITÀ Metodi microbiologici tradizionali e metodi molecolari per l analisi degli integratori alimentari a base di o con probiotici per uso umano Paolo Aureli, Alfonsina Fiore, Concetta
Lezione 7. Allineamento di sequenze biologiche
 Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
Lezione 1. Le molecole di base che costituiscono la vita
 Lezione 1 Le molecole di base che costituiscono la vita Le molecole dell ereditarietà 5 3 L informazione ereditaria di tutti gli organismi viventi, con l eccezione di alcuni virus, è a carico della molecola
Lezione 1 Le molecole di base che costituiscono la vita Le molecole dell ereditarietà 5 3 L informazione ereditaria di tutti gli organismi viventi, con l eccezione di alcuni virus, è a carico della molecola
REPLICAZIONE DEL DNA
 REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
REPLICAZIONE DEL DNA La replicazione (o anche duplicazione) è il meccanismo molecolare attraverso cui il DNA produce una copia di sé stesso. Ogni volta che una cellula si divide, infatti, l'intero genoma
Mutazioni. Un cambiamento nel materiale genetico che non venga riparato dai meccanismi di riparo costituisce una mutazione
 Mutazioni Un cambiamento nel materiale genetico che non venga riparato dai meccanismi di riparo costituisce una mutazione Le mutazioni possono essere spontanee oppure causate da agenti fisici, chimici
Mutazioni Un cambiamento nel materiale genetico che non venga riparato dai meccanismi di riparo costituisce una mutazione Le mutazioni possono essere spontanee oppure causate da agenti fisici, chimici
MFN0366-A1 (I. Perroteau) -traduzione e indirizzamento delle proteine. Solo per uso didattico, vietata la riproduzione, la diffusione o la vendita
 MFN0366-A1 (I. Perroteau) -traduzione e indirizzamento delle proteine MFN0366-A1 (I. Perroteau) -traduzione delle proteine trna Traduzione: mrna -------> proteine mrna MFN0366-A1 (I. Perroteau) -traduzione
MFN0366-A1 (I. Perroteau) -traduzione e indirizzamento delle proteine MFN0366-A1 (I. Perroteau) -traduzione delle proteine trna Traduzione: mrna -------> proteine mrna MFN0366-A1 (I. Perroteau) -traduzione
Z-score. lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random
 Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Z-score lo Z-score è definito come: Z-score = (opt query - M random)/ deviazione standard random è una misura di quanto il valore di opt si discosta dalla deviazione standard media. indica di quante dev.
Moltiplicazione. Divisione. Multipli e divisori
 Addizione Sottrazione Potenze Moltiplicazione Divisione Multipli e divisori LE QUATTRO OPERAZIONI Una operazione aritmetica è quel procedimento che fa corrispondere ad una coppia ordinata di numeri (termini
Addizione Sottrazione Potenze Moltiplicazione Divisione Multipli e divisori LE QUATTRO OPERAZIONI Una operazione aritmetica è quel procedimento che fa corrispondere ad una coppia ordinata di numeri (termini
2011 - G. Licini, Università di Padova. La riproduzione a fini commerciali è vietata
 Ammino acidi Composto che contiene una funziome acida e amminica. Usualmente però con amminoacidi si intendono gli alfa- amminoacidi. Tra questi composti ve ne sono 20 che vengono definiti geneticamente
Ammino acidi Composto che contiene una funziome acida e amminica. Usualmente però con amminoacidi si intendono gli alfa- amminoacidi. Tra questi composti ve ne sono 20 che vengono definiti geneticamente
FASTA: Lipman & Pearson (1985) BLAST: Altshul (1990) Algoritmi EURISTICI di allineamento
 Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Algoritmi EURISTICI di allineamento Sono nati insieme alle banche dati, con lo scopo di permettere una ricerca per similarità rapida anche se meno accurata contro le migliaia di sequenze depositate. Attualmente
Il flusso dell informazione genica Le proteine natura ed informazione Il codice genetico La traduzione (sintesi proteica) Cenni sul folding delle
 Il flusso dell informazione genica Le proteine natura ed informazione Il codice genetico La traduzione (sintesi proteica) Cenni sul folding delle proteine Genotipo e fenotipo Mutazioni e polimorfismi Il
Il flusso dell informazione genica Le proteine natura ed informazione Il codice genetico La traduzione (sintesi proteica) Cenni sul folding delle proteine Genotipo e fenotipo Mutazioni e polimorfismi Il
MACROMOLECOLE. Polimeri (lipidi a parte)
 MACROMOLECOLE Monomeri Polimeri (lipidi a parte) Le caratteristiche strutturali e funzionali di una cellula o di un organismo sono determinate principalmente dalle sue proteine. Ad esempio: Le proteine
MACROMOLECOLE Monomeri Polimeri (lipidi a parte) Le caratteristiche strutturali e funzionali di una cellula o di un organismo sono determinate principalmente dalle sue proteine. Ad esempio: Le proteine
Lezione 2: Allineamento di sequenze. BLAST e CLUSTALW
 Lezione 2: Allineamento di sequenze BLAST e CLUSTALW Allineamento di sequenze Allineamenti L avvento della genomica moderna permette di analizzare le similitudini e le differenze tra organismi a livello
Lezione 2: Allineamento di sequenze BLAST e CLUSTALW Allineamento di sequenze Allineamenti L avvento della genomica moderna permette di analizzare le similitudini e le differenze tra organismi a livello
Produzione di proteine eterologhe. Cosa occorre per l espressione l livello di una proteina nella cellula? Trascrizione/traduzione/stabilità
 Cosa occorre per l espressione l ad alto livello di una proteina nella cellula? Trascrizione/traduzione/stabilità Promotore forte (trascrizione) Presenza di segnali per il riconoscimento dell mrna da parte
Cosa occorre per l espressione l ad alto livello di una proteina nella cellula? Trascrizione/traduzione/stabilità Promotore forte (trascrizione) Presenza di segnali per il riconoscimento dell mrna da parte
α-amminoacidi O α O α R CH C O - NH 3 forma ionizzata sale interno (zwitterione) OH NH 2 forma non ionizzata (non esistente in realtà)
 Amminoacidi 2 forma non ionizzata (non esistente in realtà) 3 forma ionizzata sale interno (zwitterione) In soluzione acquosa c'è equilibrio tra tre forme 3 forma cationica p molto acidi 3 forma zwitterionica
Amminoacidi 2 forma non ionizzata (non esistente in realtà) 3 forma ionizzata sale interno (zwitterione) In soluzione acquosa c'è equilibrio tra tre forme 3 forma cationica p molto acidi 3 forma zwitterionica
UNIVERSITA' DEGLI STUDI DI PADOVA
 UNIVERSITA' DEGLI STUDI DI PADOVA Dipartimento di Scienze Farmaceutiche SCUOLA DI DOTTORATO DI RICERCA IN BIOLOGIA E MEDICINA DELLA RIGENERAZIONE INDIRIZZO IN INGEGNERIA DEI TESSUTI E DEI TRAPIANTI XX
UNIVERSITA' DEGLI STUDI DI PADOVA Dipartimento di Scienze Farmaceutiche SCUOLA DI DOTTORATO DI RICERCA IN BIOLOGIA E MEDICINA DELLA RIGENERAZIONE INDIRIZZO IN INGEGNERIA DEI TESSUTI E DEI TRAPIANTI XX
Sintesi e degradazione delle proteine
 Prof. Giorgio Sartor Sintesi e degradazione delle proteine Copyright 2001- by Giorgio Sartor. All rights reserved. B15 - Versione 1.4.1 may Trasmissione dell informazione L informazione è contenuta nel
Prof. Giorgio Sartor Sintesi e degradazione delle proteine Copyright 2001- by Giorgio Sartor. All rights reserved. B15 - Versione 1.4.1 may Trasmissione dell informazione L informazione è contenuta nel
Martina Torricelli, Elisa Pierboni, Gloria Raquel Tovo, Cristina Rondini
 1 Biblioteca Istituto Zooprofilattico Sperimentale dell'umbria e delle Marche Webzine Sanità Pubblica Veterinaria: Numero 87, Dicembre 2014 [http://spvet.it/] ISSN 1592-1581 Confronto quali-quantitativo
1 Biblioteca Istituto Zooprofilattico Sperimentale dell'umbria e delle Marche Webzine Sanità Pubblica Veterinaria: Numero 87, Dicembre 2014 [http://spvet.it/] ISSN 1592-1581 Confronto quali-quantitativo
Distanza di Edit. Speaker: Antinisca Di Marco Data:
 Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
Distanza di Edit Speaker: Antinisca Di Marco Data: 14-04-2016 Confronto di sequenze Il confronto tra sequenze in biologia computazionale è la base per: misurare la similarità tra le sequenze allineamento
C1. Il codone di inizio parte dal quinto nucleotide. La sequenza aminoacidica sarà Met Gly Asn Lys Pro Gly Gln STOP.
 Soluzioni ai problemi del Capitolo 13 Domande concettuali C1. Il codone di inizio parte dal quinto nucleotide. La sequenza aminoacidica sarà Met Gly Asn Lys Pro Gly Gln STOP. C2. Quando si dice che il
Soluzioni ai problemi del Capitolo 13 Domande concettuali C1. Il codone di inizio parte dal quinto nucleotide. La sequenza aminoacidica sarà Met Gly Asn Lys Pro Gly Gln STOP. C2. Quando si dice che il
3.3. UMAMI NEGLI ALIMENTI
 3.3. UMAMI NEGLI ALIMENTI In questo capitolo saranno esaminati i vari cibi, ad esclusione di quelli ittici che verranno presi in considerazione successivamente, allo scopo di scoprire se in essi è presente,
3.3. UMAMI NEGLI ALIMENTI In questo capitolo saranno esaminati i vari cibi, ad esclusione di quelli ittici che verranno presi in considerazione successivamente, allo scopo di scoprire se in essi è presente,
PROTIDI: LE PROTEINE E GLI AMMINOACIDI
 PROTIDI: LE PROTEINE E GLI AMMINOACIDI GLI AMMINOACIDI Struttura generica di un amminoacido. R rappresenta un gruppo laterale specifico di ogni amminoacido. In chimica, gli amminoacidi (impropriamente
PROTIDI: LE PROTEINE E GLI AMMINOACIDI GLI AMMINOACIDI Struttura generica di un amminoacido. R rappresenta un gruppo laterale specifico di ogni amminoacido. In chimica, gli amminoacidi (impropriamente
Struttura delle Proteine
 Chimica Biologica A.A. 2010-2011 Struttura delle Proteine Marco Nardini Dipartimento di Scienze Biomolecolari e Biotecnologie Università di Milano Macromolecole Biologiche Struttura Proteine Proteine:
Chimica Biologica A.A. 2010-2011 Struttura delle Proteine Marco Nardini Dipartimento di Scienze Biomolecolari e Biotecnologie Università di Milano Macromolecole Biologiche Struttura Proteine Proteine:
La rappresentazione dei numeri. La rappresentazione dei numeri. Aritmetica dei calcolatori. La rappresentazione dei numeri
 CEFRIEL Consorzio per la Formazione e la Ricerca in Ingegneria dell Informazione Aritmetica dei calcolatori Rappresentazione dei numeri naturali e relativi Addizione a propagazione di riporto Addizione
CEFRIEL Consorzio per la Formazione e la Ricerca in Ingegneria dell Informazione Aritmetica dei calcolatori Rappresentazione dei numeri naturali e relativi Addizione a propagazione di riporto Addizione
Biologia Molecolare Computazionale
 Biologia Molecolare Computazionale Paolo Provero - paolo.provero@unito.it 2008-2009 Argomenti Allineamento di sequenze Ricostruzione di alberi filogenetici Gene prediction Allineamento Allineamento di
Biologia Molecolare Computazionale Paolo Provero - paolo.provero@unito.it 2008-2009 Argomenti Allineamento di sequenze Ricostruzione di alberi filogenetici Gene prediction Allineamento Allineamento di
Lezione 7. Allineamento di sequenze biologiche
 Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
Lezione 7 Allineamento di sequenze biologiche Allineamento di sequenze Determinare la similarità e dedurre l omologia Allineare Definire il numero di passi necessari per trasformare una sequenza nell altra
LE PROTEINE. SONO Polimeri formati dall unione di AMMINOACIDI (AA) Rende diversi i 20 AA l uno dall altro UN ATOMO DI C AL CENTRO
 LE PROTEINE SONO Polimeri formati dall unione di ATOMI DI C, H, N, O CHE SONO AMMINOACIDI (AA) Uniti tra loro dal Legame peptidico 20 TIPI DIVERSI MA HANNO STESSA STRUTTURA GENERALE CON Catene peptidiche
LE PROTEINE SONO Polimeri formati dall unione di ATOMI DI C, H, N, O CHE SONO AMMINOACIDI (AA) Uniti tra loro dal Legame peptidico 20 TIPI DIVERSI MA HANNO STESSA STRUTTURA GENERALE CON Catene peptidiche
PROGETTO 962 SAFEMILK FINANZIATO DALLA REGIONE LOMBARDIA, Piano 2006
 RISULTATI CONSEGUITI DAL PROGETTO Diagnosi precoce di mastiti subcliniche per un miglioramento quali-quantitativo delle produzioni lattiero-casearie (SAFEMILK) ENTE PROPONENTE Università degli Studi di
RISULTATI CONSEGUITI DAL PROGETTO Diagnosi precoce di mastiti subcliniche per un miglioramento quali-quantitativo delle produzioni lattiero-casearie (SAFEMILK) ENTE PROPONENTE Università degli Studi di
1.4 PRODOTTI NOTEVOLI
 Matematica C Algebra. Le basi del calcolo letterale.4 Prodotti notevoli.4 PRODOTTI NOTEVOLI Il prodotto fra due polinomi si calcola moltiplicando ciascun termine del primo polinomio per ciascun termine
Matematica C Algebra. Le basi del calcolo letterale.4 Prodotti notevoli.4 PRODOTTI NOTEVOLI Il prodotto fra due polinomi si calcola moltiplicando ciascun termine del primo polinomio per ciascun termine
Codoni di STOP: UAA UAG UGA
 PARTECIPANO ALLA TRADUZIONE: trna e aminoacidi Aminoacil-tRNA sintetasi Ribosomi mrna, che contiene una Open Reading Frame (ORF) CODONE DI INIZIO CODONE DI STOP 5 Cap NNNNNN AUG AAA GCA AUU----(n codoni)----uga
PARTECIPANO ALLA TRADUZIONE: trna e aminoacidi Aminoacil-tRNA sintetasi Ribosomi mrna, che contiene una Open Reading Frame (ORF) CODONE DI INIZIO CODONE DI STOP 5 Cap NNNNNN AUG AAA GCA AUU----(n codoni)----uga
Sistemi e problemi, Pag. 1\10 Prof. I. Savoia - Giugno 2011 SISTEMI E PROBLEMI
 Sistemi e problemi, Pag. 1\10 Prof. I. Savoia - Giugno 2011 SISTEMI E PROBLEMI Affrontare un problema richiede spesso l'uso di alcuni strumenti algebrici: fra essi vi sono i sistemi di equazioni. Infatti,
Sistemi e problemi, Pag. 1\10 Prof. I. Savoia - Giugno 2011 SISTEMI E PROBLEMI Affrontare un problema richiede spesso l'uso di alcuni strumenti algebrici: fra essi vi sono i sistemi di equazioni. Infatti,
Indicatori di Posizione e di Variabilità. Corso di Laurea Specialistica in SCIENZE DELLE PROFESSIONI SANITARIE DELLA RIABILITAZIONE Statistica Medica
 Indicatori di Posizione e di Variabilità Corso di Laurea Specialistica in SCIENZE DELLE PROFESSIONI SANITARIE DELLA RIABILITAZIONE Statistica Medica Indici Sintetici Consentono il passaggio da una pluralità
Indicatori di Posizione e di Variabilità Corso di Laurea Specialistica in SCIENZE DELLE PROFESSIONI SANITARIE DELLA RIABILITAZIONE Statistica Medica Indici Sintetici Consentono il passaggio da una pluralità
CARATTERISTICHE DELLE TAG PIU USATE
 Proteine di fusione Proteine di fusione tag proteina Aumento stabilità Purificazione per affinità Metodo di identificazione spettrofotometria Saggio di legame anticorpo Esporto in compartimenti CARATTERISTICHE
Proteine di fusione Proteine di fusione tag proteina Aumento stabilità Purificazione per affinità Metodo di identificazione spettrofotometria Saggio di legame anticorpo Esporto in compartimenti CARATTERISTICHE
i dati escludono vi sia una relazione tra variabile indipendente e variabile dipendente (rispettivamente
 TEST DI AUTOVALUTAZIONE - SETTIMANA 6 I diritti d autore sono riservati. Ogni sfruttamento commerciale non autorizzato sarà perseguito. Metodi statistici per la biologia Parte A. La retta di regressione.2
TEST DI AUTOVALUTAZIONE - SETTIMANA 6 I diritti d autore sono riservati. Ogni sfruttamento commerciale non autorizzato sarà perseguito. Metodi statistici per la biologia Parte A. La retta di regressione.2
Conversione di base. Conversione decimale binario. Si calcolano i resti delle divisioni per due
 Conversione di base Dato N>0 intero convertirlo in base b dividiamo N per b, otteniamo un quoto Q 0 ed un resto R 0 dividiamo Q 0 per b, otteniamo un quoto Q 1 ed un resto R 1 ripetiamo finché Q n < b
Conversione di base Dato N>0 intero convertirlo in base b dividiamo N per b, otteniamo un quoto Q 0 ed un resto R 0 dividiamo Q 0 per b, otteniamo un quoto Q 1 ed un resto R 1 ripetiamo finché Q n < b
Equazioni di 2 grado
 Equazioni di grado Tipi di equazioni: Un equazione (ad una incognita) è di grado se può essere scritta nella forma generale (o forma tipica o ancora forma canonica): a b c con a, b e c numeri reali (però
Equazioni di grado Tipi di equazioni: Un equazione (ad una incognita) è di grado se può essere scritta nella forma generale (o forma tipica o ancora forma canonica): a b c con a, b e c numeri reali (però
Lezione 8. Selezione positiva o darwiniana
 Lezione 8 Selezione positiva o darwiniana Graur and Li: Capitolo 4 (+Cap 2 p63-65) Graur: lecture 20 Ziheng Yang: computational molecular evolution In generale il tasso di sostituzione in geni e regioni
Lezione 8 Selezione positiva o darwiniana Graur and Li: Capitolo 4 (+Cap 2 p63-65) Graur: lecture 20 Ziheng Yang: computational molecular evolution In generale il tasso di sostituzione in geni e regioni
Fondamenti di Informatica - 1. Prof. B.Buttarazzi A.A. 2011/2012
 Fondamenti di Informatica - 1 Prof. B.Buttarazzi A.A. 2011/2012 Sommario I sistemi di numerazione Il sistema binario Altri sistemi di numerazione Algoritmi di conversione Esercizi 07/03/2012 2 Sistemi
Fondamenti di Informatica - 1 Prof. B.Buttarazzi A.A. 2011/2012 Sommario I sistemi di numerazione Il sistema binario Altri sistemi di numerazione Algoritmi di conversione Esercizi 07/03/2012 2 Sistemi
Metodi di Distanza. G.Allegrucci riproduzione vietata
 Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
Metodi di Distanza La misura più semplice della distanza tra due sequenze nucleotidiche è contare il numero di siti nucleotidici che differiscono tra le due sequenze Quando confrontiamo siti omologhi in
ISTITUTO SUPERIORE DI SANITÀ
 ISTITUTO SUPERIORE DI SANITÀ Metodi microbiologici tradizionali e metodi molecolari per l analisi degli integratori alimentari a base di o con probiotici per uso umano Paolo Aureli, Alfonsina Fiore, Concetta
ISTITUTO SUPERIORE DI SANITÀ Metodi microbiologici tradizionali e metodi molecolari per l analisi degli integratori alimentari a base di o con probiotici per uso umano Paolo Aureli, Alfonsina Fiore, Concetta
La codifica. dell informazione
 00010010101001110101010100010110101000011100010111 00010010101001110101010100010110101000011100010111 La codifica 00010010101001110101010100010110101000011100010111 dell informazione 00010010101001110101010100010110101000011100010111
00010010101001110101010100010110101000011100010111 00010010101001110101010100010110101000011100010111 La codifica 00010010101001110101010100010110101000011100010111 dell informazione 00010010101001110101010100010110101000011100010111
ESPONENZIALI E LOGARITMI. chiameremo logaritmica (e si legge il logaritmo in base a di c è uguale a b ).
 ESPONENZIALI E LOGARITMI Data una espressione del tipo a b = c, che chiameremo notazione esponenziale (e dove a>0), stabiliamo di scriverla anche in un modo diverso: log a c = b che chiameremo logaritmica
ESPONENZIALI E LOGARITMI Data una espressione del tipo a b = c, che chiameremo notazione esponenziale (e dove a>0), stabiliamo di scriverla anche in un modo diverso: log a c = b che chiameremo logaritmica
Anno 4 Matrice inversa
 Anno 4 Matrice inversa 1 Introduzione In questa lezione parleremo della matrice inversa di una matrice quadrata: definizione metodo per individuarla Al termine della lezione sarai in grado di: descrivere
Anno 4 Matrice inversa 1 Introduzione In questa lezione parleremo della matrice inversa di una matrice quadrata: definizione metodo per individuarla Al termine della lezione sarai in grado di: descrivere
Bioinformatica e Biologia Computazionale per la Medicina Molecolare
 Facoltà di Ingegneria dell Informazione Laurea Specialistica e Magistrale in Ingegneria Informatica Facoltà di Ingegneria dei Sistemi Laurea Magistrale in Ingegneria Biomedica Dipartimento di Elettronica
Facoltà di Ingegneria dell Informazione Laurea Specialistica e Magistrale in Ingegneria Informatica Facoltà di Ingegneria dei Sistemi Laurea Magistrale in Ingegneria Biomedica Dipartimento di Elettronica
amminico è legato all atomo di carbonio immediatamente adiacente al gruppo carbonilico e hanno la seguente
 Gli amminoacidi naturali sono α-amminoacidi : il gruppo amminico è legato all atomo di carbonio immediatamente adiacente al gruppo carbonilico e hanno la seguente formula generale: gruppo funzionale carbossilico
Gli amminoacidi naturali sono α-amminoacidi : il gruppo amminico è legato all atomo di carbonio immediatamente adiacente al gruppo carbonilico e hanno la seguente formula generale: gruppo funzionale carbossilico
4.2. PRESENZA DI UMAMI NEGLI ALIMENTI ITTICI E DERIVATI
 4.2. PRESENZA DI UMAMI NEGLI ALIMENTI ITTICI E DERIVATI 4.2.1. PESCI, MOLLUSCHI E CROSTACEI Per la compilazione di questa parte, sono affiorati numerosi problemi di natura bibliografica che dimostrano
4.2. PRESENZA DI UMAMI NEGLI ALIMENTI ITTICI E DERIVATI 4.2.1. PESCI, MOLLUSCHI E CROSTACEI Per la compilazione di questa parte, sono affiorati numerosi problemi di natura bibliografica che dimostrano
Bioinformatica: Allineamento di Sequenze di Aminoacidi di Bandiera Roberto
 Bioinformatica: Allineamento di Sequenze di Aminoacidi di Bandiera Roberto La Bioinformatica è una disciplina che si occupa dell applicazione dell informatica nell ambito biologico per consentire lo studio
Bioinformatica: Allineamento di Sequenze di Aminoacidi di Bandiera Roberto La Bioinformatica è una disciplina che si occupa dell applicazione dell informatica nell ambito biologico per consentire lo studio
Visualizzazioni 3D. Informatica. Matrice di voxel. Tipi di dato. Dati vettoriali. Tecniche di rappresentazione
 Informatica Lezione VIII Visualizzazione 3D di proteine Visualizzazioni 3D Rappresentazione di strutture/oggetti tridimensionali Risultato di un esperimento modello teorico dati fisici astrazione 1 Lezione
Informatica Lezione VIII Visualizzazione 3D di proteine Visualizzazioni 3D Rappresentazione di strutture/oggetti tridimensionali Risultato di un esperimento modello teorico dati fisici astrazione 1 Lezione
Misure di diversità tra unità statistiche. Loredana Cerbara
 Misure di diversità tra unità statistiche Loredana Cerbara LA DISTANZA IN STATISTICA In statistica la distanza ha un significato diverso da quello che si può intuire in altre discipline, dove, peraltro,
Misure di diversità tra unità statistiche Loredana Cerbara LA DISTANZA IN STATISTICA In statistica la distanza ha un significato diverso da quello che si può intuire in altre discipline, dove, peraltro,
Ricerca di omologia di sequenza
 Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Ricerca di omologia di sequenza RICERCA DI OMOLOGIA DI SEQUENZA := Data una sequenza (query), una banca dati, un sistema per il confronto e una soglia statistica trovare le sequenze della banca più somiglianti
Corso di Bioinformatica
 Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
Corso di Bioinformatica Cortona - Novembre 2002 Metodi Computazionali per l'analisi delle sequenze Dr. Sabino Liuni Istituto di Tecnologie Biomediche- CNR Sezione di Bioinformatica e Genomica - Bari Sabino@area.ba
Stabilire se il punto di coordinate (1,1) appartiene alla circonferenza centrata nell origine e di raggio 1.
 Definizione di circonferenza e cerchio. Equazione della circonferenza centrata in O e di raggio R. Esercizi. La circonferenza e il cerchio Definizioni: dato un punto C nel piano cartesiano e dato un numero
Definizione di circonferenza e cerchio. Equazione della circonferenza centrata in O e di raggio R. Esercizi. La circonferenza e il cerchio Definizioni: dato un punto C nel piano cartesiano e dato un numero
3. Le coordinate geografiche: latitudine e longitudine
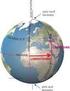 Introduzione 3. Le coordinate geografiche: latitudine e longitudine Ogni volta che vogliamo individuare un punto sulla superficie terrestre gli associamo due numeri, le coordinate geografiche: la latitudine
Introduzione 3. Le coordinate geografiche: latitudine e longitudine Ogni volta che vogliamo individuare un punto sulla superficie terrestre gli associamo due numeri, le coordinate geografiche: la latitudine
Possibile applicazione
 p. 1/4 Assegnamento Siano dati due insiemi A e B entrambi di cardinalità n. Ad ogni coppia (a i,b j ) A B è associato un valore d ij 0 che misura la "incompatibilità" tra a i e b j, anche interpretabile
p. 1/4 Assegnamento Siano dati due insiemi A e B entrambi di cardinalità n. Ad ogni coppia (a i,b j ) A B è associato un valore d ij 0 che misura la "incompatibilità" tra a i e b j, anche interpretabile
Calcolo numerico e programmazione Rappresentazione dei numeri
 Calcolo numerico e programmazione Rappresentazione dei numeri Tullio Facchinetti 16 marzo 2012 10:54 http://robot.unipv.it/toolleeo Rappresentazione dei numeri nei calcolatori
Calcolo numerico e programmazione Rappresentazione dei numeri Tullio Facchinetti 16 marzo 2012 10:54 http://robot.unipv.it/toolleeo Rappresentazione dei numeri nei calcolatori
Perché considerare la struttura 3D di una proteina
 Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
Modelling Perché considerare la struttura 3D di una proteina Implicazioni in vari campi : biologia, evoluzione, biotecnologie, medicina, chimica farmaceutica... Metodi di studio della struttura di una
Chimica Biologica A.A α-elica foglietto β reverse turn
 Chimica Biologica A.A. 2010-2011 α-elica foglietto β reverse turn Str. Secondaria sperimentalmente osservata: Si distinguono fondamentalmente tre tipi di strutture secondarie: α elica foglietto β reverse
Chimica Biologica A.A. 2010-2011 α-elica foglietto β reverse turn Str. Secondaria sperimentalmente osservata: Si distinguono fondamentalmente tre tipi di strutture secondarie: α elica foglietto β reverse
Sistemi di numerazione
 Sistemi di numerazione Introduzione Un sistema di numerazione è un sistema utilizzato per esprimere i numeri e possibilmente alcune operazioni che si possono effettuare su di essi. Storicamente i sistemi
Sistemi di numerazione Introduzione Un sistema di numerazione è un sistema utilizzato per esprimere i numeri e possibilmente alcune operazioni che si possono effettuare su di essi. Storicamente i sistemi
Sistemi di numerazione
 Sistemi di numerazione Sistema di numerazione decimale Sapete già che il problema fondamentale della numerazione consiste nel rappresentare con un limitato numero di segni particolari, detti cifre, tutti
Sistemi di numerazione Sistema di numerazione decimale Sapete già che il problema fondamentale della numerazione consiste nel rappresentare con un limitato numero di segni particolari, detti cifre, tutti
Geometria BIAR Esercizi 2
 Geometria BIAR 0- Esercizi Esercizio. a Si consideri il generico vettore v b R c (a) Si trovi un vettore riga x (x, y, z) tale che x v a (b) Si trovi un vettore riga x (x, y, z) tale che x v kb (c) Si
Geometria BIAR 0- Esercizi Esercizio. a Si consideri il generico vettore v b R c (a) Si trovi un vettore riga x (x, y, z) tale che x v a (b) Si trovi un vettore riga x (x, y, z) tale che x v kb (c) Si
Lezione 4. Problemi trattabili e soluzioni sempre più efficienti. Gianluca Rossi
 Lezione 4 Problemi trattabili e soluzioni sempre più efficienti Gianluca Rossi Trattabile o intrattabile? Consideriamo ora il problema, ben noto a tutti gli studenti a partire dalla scuola media, di calcolare
Lezione 4 Problemi trattabili e soluzioni sempre più efficienti Gianluca Rossi Trattabile o intrattabile? Consideriamo ora il problema, ben noto a tutti gli studenti a partire dalla scuola media, di calcolare
PORTE LOGICHE. Si effettua su due o più variabili, l uscita assume lo stato logico 1 se almeno una variabile di ingresso è allo stato logico 1.
 PORTE LOGICHE Premessa Le principali parti elettroniche dei computer sono costituite da circuiti digitali che, come è noto, elaborano segnali logici basati sullo 0 e sull 1. I mattoni fondamentali dei
PORTE LOGICHE Premessa Le principali parti elettroniche dei computer sono costituite da circuiti digitali che, come è noto, elaborano segnali logici basati sullo 0 e sull 1. I mattoni fondamentali dei
La ricerca di similarità: i metodi
 La ricerca di similarità: i metodi Pairwise alignment allineamenti a coppie 1. Analisi della matrice a punti (dot matrix) 2. Programmazione dinamica (dynamic programming) allineamenti locale e globale.
La ricerca di similarità: i metodi Pairwise alignment allineamenti a coppie 1. Analisi della matrice a punti (dot matrix) 2. Programmazione dinamica (dynamic programming) allineamenti locale e globale.
MISURE DI SINTESI 54
 MISURE DI SINTESI 54 MISURE DESCRITTIVE DI SINTESI 1. MISURE DI TENDENZA CENTRALE 2. MISURE DI VARIABILITÀ 30 0 µ Le due distribuzioni hanno uguale tendenza centrale, ma diversa variabilità. 30 0 Le due
MISURE DI SINTESI 54 MISURE DESCRITTIVE DI SINTESI 1. MISURE DI TENDENZA CENTRALE 2. MISURE DI VARIABILITÀ 30 0 µ Le due distribuzioni hanno uguale tendenza centrale, ma diversa variabilità. 30 0 Le due
PROBLEMI ALGORITMI E PROGRAMMAZIONE
 PROBLEMI ALGORITMI E PROGRAMMAZIONE SCIENZE E TECNOLOGIE APPLICATE CLASSE SECONDA D PROGRAMMARE = SPECIFICARE UN PROCEDIMENTO CAPACE DI FAR SVOLGERE AD UNA MACCHINA UNA SERIE ORDINATA DI OPERAZIONI AL
PROBLEMI ALGORITMI E PROGRAMMAZIONE SCIENZE E TECNOLOGIE APPLICATE CLASSE SECONDA D PROGRAMMARE = SPECIFICARE UN PROCEDIMENTO CAPACE DI FAR SVOLGERE AD UNA MACCHINA UNA SERIE ORDINATA DI OPERAZIONI AL
3 Omotetie del piano. 4 Omotetie del piano. Fondamenti e didattica della matematica B. Geometria delle similitudini. k = 3.
 1 2 Fondamenti e didattica della matematica B 5 marzo 2007 Geometria delle similitudini Marina Bertolini (marina.bertolini@mat.unimi.it) Dipartimento di Matematica F.Enriques Università degli Studi di
1 2 Fondamenti e didattica della matematica B 5 marzo 2007 Geometria delle similitudini Marina Bertolini (marina.bertolini@mat.unimi.it) Dipartimento di Matematica F.Enriques Università degli Studi di
Soluzioni Esercizi su rappresentazione binaria dell informazione
 Soluzioni Esercizi su rappresentazione binaria dell informazione Mauro Bianco 1 Numeri naturali Esercizi: 1. Si calcoli 323 4 + 102 4. Partendo da destra a sinistra 2 4 + 3 4 5 10 4 + 1 10 11 4. La cifra
Soluzioni Esercizi su rappresentazione binaria dell informazione Mauro Bianco 1 Numeri naturali Esercizi: 1. Si calcoli 323 4 + 102 4. Partendo da destra a sinistra 2 4 + 3 4 5 10 4 + 1 10 11 4. La cifra
Copyright Esselibri S.p.A.
 ..3. Prodotti notevoli Per quanto visto in precedenza, in generale per moltiplicare un polinomio di m termini per uno di n termini devono effettuarsi m n moltiplicazioni, così per esempio per moltiplicare
..3. Prodotti notevoli Per quanto visto in precedenza, in generale per moltiplicare un polinomio di m termini per uno di n termini devono effettuarsi m n moltiplicazioni, così per esempio per moltiplicare
PRODUZIONE DI PROTEINE RICOMBINANTI UNO DEI PRINCIPALI INTERESSI DELLE INDUSTRIE NON RICOMBINANTI DIFFICOLTA DI OTTENERE LE FONTI
 PRODUZIONE DI PROTEINE RICOMBINANTI = UNO DEI PRINCIPALI INTERESSI DELLE INDUSTRIE NON RICOMBINANTI DIFFICOLTA DI OTTENERE LE FONTI SCARSITA DEL PRODOTTO DIFFICOLTA DI PURIFICAZIONE POSSIBILE PERICOLOSITA
PRODUZIONE DI PROTEINE RICOMBINANTI = UNO DEI PRINCIPALI INTERESSI DELLE INDUSTRIE NON RICOMBINANTI DIFFICOLTA DI OTTENERE LE FONTI SCARSITA DEL PRODOTTO DIFFICOLTA DI PURIFICAZIONE POSSIBILE PERICOLOSITA
LOGARITMI. Corso di laurea: BIOLOGIA Tutor: Floris Marta; Max Artizzu PRECORSI DI MATEMATICA. L uguaglianza: a x = b
 Corso di laurea: BIOLOGIA Tutor: Floris Marta; Max Artizzu PRECORSI DI MATEMATICA LOGARITMI L uguaglianza: a x = b nella quale a e b rappresentano due numeri reali noti ed x un incognita, è un equazione
Corso di laurea: BIOLOGIA Tutor: Floris Marta; Max Artizzu PRECORSI DI MATEMATICA LOGARITMI L uguaglianza: a x = b nella quale a e b rappresentano due numeri reali noti ed x un incognita, è un equazione
Scale Logaritmiche. Matematica con Elementi di Statistica a.a. 2015/16
 Scale Logaritmiche Scala Logaritmica: sull asse prescelto (ad esempio, l asse x) si rappresenta il punto di ascissa = 0 0 nella direzione positiva si rappresentano, a distanze uguali fra di loro, i punti
Scale Logaritmiche Scala Logaritmica: sull asse prescelto (ad esempio, l asse x) si rappresenta il punto di ascissa = 0 0 nella direzione positiva si rappresentano, a distanze uguali fra di loro, i punti
R. Cusani, F. Cuomo: Telecomunicazioni - DataLinkLayer: Gestione degli errori, Aprile 2010
 1 11. Data link layer: codici di rilevazione di errore, gestione degli errori La rilevazione di errore Un codice a rilevazione di errore ha lo scopo di permettere al ricevente di determinare se vi sono
1 11. Data link layer: codici di rilevazione di errore, gestione degli errori La rilevazione di errore Un codice a rilevazione di errore ha lo scopo di permettere al ricevente di determinare se vi sono
Rappresentazione dell informazione
 Rappresentazione dell informazione Problema che coinvolge aspetti filosofici Interessa soprattutto distinguere informazioni diverse Con un solo simbolo è impossibile Pertanto l insieme minimo è costituito
Rappresentazione dell informazione Problema che coinvolge aspetti filosofici Interessa soprattutto distinguere informazioni diverse Con un solo simbolo è impossibile Pertanto l insieme minimo è costituito
CALCOLO LETTERALE I MONOMI. Il primo tipo di oggetto che incontriamo nel calcolo letterale è il MONOMIO.
 CALCOLO LETTERALE Il calcolo letterale è importante perchè ci consente di realizzare un meccanismo di astrazione fondamentale per l'apprendimento in generale. Scrivere, ad esempio, che l'area di un rettangolo
CALCOLO LETTERALE Il calcolo letterale è importante perchè ci consente di realizzare un meccanismo di astrazione fondamentale per l'apprendimento in generale. Scrivere, ad esempio, che l'area di un rettangolo
Ø Gel Elettroforesi (GE) Ø Spettrometria di Massa (MS)
 Tecniche di indagine nello studio delle PTMs Ø Gel Elettroforesi (GE) Ø Tecniche cromatografiche (LC, HPLC) Ø Spettrometria di Massa (MS) Possono essere utilizzate da sole o in combinazione, direttamente
Tecniche di indagine nello studio delle PTMs Ø Gel Elettroforesi (GE) Ø Tecniche cromatografiche (LC, HPLC) Ø Spettrometria di Massa (MS) Possono essere utilizzate da sole o in combinazione, direttamente
Struttura degli amminoacidi
 AMMINOACIDI, PEPTIDI E PROTEINE AMMINOACIDI, PEPTIDI E PROTEINE AMMINOACIDI, PEPTIDI E PROTEINE Le proteine sono macromolecole costituite dall unione di un grande numero di unità elementari: gli amminoacidi
AMMINOACIDI, PEPTIDI E PROTEINE AMMINOACIDI, PEPTIDI E PROTEINE AMMINOACIDI, PEPTIDI E PROTEINE Le proteine sono macromolecole costituite dall unione di un grande numero di unità elementari: gli amminoacidi
Concentriamo la nostra attenzione sull insieme dei numeri razionali Q. In Q sono definite
 Lezioni del 22 e 24 settembre. Numeri razionali. 1. Operazioni, ordinamento. Indichiamo con N, Z, Q gli insiemi dei numeri naturali, interi relativi, e razionali: N = {0, 1, 2,...} Z = {0, ±1, ±2,...}
Lezioni del 22 e 24 settembre. Numeri razionali. 1. Operazioni, ordinamento. Indichiamo con N, Z, Q gli insiemi dei numeri naturali, interi relativi, e razionali: N = {0, 1, 2,...} Z = {0, ±1, ±2,...}
