Sequenziamento ed analisi con il sistema ION TORRENT
|
|
|
- Erico Vaccaro
- 5 anni fa
- Visualizzazioni
Transcript
1 Obiettivi Sequenziamento ed analisi con il sistema ION TORRENT La piattaforma Ion Torrent di cui dispone Genomnia si compone di due sequenziatori, complementari per capacità e throughput: 1. Ion Personal Genome Machine (PGM) 2. Ion S5 A seconda della profondità di sequenziamento desiderata, dell applicazione scelta e del tipo di sequenziatore, si possono utilizzare chip differenti che permettono di produrre in una sola corsa da un minimo di circa reads fino ad un massimo di milioni. Le caratteristiche di questa piattaforma le conferiscono una versatilità adatta a garantire l esecuzione di differenti applicazioni: SEQUENZIAMENTO MIRATO DI DNA, tramite tecnologia Ampliseq, per lo studio di alterazioni in singoli geni o in pannelli di geni legati ad una patologia; TRASCRITTOMICA (Total RNA-seq, Small RNA-seq, trascrittoma Ampliseq); SEQUENZIAMENTO MIRATO DI RNA, tramite tecnologia Ampliseq, per lo studio dell espressione genica differenziale focalizzata su specifici pathways o gruppi di geni; EPIGENOMICA, per l analisi su scala genomica dei siti di legame per fattori trascrizionali (Chip-seq) o delle regioni arricchite in CpG metilate (MBD-seq); METAGENOMICA; SEQUENZIAMENTO DE NOVO O RI-SEQUENZIAMENTO DI INTERI GENOMI virali o batterici. Il SEQUENZIAMENTO MIRATO DI DNA si esegue quando è nota a priori la sequenza di specifici geni di interesse, siano essi pochi o alcune migliaia come nel caso dell analisi dell esoma umano. Il sequenziamento mirato di specifiche regioni può essere utilizzato per valutare la presenza di snv/indel in sequenze genomiche potenzialmente connesse all insorgenza di malattie ereditarie a carattere mendeliano o di patologie somatiche, quali il cancro. Le regioni genomiche di interesse vengono dapprima amplificate mediante PCR e quindi usate come templato nel processo di preparazione delle librerie. Questa metodica si basa sulla tecnologia Ion AmpliSeq TM che, utilizzando in multiplex diverse centinaia di coppie di primers, permette di selezionare, amplificandole contemporaneamente, tutte le regioni di DNA bersaglio; i prodotti di amplificazione così ottenuti sono successivamente sequenziati su piattaforma Ion Torrent. Il sistema Ion AmpliSeq TM richiede come input quantitativi di DNA piuttosto bassi (da 1 a 100 ng a seconda dell applicazione), permettendo quindi il sequenziamento di campioni disponibili in esigue quantità di partenza (es. biopsie). Le ridotte dimensioni degli ampliconi, inoltre, rendono il sistema adatto anche per l amplificazione di campioni di DNA parzialmente degradato, come quello estratto da materiale biologico conservato in paraffina (FFPE). Il sequenziamento dell esoma, disponibile solo per il DNA umano, è un sequenziamento mirato delle sole regioni codificanti del genoma, che pur rappresentandone solo l 1% contengono circa l 85% delle mutazioni causative di malattie. Lo studio dell esoma permette quindi di identificare singole varianti nucleotidiche (SNVs), variazioni del numero di copie (CNVs) piccole inserzioni e delezioni (indels), ma anche mutazioni nuove e non ancora caratterizzate alla base di malattie ereditarie a carattere mendeliano o complesse. Esistono inoltre pannelli a DNA specifici per determinati set di geni, sia pre-disegnati (ready to use) da Life Technologies che realizzati su misura da Genomnia. I pannelli disponibili si suddividono in Hot-Spot, che permettono di esaminare regioni non contigue del genoma alla ricerca di SNP già noti, isolati o organizzati in gruppi, e pannelli per il risequenziamento di intere regioni geniche contigue, alla ricerca di mutazioni sia già note che ancora sconosciute. Per la costruzione di pannelli personalizzati (custom) si utilizza il software Ion AmpliSeq Designer e le informazioni contenute in database di riferimento quali COSMIC (Catalogue of Somatic Mutations in Cancer) per i geni legati al cancro, NCBI ClinVar, per le mutazioni associate a malattie ereditarie, e dbsnp, per una collezione di tutte le alterazioni geniche note. I pannelli personalizzati a DNA permettono il sequenziamento mirato di regioni genomiche di interesse dei genomi di diversi organismi, quali uomo, topo, maiale, pecora, mucca, pollo, mais e altri ancora. È inoltre possibile eseguire l upload della sequenza di riferimento dell organismo d interesse, se non già presente. I primers sono raggruppati in uno o più pool di amplificazione multiplexed; e il numero dei pool è funzione della complessità e delle dimensioni della regione genomica da analizzare.
2 Per dettagli sulla TRASCRITTOMICA, che comprende le analisi di Total RNA-seq e Small RNA-seq, si rimanda alla scheda specifica. E inoltre possibile realizzare l analisi del trascrittoma utilizzando la tecnologia Ampliseq, attualmente disponibile sia per sequenze umane che per sequenze murine. Il trascrittoma Ampliseq permette di valutare i livelli di espressione genica grazie ad un singolo pannello di primers progettati per consentire l interrogazione in multiplexing di più di geni (oltre il 95% dei geni RefSeq di Homo sapiens e oltre il 90% per Mus musculus). Il disegno del pannello, composto da piccoli ampliconi (uno per gene), permette di ottenere ottimi risultati anche utilizzando RNA estratti da tessuti fissati, quali campioni fissati in formalina e inclusi in paraffina (FFPE). Il protocollo sviluppato per questa applicazione permette di evitare la selezione dell RNA poly(a) o l eliminazione dell RNA ribosomiale prevedendo l utilizzo come input di campioni di RNA totale, inizialmente retrotrascritto con primers random. La quantità di RNA totale normalmente richiesta è di soli 10 ng, ma, in caso di particolari necessità, è possibile eseguire la procedura anche a partire da quantità inferiori. Il SEQUENZIAMENTO MIRATO DI RNA tramite tecnologia Ampliseq si basa sullo stesso principio descritto per il trascrittoma Ampliseq al paragrafo precedente. Pannelli di primers amplificano in multiplex un solo amplicone specifico per ognuno dei geni bersaglio, permettendo l analisi contemporanea dei livelli di espressione di centinaia o migliaia di geni selezionati. Anche in questo caso sono disponibili pannelli ready-to-use e pannelli personalizzati che Genomnia può creare ad hoc utilizzando il software Ion AmpliSeq Designer. Dove possibile, l amplicone disegnato su ognuno dei geni bersaglio copre una giunzione esone-esone, così da minimizzare l amplificazione di eventuali tracce di DNA genomico. Inoltre, la regione target è scelta tra quelle presenti nei trascritti più rappresentati per ogni singolo gene RefSeq. Con la stessa tecnologia di sequenziamento mirato di RNA è possibile analizzare anche la presenza nel trascrittoma di fusioni geniche specifiche. La lista completa ed aggiornata dei pannelli a DNA e a RNA attualmente disponibili in commercio è consultabile registrandosi al sito Le analisi di EPIGENOMICA offerte da Genomnia comprendono ChIP-seq, per lo studio su scala genomica di specifiche interazioni DNA-proteina, e MBD-seq, per lo studio delle regioni genomiche arricchite in CpG metilate (riferirsi alla scheda specifica). Con l analisi ChIP-seq è possibile determinare la distribuzione nel genoma dei siti di legame di fattori trascrizionali o delle modificazioni epigenetiche a carico degli istoni. La cromatina dei campioni, nativa o fissata con formaldeide, viene frammentata per digestione enzimatica o per sonicazione, e quindi immunoprecipitata con anticorpi specifici per la proteina o la modifica epigenetica di interesse. I frammenti di DNA contenuti nei complessi cromatinici immunoprecipitati vengono poi estratti e ligati ad adattatori specifici per la successiva preparazione della libreria. Il mappaggio delle reads ottenute dal sequenziamento della libreria fornisce le coordinate genomiche delle regioni a cui si trovavano associate le molecole del fattore trascrizionale di interesse o della modificazione istonica in esame. L analisi di MBD-seq permette invece di determinare la distribuzione genomica delle regioni più ricche in CpG metilate. I campioni di DNA vengono frammentati tramite sonicazione e quindi sottoposte ad un passaggio di selezione mediante pull-down con biglie magnetiche funzionalizzate con il methyl binding domain della proteina umana MBD. Tale funzionalizzazione permette il legame delle regioni CpG metilate, consentendo la conseguente selezione dei frammenti genomici con livello più alto di metilazione. I frammenti ottenuti vengono usati per costruire librerie il cui sequenziamento consente di mappare sul genoma le regioni con livello di metilazione CpG elevato. Sia ChIP-seq che MBD-seq forniscono informazioni quantitative e permettono quindi di confrontare campioni diversi, eseguendo analisi differenziali. L analisi METAGENOMICA di popolazioni miste di microorganismi può essere eseguita mediante sequenziamento delle regioni ipervariabili del gene codificante per l RNA ribosomiale 16S e permette di identificare le differenti specie batteriche presenti nel campione. Due set di primers disegnati per coprire, rispettivamente, le regioni ipervariabili V2, V4 e V8, e V3, V6-7 e V9 del 16S rdna, permettono l identificazione di un ampio spettro di batteri, a livello di famiglia, di genere e in certi casi anche di specie. Il SEQUENZIAMENTO DI INTERI GENOMI virali e batterici e il sequenziamento de novo di microrganismi, può essere eseguito al fine di identificare nuove specie o di monitorare le condizioni di salute e di malattia o di valutare la presenza di specifiche contaminazioni ambientali, tipizzando genomi di virus e batteri noti.
3 Costruzione delle librerie Il procedimento per la costruzione delle librerie AmpliSeq è comune per campioni di DNA e di RNA. Come mostrato in figura, se il materiale di partenza è costituito da campioni di RNA viene eseguito un iniziale passaggio di retrotrascrizione in cdna. Le regioni bersaglio del DNA genomico o del cdna sono amplificate in multiplex utilizzando pools di coppie di primers specifici. Gli ampliconi ottenuti vengono quindi trattati enzimaticamente in modo da digerire parzialmente i primers di amplificazione e fosforilare le estremità degli ampliconi stessi. Quindi, alle due estremità di ciascun amplicone vengono ligati covalentemente adattatori specifici (A/P1), richiesti per il sequenziamento con piattaforma Ion Torrent. In questa fase è possibile sostituire l adattatore A con adattatori dotati di brevi sequenze di DNA differenti che fungono da codici a barre per l identificazione univoca dei diversi campioni in caso di sequenziamento in multiplex. I prodotti della ligazione vengono purificati con biglie magnetiche Agencourt AMPure XP (SPRI technology) e sottoposti ad amplificazione tramite PCR, allestita in modo da selezionare solo le molecole dotate degli adattatori ad entrambe le estremità. Le librerie così ottenute vengono sottoposte ad analisi quali-quantitativa mediante fluorimetria (Qubit) o microelettroforesi capillare (Agilent Bioanalyzer). Le librerie sono quindi pronte ad essere sequenziate, singolarmente o in multiplex. La procedura eseguita per la costruzione di librerie di frammenti prevede l iniziale frammentazione del DNA genomico mediante digestione enzimatica o per sonicazione, per ottenere frammenti omogenei della grandezza desiderata. In alcuni casi (es. ChIP-seq), il campione di DNA frammentato può essere il prodotto di specifici passaggi di lavorazione precedenti. Le estremità dei frammenti vengono riparate per essere poi unite covalentemente agli adattatori specifici mediante trattamento con ligasi. Le molecole della libreria così ottenute sono quindi sottoposte a selezione dimensionale e amplificate tramite PCR per ottenere la quantità di DNA necessaria a procedere con la preparazione del templato sulle particelle IonSphere e con il successivo sequenziamento. Come già accennato, librerie di frammenti possono essere preparate anche a partire dal DNA estratto da frammenti di cromatina immunoprecipitata con anticorpi specifici (ChIPseq) o da campioni di DNA arricchito per regioni CpG metilate tramite MBD pull-down. Appartengono a questa categoria di librerie anche quelle utilizzate negli studi di metagenomica. In particolare, le regioni ipervariabili del 16S rdna sono amplificate con primer specifici e i prodotti di PCR così ottenuti sono usati per la costruzione di libreria di frammenti secondo la procedura sopra descritta.
4 Caratteristiche quali-quantitativi dei campioni La quantità e qualità richiste per i campioni di partenza variano a seconda dell applicazione e del tipo di controllo eseguito sul campione stesso prima di procedere con la costruzione della libreria. Le quantità richieste per la costruzione della libreria con i sistemi AmpliSeq sono comprese tra 1 e 100 ng di acidi nucleici. È tuttavia preferibile disporre di quantità di partenza maggiori, in modo da poter eseguire anche un controllo di qualità iniziale. Per le applicazioni che comportano la preparazione di una libreria di frammenti sono richiesti da 10 ng ad 1 µg di DNA. Maggiori dettagli saranno specificati in fase di progettazione del servizio. Analisi bioinformatica Le analisi bioinformatiche sono differenziate per applicazione, ma, in generale, per i progetti riferiti all uomo o ad organismi modello, partono tutte dal mappaggio delle read di sequenza sul genoma di riferimento in maniera non ambigua, ad alta precisione e quantificabile con sicurezza. Il segnale di sequenza (che sia mutazionale, epigenetico o trascrittomico) è poi quantificato e filtrato. Sono utilizzati approcci statistici per la valutazione degli aspetti differenziali (analisi della trascrizione, del controllo trascrizionale epigenetico) o per la descrizione del panorama delle variazioni genomiche (SNP, inserzioni/delezioni). Infine, i risultati sono annotati con riferimento all annotazione della struttura genica nei suoi aspetti di base (esoni, introni, inizio della trascrizione, promotori, ecc) e in relazione ad elementi di annotazione avanzata specifici per il tipo di applicazione. I risultati dell analisi delle mutazioni (da pannelli di geni o esomi interi) vengono analizzati con algoritmi integrati di predizione funzionale e confrontati con liste di putativi geni malattia derivati da letteratura, da database o forniti dal cliente. Sempre per l analisi di mutazione, disponiamo di una procedura ad hoc per l analisi di mutazione in trio, particolarmente potente per l identificazione di putativi geni malattia (maggiori dettagli nella relativa scheda servizio). Per quanto riguarda le analisi epigenetiche (ChIP-seq, MBD-seq), le regioni di interesse vengono annotate in relazione alle isole CpG, alle sequenze ripetute e a tracce UCSC relative a modifiche epigenetiche, come la metilazione e l acetilazione di istoni. All analisi MBD-seq è dedicata una scheda servizio specifica. L analisi del trascrittoma con tecnologia Ampliseq consente di quantificare il livello di espressione genica e identificare geni differenzialmente espressi tra due gruppi biologici. All analisi RNA-seq è dedicata una scheda specifica. Per quanto riguarda l analisi degli smallrna, siamo in grado di identificare e quantificare, oltre ai microrna noti, anche le varianti di sequenza e lunghezza (isomir) e i putativi nuovi microrna. Per tutte queste specie molecolari, la procedura analitica si conclude con l analisi differenziale. Infine, per gli approcci legati al mondo della microbiologia proponiamo analisi metagenomiche che permettono non solo di identificare la composizione e quantificare la diversità di campioni singoli, ma anche di confrontare statisticamente le dinamiche temporali o dovute a trattamenti farmacologici e/o biologici in diversi campioni. Informazioni per gli ordini Richiedere ad admin@genomnia.it un offerta per l applicazione di interesse.
5 Trattamenti per DNA e RNA QC: Controllo di qualità e dimensioni di DNA MBD/CHIP arricchito QC: controllo di qualità di preparazioni di DNA QC: controllo di qualità di preparazioni di RNA totale Purificazione di RNA Poly(A) + da RNA totale Arricchimento nella frazione degli Small RNA Deplezione di rrna da RNA totale Arricchimento delle regioni ipervaribili del 16S Arricchimento per MBD da DNA genomico Arricchimento per CHIP da DNA genomico DNA05 DNA06 RNA03 RNA06 RNA07 RNA10 16S MBD CHIP Preparazioni di librerie Disegno e sintesi pannello custom Ampliseq Preparazione di libreria DNA con codice a barre Preparazione di libreria RNA con codice a barre PAN LDb LRb Sequenziamento Sequenziamento forward 200 bp tags con codice a barre Sequenziamento forward 400 bp tags con codice a barre Sequenziamento forward 600 bp tags con codice a barre SEQI200B SEQI400B SEQI600B
6 Analisi Bioinformatica Analisi Bioinformatica I: DNA (pannelli) Analisi Bioinformatica II: DNA (esoma intero) Analisi Bioinformatica III: DNA (esoma intero in trio) Analisi Bioinformatica I: RNA (analisi del trascrittoma) Analisi Bioinformatica II: RNA (isoforme e network) Analisi Bioinformatica I: smallrna (microrna noti) Analisi Bioinformatica II: smallrna (nuovi microrna, isomir e targets) Analisi Bioinformatica I: Metagenomica Analisi Bioinformatica I: De Novo Analisi Bioinformatica I: Metilazione (identificazione delle regioni metilate) Analisi Bioinformatica II: Metilazione (analisi di metilazione differenziale) Analisi Bioinformatica III: Metilazione (analisi differenziale delle LINE/SINE) Analisi Bioinformatica ChIP-Seq I DNA-BF01 DNA-BF02 DNA-BF03 RNA-BF01 RNA-BF02 Small-BF01 Small-BF02 METAGEN-BF01 ASSEMBLY-BF01 MBD-BF01 MBD-BF02 MBD-BF03 CHIP-BF01 ION Torrent Rev. 3 02/2018
CAPITOLATO TECNICO. Kit di controllo qualità delle sfere ioniche template-positive prearricchite e post-arricchite mediante il fluorimetro Qubit (in
 REAGENTI 1 Kit di preparazione PCR in emulsione per sequenziamento Kit per la preparazione, mediante reazione di PCR in emulsione, di 120 test 240 test 163,1 39.144,00 77302685FD sfere ioniche template-positive
REAGENTI 1 Kit di preparazione PCR in emulsione per sequenziamento Kit per la preparazione, mediante reazione di PCR in emulsione, di 120 test 240 test 163,1 39.144,00 77302685FD sfere ioniche template-positive
GENOMA. Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine CONTENUTO FUNZIONE. Progetti genoma in centinaia di organismi
 GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
Analisi bioinformatica di esperimenti di Esoma Intero in trio
 Analisi bioinformatica di esperimenti di Esoma Intero in trio L analisi dell esoma intero applicata ai trio (genitori e figlio/figlia affetto) è un approccio molto potente al fine di identificare mutazioni
Analisi bioinformatica di esperimenti di Esoma Intero in trio L analisi dell esoma intero applicata ai trio (genitori e figlio/figlia affetto) è un approccio molto potente al fine di identificare mutazioni
Patologie da analizzare
 Fasi cruciali Scelta della patologia da analizzare Scelta del campione da analizzare Scelta dell approccio da utilizzare Scelta della tecnica da utilizzare Analisi statistica del dati Conferme con approcci
Fasi cruciali Scelta della patologia da analizzare Scelta del campione da analizzare Scelta dell approccio da utilizzare Scelta della tecnica da utilizzare Analisi statistica del dati Conferme con approcci
Ottimizzazione del protocollo bioinformatico per l annotazione di geni codificanti proteine in genomi complessi. Marin Vargas, Sergio Paul
 Ottimizzazione del protocollo bioinformatico per l annotazione di geni codificanti proteine in genomi complessi Marin Vargas, Sergio Paul 2012 Con l avvento del sequenziamento NGS a costi sempre più contenuti,
Ottimizzazione del protocollo bioinformatico per l annotazione di geni codificanti proteine in genomi complessi Marin Vargas, Sergio Paul 2012 Con l avvento del sequenziamento NGS a costi sempre più contenuti,
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Percorso di Biologia Molecolare
 La Biologia Molecolare è oggi alla base di qualunque indagine analitica in campo clinico, alimentare, ambientale forense e nella ricerca di base. Le Biotecnologie sono in costante e continua evoluzione,
La Biologia Molecolare è oggi alla base di qualunque indagine analitica in campo clinico, alimentare, ambientale forense e nella ricerca di base. Le Biotecnologie sono in costante e continua evoluzione,
Utilizzo di marcatori molecolari in evoluzione e conservazione
 Utilizzo di marcatori molecolari in evoluzione e conservazione Un marcatore genetico è qualsiasi elemento con una base genetica, in genere identificabile con facilità, che permette di caratterizzare un
Utilizzo di marcatori molecolari in evoluzione e conservazione Un marcatore genetico è qualsiasi elemento con una base genetica, in genere identificabile con facilità, che permette di caratterizzare un
IL GENOMA DELLA CELLULA VEGETALE
 IL GENOMA DELLA CELLULA VEGETALE I GENOMI DELLE CELLULE VEGETALI Genoma nucleare Geni per il funzionamento globale della cellula vegetale Condivisi o specifici per la cellula vegetale Genoma plastidiale
IL GENOMA DELLA CELLULA VEGETALE I GENOMI DELLE CELLULE VEGETALI Genoma nucleare Geni per il funzionamento globale della cellula vegetale Condivisi o specifici per la cellula vegetale Genoma plastidiale
Studio della regolazione dell espressione genica
 Studio della regolazione dell espressione genica Un gene soggetto a regolazione possiede una o più sequenze regolatorie posizionate in maniera diversa rispetto al gene. I regolatori possono essere positivi
Studio della regolazione dell espressione genica Un gene soggetto a regolazione possiede una o più sequenze regolatorie posizionate in maniera diversa rispetto al gene. I regolatori possono essere positivi
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Frontiere della Biologia Molecolare
 Prof. Giorgio DIECI Dipartimento di Bioscienze Università degli Studi di Parma Frontiere della Biologia Molecolare Milano, 4 marzo 2016 Fotografia al microscopio elettronico di una plasmacellula NUCLEO
Prof. Giorgio DIECI Dipartimento di Bioscienze Università degli Studi di Parma Frontiere della Biologia Molecolare Milano, 4 marzo 2016 Fotografia al microscopio elettronico di una plasmacellula NUCLEO
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Ibridazione in situ per mrna: tecnica e interpretazione
 Patologia nodulare della tiroide: focus sugli approcci diagnostici Ibridazione in situ per mrna: tecnica e interpretazione Isabella Giovannoni U.O.C. Anatomia Patologica Ibridazione -> appaiamento di sequenze
Patologia nodulare della tiroide: focus sugli approcci diagnostici Ibridazione in situ per mrna: tecnica e interpretazione Isabella Giovannoni U.O.C. Anatomia Patologica Ibridazione -> appaiamento di sequenze
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Marcatori molecolari per l analisi genica, genetica e genomica
 Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Banche Dati proteiche
 Banche Dati proteiche Un altro grande database è UniProt, The Universal Protein Resource (http://www.uniprot.org/) nel quale sono radunate le sequenze proteiche, e le annotazione delle stesse, ottenute
Banche Dati proteiche Un altro grande database è UniProt, The Universal Protein Resource (http://www.uniprot.org/) nel quale sono radunate le sequenze proteiche, e le annotazione delle stesse, ottenute
LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI
 CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
Indice generale. Nozioni fondamentali. Prefazione XIII
 Prefazione XIII A Nozioni fondamentali CAPITOLO 1 La biologia essenziale 3 1.1 Genomi, genomica e avvento della Bioinformatica 3 1.2 Genoma dei procarioti 5 1.2.1 Struttura e dimensioni 5 1.2.2 Proprietà
Prefazione XIII A Nozioni fondamentali CAPITOLO 1 La biologia essenziale 3 1.1 Genomi, genomica e avvento della Bioinformatica 3 1.2 Genoma dei procarioti 5 1.2.1 Struttura e dimensioni 5 1.2.2 Proprietà
Importanza della genetica dei microrganismi
 Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Nucleosomi e trascrizione: Rimodellamento cromatinico e regolazione dell'espressione genica
 Nucleosomi e trascrizione: Rimodellamento cromatinico e regolazione dell'espressione genica La trascrizione di un gene richiede delle modifiche nell'organizzazione della cromatina Figura 4.15C Organizzazione
Nucleosomi e trascrizione: Rimodellamento cromatinico e regolazione dell'espressione genica La trascrizione di un gene richiede delle modifiche nell'organizzazione della cromatina Figura 4.15C Organizzazione
Caratteristiche prestazionali
 Caratteristiche prestazionali Kit RNeasy DSP FFPE, Versione 1 73604 Gestione della versione Il presente è il documento Caratteristiche prestazionali del kit RNeasy DSP FFPE, Versione 1, R1. Prima di eseguire
Caratteristiche prestazionali Kit RNeasy DSP FFPE, Versione 1 73604 Gestione della versione Il presente è il documento Caratteristiche prestazionali del kit RNeasy DSP FFPE, Versione 1, R1. Prima di eseguire
Corso di BioMedicina Molecolare Genomica e dei Sistemi Complessi
 Corso di BioMedicina Molecolare Genomica e dei Sistemi Complessi CFU: 6 Anno accademico 2010-2011 Logica del Corso Il completamento del Progetto Genoma di Homo sapiens e di molti altri organismi e virus
Corso di BioMedicina Molecolare Genomica e dei Sistemi Complessi CFU: 6 Anno accademico 2010-2011 Logica del Corso Il completamento del Progetto Genoma di Homo sapiens e di molti altri organismi e virus
Genomica, proteomica, genomica strutturale, banche dati.
 Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomi dei procarioti
 Genomi dei procarioti Una molecola circolare di DNA E.coli circa 4 x 10 6 coppie di basi Il genoma è quasi tutto codificante Viene trascritto in mrna policistronici Il genoma eucariotico Il genoma eucariotico
Genomi dei procarioti Una molecola circolare di DNA E.coli circa 4 x 10 6 coppie di basi Il genoma è quasi tutto codificante Viene trascritto in mrna policistronici Il genoma eucariotico Il genoma eucariotico
Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti
 Seminario per il corso misure con biomarcatori A.A. 2005-2006 Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti Perché utilizzare
Seminario per il corso misure con biomarcatori A.A. 2005-2006 Il biomonitoraggio ambientale attraverso lo studio della regolazione genica della risposta allo stress Silvia Franzellitti Perché utilizzare
Bioinformatica. Marin Vargas, Sergio Paul
 Bioinformatica Marin Vargas, Sergio Paul 2014 Wikipedia: La bioinformatica è una disciplina scientifica dedicata alla risoluzione di problemi biologici a livello molecolare con metodi informatici. La bioinformatica
Bioinformatica Marin Vargas, Sergio Paul 2014 Wikipedia: La bioinformatica è una disciplina scientifica dedicata alla risoluzione di problemi biologici a livello molecolare con metodi informatici. La bioinformatica
TRASCRIZIONE DEL DNA. Formazione mrna
 TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
Jay Phelan, Maria Cristina Pignocchino. Scopriamo la biologia
 Jay Phelan, Maria Cristina Pignocchino Scopriamo la biologia Capitolo 6 Il DNA in azione 3 1. Il DNA è il materiale genetico Il DNA è composto da una sequenza di nucleotidi. Ogni nucleotide comprende:
Jay Phelan, Maria Cristina Pignocchino Scopriamo la biologia Capitolo 6 Il DNA in azione 3 1. Il DNA è il materiale genetico Il DNA è composto da una sequenza di nucleotidi. Ogni nucleotide comprende:
Applicazione della biologia molecolare nella valutazione del benessere del cavallo
 UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
GRANDE OSPEDALE METROPOLITANO Bianchi Melacrino Morelli Reggio Calabria REGIONE CALABRIA
 ALLEGATO A LOTTO 1 Estrazione DNA da tessuti paraffinati con biglie magnetiche. Tecnologia basata su biglie magnetiche con reagenti pronti all'uso monodose Sparaffinatura dei campioni compresa nel kit
ALLEGATO A LOTTO 1 Estrazione DNA da tessuti paraffinati con biglie magnetiche. Tecnologia basata su biglie magnetiche con reagenti pronti all'uso monodose Sparaffinatura dei campioni compresa nel kit
È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale
 ? what is ChIP?? È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale E uno strumento potente per ottenere immagini ad alta risoluzione di proteine nel contesto
? what is ChIP?? È un metodo in vivo per analizzare qualsiasi componente della cromatina nel suo contesto naturale E uno strumento potente per ottenere immagini ad alta risoluzione di proteine nel contesto
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
La regolazione genica negli eucarioti
 La regolazione genica negli eucarioti neuroni globuli rossi globulo bianco fibroblasti adipociti Sezione di testicolo Surrene Come mai alcuni geni sono trascritti e tradotti in alcune cellule ma non in
La regolazione genica negli eucarioti neuroni globuli rossi globulo bianco fibroblasti adipociti Sezione di testicolo Surrene Come mai alcuni geni sono trascritti e tradotti in alcune cellule ma non in
GENETICA DELLE MALATTIE COMPLESSE. Dr. Giovanni Malerba Biologia e Genetica, UniVR
 GENETICA DELLE MALATTIE COMPLESSE Dr. Giovanni Malerba Biologia e Genetica, UniVR giovanni.malerba@univr.it CARATTERE COMPLESSO Predisposizione Genetica interazione Diagnosi Prevenzione Terapia efficiente
GENETICA DELLE MALATTIE COMPLESSE Dr. Giovanni Malerba Biologia e Genetica, UniVR giovanni.malerba@univr.it CARATTERE COMPLESSO Predisposizione Genetica interazione Diagnosi Prevenzione Terapia efficiente
Il flusso e la regolazione dell informazione genica. Lezione nr. 8 Psicobiologia
 Il flusso e la regolazione dell informazione genica Lezione nr. 8 Psicobiologia L informazione che viene trascritta non riguarda tutto il DNA ma solo delle particolari sequenze definite GENI. Tipologie
Il flusso e la regolazione dell informazione genica Lezione nr. 8 Psicobiologia L informazione che viene trascritta non riguarda tutto il DNA ma solo delle particolari sequenze definite GENI. Tipologie
Biologia dei sistemi
 Biologia dei sistemi Fino poco tempo fa i ricercatori studiavano l espressione di un singolo gene alla volta Ora è possibile studiare simultaneamente l espressione di tutti i geni di un organismo (questo
Biologia dei sistemi Fino poco tempo fa i ricercatori studiavano l espressione di un singolo gene alla volta Ora è possibile studiare simultaneamente l espressione di tutti i geni di un organismo (questo
Database di sequenze. Dati di sequenza. Caratteristiche dei dati della biologia molecolare. I dati ed i problemi della bioinformatica
 I dati ed i problemi della bioinformatica Giorgio Valentini DSI Università degli Studi di Milano 1 Caratteristiche dei dati della biologia molecolare Diverse tipologie di dati bio-molecolari Per ogni tipo
I dati ed i problemi della bioinformatica Giorgio Valentini DSI Università degli Studi di Milano 1 Caratteristiche dei dati della biologia molecolare Diverse tipologie di dati bio-molecolari Per ogni tipo
Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico Padano Pietro Piffanelli, Pamela Abbruscato, barbara Menin
 Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Interazioni proteina-dna
 Interazioni proteina-dna 1) Proteine che legano la doppia elica del DNA in maniera non sequenza-specifica: histone-like proteins (HU protein) 2) Proteine che legano strutture particolari del DNA: - single
Interazioni proteina-dna 1) Proteine che legano la doppia elica del DNA in maniera non sequenza-specifica: histone-like proteins (HU protein) 2) Proteine che legano strutture particolari del DNA: - single
Gli rrnas sono gli RNAs più abbondanti nelle cellule. Nelle cellule in attiva proliferazione rappresentano l 80% dell RNA totale
 Gli rrnas sono gli RNAs più abbondanti nelle cellule. Nelle cellule in attiva proliferazione rappresentano l 80% dell RNA totale I geni che codificano gli rrna sono presenti in copia multipla nel genoma
Gli rrnas sono gli RNAs più abbondanti nelle cellule. Nelle cellule in attiva proliferazione rappresentano l 80% dell RNA totale I geni che codificano gli rrna sono presenti in copia multipla nel genoma
L organizzazione del genoma. Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
 L organizzazione del genoma L organizzazione del genoma Fino ad ora abiamo studiato la regolazione dell espressione genica prendendo come esempio singoli geni dei batteri. Ma quanti geni ci sono in un
L organizzazione del genoma L organizzazione del genoma Fino ad ora abiamo studiato la regolazione dell espressione genica prendendo come esempio singoli geni dei batteri. Ma quanti geni ci sono in un
Corso di Laurea in Chimica e Tecnologie Farmaceu6che a.a Università di Catania. La stru(ura del gene. Stefano Forte
 Corso di Laurea in Chimica e Tecnologie Farmaceu6che a.a. 2014-2015 Università di Catania La stru(ura del gene Stefano Forte I Geni Il gene è l'unità ereditaria e funzionale degli organismi viventi. La
Corso di Laurea in Chimica e Tecnologie Farmaceu6che a.a. 2014-2015 Università di Catania La stru(ura del gene Stefano Forte I Geni Il gene è l'unità ereditaria e funzionale degli organismi viventi. La
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
BIOCHIMICA MOLECOLARE CLINICA
 Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
lezione martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
lezione 15-16 martedì 6 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cdna Se si vuole identificare
Bioinformatica. :studio dei problemi biologici attraverso le metodologie dell'informatica
 Bioinformatica :studio dei problemi biologici attraverso le metodologie dell'informatica Sinomimi: biochimica computazionale, biologia molecolare computazionale Viceversa: Biocomputazione, algoritmi genetici,
Bioinformatica :studio dei problemi biologici attraverso le metodologie dell'informatica Sinomimi: biochimica computazionale, biologia molecolare computazionale Viceversa: Biocomputazione, algoritmi genetici,
Una completa descrizione della sequenza di DNA di un organismo non sarebbe in grado di ricostruire lo stesso organismo più di quanto una lista di voca
 REGOLAZIONE DELLA ESPRESSIONE GENICA Una completa descrizione della sequenza di DNA di un organismo non sarebbe in grado di ricostruire lo stesso organismo più di quanto una lista di vocaboli inglesi non
REGOLAZIONE DELLA ESPRESSIONE GENICA Una completa descrizione della sequenza di DNA di un organismo non sarebbe in grado di ricostruire lo stesso organismo più di quanto una lista di vocaboli inglesi non
Biologia Molecolare e nuove tecnologie. M.Favarato ULSS13 Mirano (Ve) Medicina di Laboratorio Biologia Molecolare
 Biologia Molecolare e nuove tecnologie M.Favarato ULSS13 Mirano (Ve) Medicina di Laboratorio Biologia Molecolare Identificazione, virulenza, resistenza antibiotici Resistenza antibiotici (4-5 gg) Tipizzazione,
Biologia Molecolare e nuove tecnologie M.Favarato ULSS13 Mirano (Ve) Medicina di Laboratorio Biologia Molecolare Identificazione, virulenza, resistenza antibiotici Resistenza antibiotici (4-5 gg) Tipizzazione,
Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato
 Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato Lo strumento è composto da:! Un sistema ottico per eccitare il marcatore fluorescente
Permette di monitorare in tempo reale l andamento della reazione ed effettuare una quantizzazione del frammento amplificato Lo strumento è composto da:! Un sistema ottico per eccitare il marcatore fluorescente
Modello computazionale per la predizione di siti di legame per fattori di trascrizione
 Modello computazionale per la predizione di siti di legame per fattori di trascrizione Attività di tirocinio svolto presso il Telethon Institute of Genetics and Medicine Relatori Prof. Giuseppe Trautteur
Modello computazionale per la predizione di siti di legame per fattori di trascrizione Attività di tirocinio svolto presso il Telethon Institute of Genetics and Medicine Relatori Prof. Giuseppe Trautteur
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
UNIVERSITÀ DEGLI STUDI DI NAPOLI FEDERICO II DIPARTIMENTO DI AGRARIA Via Università, PORTICI (NA) Italia RELAZIONE FINALE
 RELAZIONE FINALE Convenzione di Ricerca: ACCERTAMENTO, MEDIANTE METODI MOLECOLARI, DELLA DIVERSITÀ MICROBICA RICORRENTE IN IMPASTI ACIDI IMPIEGATI PER LA PRODUZIONE DI PRODOTTI DA FORNO A LIEVITAZIONE
RELAZIONE FINALE Convenzione di Ricerca: ACCERTAMENTO, MEDIANTE METODI MOLECOLARI, DELLA DIVERSITÀ MICROBICA RICORRENTE IN IMPASTI ACIDI IMPIEGATI PER LA PRODUZIONE DI PRODOTTI DA FORNO A LIEVITAZIONE
David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis. Biologia La scienza della vita
 1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione La regolazione genica negli eucarioti 3 I genomi
1 David Sadava, H. Craig Heller, Gordon H. Orians, William K. Purves, David M. Hillis Biologia La scienza della vita 2 B - L ereditarietà e l evoluzione La regolazione genica negli eucarioti 3 I genomi
Introduzione alla Genomica
 Laboratorio di Bioinformatica I Introduzione alla Genomica Dott. Sergio Marin Vargas (2014 / 2015) Il Genoma umano Gene codificanti proteine Gene non codificanti proteine Geni codificanti proteine 3 Il
Laboratorio di Bioinformatica I Introduzione alla Genomica Dott. Sergio Marin Vargas (2014 / 2015) Il Genoma umano Gene codificanti proteine Gene non codificanti proteine Geni codificanti proteine 3 Il
Rilevamento e tracciabilità dei prodotti delle NBT
 Incontro tecnico-scientifico sulle nuove tecniche di miglioramento genetico in agricoltura Roma 22 febbraio 2018 Sala Polifunzionale della Presidenza del Consiglio dei Ministri Ugo Marchesi Rilevamento
Incontro tecnico-scientifico sulle nuove tecniche di miglioramento genetico in agricoltura Roma 22 febbraio 2018 Sala Polifunzionale della Presidenza del Consiglio dei Ministri Ugo Marchesi Rilevamento
Lo studio delle comunità microbiche. È stato reso possibile dalle Tecniche molecolari
 Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
microrna Struttura e Funzione
 microrna Struttura e Funzione Cinzia Di Pietro Università degli Studi di Catania Dipartimento di Scienze Biomediche e Biotecnologiche Sezione di Biologia e Genetica G. Sichel I MicroRNAs (mirnas) sono
microrna Struttura e Funzione Cinzia Di Pietro Università degli Studi di Catania Dipartimento di Scienze Biomediche e Biotecnologiche Sezione di Biologia e Genetica G. Sichel I MicroRNAs (mirnas) sono
Relatrice: dott.ssa Ilaria Pegoretti
 Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
Relatrice: dott.ssa Ilaria Pegoretti IL LABORATORIO DI BIOLOGIA MOLECOLARE: introduzione alle tecniche e alle loro applicazioni 26 Novembre 2011 Auditorium Presidio Ospedaliero S.Chiara, Trento Scoperta
BIOLOGIA E GENETICA - canale 3
 DIPARTIMENTO DI CHIRURGIA GENERALE E SPECIALITÀ MEDICO-CHIRURGICHE Corso di laurea magistrale in Medicina e chirurgia Anno accademico 2016/2017-1 anno BIOLOGIA E GENETICA - canale 3 BIO/13-10 CFU - 1 semestre
DIPARTIMENTO DI CHIRURGIA GENERALE E SPECIALITÀ MEDICO-CHIRURGICHE Corso di laurea magistrale in Medicina e chirurgia Anno accademico 2016/2017-1 anno BIOLOGIA E GENETICA - canale 3 BIO/13-10 CFU - 1 semestre
Regolazione dell espressione genica
 Regolazione dell espressione genica definizioni Gene attivato quando viene trascritto in RNA e il suo messaggio tradotto in molecole proteiche specifiche Espressione genica processo complessivo con cui
Regolazione dell espressione genica definizioni Gene attivato quando viene trascritto in RNA e il suo messaggio tradotto in molecole proteiche specifiche Espressione genica processo complessivo con cui
PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay)
 Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Sommario. Presentazione dell opera Ringraziamenti
 Sommario Presentazione dell opera Ringraziamenti XI XII Capitolo 1 Introduzione alla bioinformatica 1 1.1 Cenni introduttivi 1 1.2 Pietre miliari della bioinformatica 2 1.3 Infrastrutture bioinformatiche
Sommario Presentazione dell opera Ringraziamenti XI XII Capitolo 1 Introduzione alla bioinformatica 1 1.1 Cenni introduttivi 1 1.2 Pietre miliari della bioinformatica 2 1.3 Infrastrutture bioinformatiche
BIOMEDICINA GENOMICA E DEI SISTEMI COMPLESSI
 DIPARTIMENTO DI SCIENZE BIOLOGICHE, GEOLOGICHE E AMBIENTALI Corso di laurea magistrale in Biologia sanitaria e cellularemolecolare Anno accademico 2018/2019-1 anno - Curriculum Biologia cellulare e molecolare
DIPARTIMENTO DI SCIENZE BIOLOGICHE, GEOLOGICHE E AMBIENTALI Corso di laurea magistrale in Biologia sanitaria e cellularemolecolare Anno accademico 2018/2019-1 anno - Curriculum Biologia cellulare e molecolare
I telomeri. In molti eucarioti le estremità dei cromosomi presentano delle sequenze ripetitive: i telomeri.
 I telomeri 1 I telomeri In molti eucarioti le estremità dei cromosomi presentano delle sequenze ripetitive: i telomeri. A ogni duplicazione la cellula perde una porzione del DNA telomerico, fino a quando
I telomeri 1 I telomeri In molti eucarioti le estremità dei cromosomi presentano delle sequenze ripetitive: i telomeri. A ogni duplicazione la cellula perde una porzione del DNA telomerico, fino a quando
MESSA A PUNTO DI UN KIT DIAGNOSTICO MOLECOLARE UNIVERSALE PER LA RILEVAZIONE DI PATOGENI IN ALIMENTI E ACQUE
 MESSA A PUNTO DI UN KIT DIAGNOSTICO MOLECOLARE UNIVERSALE PER LA RILEVAZIONE DI PATOGENI IN ALIMENTI E ACQUE Regione del Veneto - POR FESR 2014-2020 BANDO PER IL SOSTEGNO A PROGETTI DI RICERCA CHE PREVEDONO
MESSA A PUNTO DI UN KIT DIAGNOSTICO MOLECOLARE UNIVERSALE PER LA RILEVAZIONE DI PATOGENI IN ALIMENTI E ACQUE Regione del Veneto - POR FESR 2014-2020 BANDO PER IL SOSTEGNO A PROGETTI DI RICERCA CHE PREVEDONO
Graziella Bongioni, Sabina Arabi e Rossana Capoferri. Istituto Spallanzani
 Utilizzo di metodi molecolari per l identificazione di specie microalgali Rivolta d Adda, 22 giugno 2017 Graziella Bongioni, Sabina Arabi e Rossana Capoferri Istituto Spallanzani Progetto finanziato da:
Utilizzo di metodi molecolari per l identificazione di specie microalgali Rivolta d Adda, 22 giugno 2017 Graziella Bongioni, Sabina Arabi e Rossana Capoferri Istituto Spallanzani Progetto finanziato da:
Corso di Bioinformatica e analisi dei genomi, docente Silvia Fuselli. Esercizi ricerche in banche dati
 Corso di Bioinformatica e analisi dei genomi, docente Silvia Fuselli Esercizi ricerche in banche dati 1) Nel romanzo fantasy Jurassic Park di Michael Crichton sulla possibilità di clonare i dinosauri,
Corso di Bioinformatica e analisi dei genomi, docente Silvia Fuselli Esercizi ricerche in banche dati 1) Nel romanzo fantasy Jurassic Park di Michael Crichton sulla possibilità di clonare i dinosauri,
Analisi denitrificazione del suolo. Dipartimento Biotecnologie Agrarie Università di Padova
 Agripolis Analisi denitrificazione del suolo Analisi denitrificazione del suolo del suolo del suolo del suolo del suolo Inoltre -Quantificazione CFU - Caratterizzazione molecolare CFU - Valutazione vitali
Agripolis Analisi denitrificazione del suolo Analisi denitrificazione del suolo del suolo del suolo del suolo del suolo Inoltre -Quantificazione CFU - Caratterizzazione molecolare CFU - Valutazione vitali
Cenni di genetica di popolazione delle specie native
 Ariis di Rivignano, 8 Novembre Cenni di genetica di popolazione delle specie native prof. Pallavicini 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Ariis di Rivignano, 8 Novembre Cenni di genetica di popolazione delle specie native prof. Pallavicini 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Diapositiva preparata da SABRINA PRUDENTE e ceduta alla Società Italiana di Diabetologia. Per ricevere la versione originale si prega di scrivere a
 Developmental Origins of Health And Disease (DOHAD) Il XX secolo è stato segnato da una delle più importanti e rivoluzionarie osservazioni in campo epidemiologico: l ipotesi di Barker. Questa ipotesi si
Developmental Origins of Health And Disease (DOHAD) Il XX secolo è stato segnato da una delle più importanti e rivoluzionarie osservazioni in campo epidemiologico: l ipotesi di Barker. Questa ipotesi si
ready to use - caratteristiche principali
 Ready to youse Ready to youse ready to use - caratteristiche principali Pronto all uso: tutti i reagenti necessari, completi di mastermix sono pre-aliquotati in strip pronte all uso. Le strip possono essere
Ready to youse Ready to youse ready to use - caratteristiche principali Pronto all uso: tutti i reagenti necessari, completi di mastermix sono pre-aliquotati in strip pronte all uso. Le strip possono essere
Rivelazione, identificazione, caratterizzazione strutturale
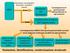 HPLC Separazione cromatografica di proteine e peptidi in miscela Eluato Rivelatore UV (fotodiodi) Cromatogramma UV (picchi generati dall assorbimento di ogni proteina) Eluato Spettrometro di massa ESI-Trappola
HPLC Separazione cromatografica di proteine e peptidi in miscela Eluato Rivelatore UV (fotodiodi) Cromatogramma UV (picchi generati dall assorbimento di ogni proteina) Eluato Spettrometro di massa ESI-Trappola
1 Gene => + fenotipi. ambiente
 1 Gene => + fenotipi (Fattori di) trascrizione Cromatina (etero,eu, remodeling) Splicing Silenziamento (Interference) Modificazioni post traduzionali ambiente Lattosio nel mezzo Ormoni Heat shock Luce
1 Gene => + fenotipi (Fattori di) trascrizione Cromatina (etero,eu, remodeling) Splicing Silenziamento (Interference) Modificazioni post traduzionali ambiente Lattosio nel mezzo Ormoni Heat shock Luce
RELAZIONE di BIOLOGIA MOLECOLARE
 NOME: Marini Selena MATRICOLA: 592330 RELAZIONE di BIOLOGIA MOLECOLARE CHE ORGANISMO MODELLO È DICTYOSTELIUM? CHE RISORSE BIOINFORMATICHE AGEVOLANO I RICERCATORI CHE LO STUDIANO? Dictyostelium è un genere
NOME: Marini Selena MATRICOLA: 592330 RELAZIONE di BIOLOGIA MOLECOLARE CHE ORGANISMO MODELLO È DICTYOSTELIUM? CHE RISORSE BIOINFORMATICHE AGEVOLANO I RICERCATORI CHE LO STUDIANO? Dictyostelium è un genere
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
ESPRESSIONE DEL GENOMA CORSO DI BIOLOGIA, PER OSTETRICIA
 ESPRESSIONE DEL GENOMA CORSO DI BIOLOGIA, PER OSTETRICIA IL DOGMA CENTRALE DELLA BIOLOGIA TRASCRIZIONE DEL DNA E TRADUZIONE DELL RNA ESPRESSIONE DEL DNA - Solo una frazione minore del DNA presente nelle
ESPRESSIONE DEL GENOMA CORSO DI BIOLOGIA, PER OSTETRICIA IL DOGMA CENTRALE DELLA BIOLOGIA TRASCRIZIONE DEL DNA E TRADUZIONE DELL RNA ESPRESSIONE DEL DNA - Solo una frazione minore del DNA presente nelle
Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna Istituto Superiore di Sanità
 Piani per la Sicurezza delle Acque Nuove metodologie di analisi ed inquinanti emergenti Bologna, 12 ottobre 2017 Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna
Piani per la Sicurezza delle Acque Nuove metodologie di analisi ed inquinanti emergenti Bologna, 12 ottobre 2017 Microbiologia Controlli batteriologici con test del DNA e test di tossicità Lucia Bonadonna
LA TRASCRIZIONE NEGLI EUCARIOTI
 LA TRASCRIZIONE NEGLI EUCARIOTI NEGLI EUCARIOTI TRASCRIZIONE E TRADUZIONE SONO DUE EVENTI SEPARATI CHE AVVENGONO IN DUE DIVERSI COMPARTIMENTI CELLULARI: NUCLEO E CITOPLASMA. INOLTRE, A DIFFERENZA DEI
LA TRASCRIZIONE NEGLI EUCARIOTI NEGLI EUCARIOTI TRASCRIZIONE E TRADUZIONE SONO DUE EVENTI SEPARATI CHE AVVENGONO IN DUE DIVERSI COMPARTIMENTI CELLULARI: NUCLEO E CITOPLASMA. INOLTRE, A DIFFERENZA DEI
LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE
 LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE Metodi e tecniche per la rilevazione e quantificazione della resistenza Insetticidi ed acaricidi Manicardi
LA RESISTENZA A PRODOTTI FITOSANITARI: UNA SFIDA PER LA MODERNA PROTEZIONE INTEGRATA DELLE COLTURE Metodi e tecniche per la rilevazione e quantificazione della resistenza Insetticidi ed acaricidi Manicardi
NGS 3.0 GUIDA AL MONDO NGS 3.0
 NGS 3.0 GUIDA AL MONDO NGS 3.0 NEXT GENERATION SEQUENCING (NGS) Il Next Generation Sequencing è una tecnologia di sequenziamento high-throughput per l analisi di un altissimo numero di frammenti in parallelo.
NGS 3.0 GUIDA AL MONDO NGS 3.0 NEXT GENERATION SEQUENCING (NGS) Il Next Generation Sequencing è una tecnologia di sequenziamento high-throughput per l analisi di un altissimo numero di frammenti in parallelo.
Relazione finale. Fruitore: Dott. Esposito Teresa Proponente: Dott. Siani Alfonso
 Relazione finale Fruitore: Dott. Esposito Teresa Proponente: Dott. Siani Alfonso Titolo del programma: Genetica dell'emicrania: analisi molecolare di un cluster di geni candidati X-linked (GABRE-GABRA3-GABRQ)
Relazione finale Fruitore: Dott. Esposito Teresa Proponente: Dott. Siani Alfonso Titolo del programma: Genetica dell'emicrania: analisi molecolare di un cluster di geni candidati X-linked (GABRE-GABRA3-GABRQ)
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Progetto Lars-Biotec
 Unità didattiche: prima fase: Progetto Lars-Biotec Laboratorio di Ricerca sperimentale nel settore delle Biotecnologie Bioinformatica: vengono scelti e analizzati geni appartenente al genoma umano conosciuti
Unità didattiche: prima fase: Progetto Lars-Biotec Laboratorio di Ricerca sperimentale nel settore delle Biotecnologie Bioinformatica: vengono scelti e analizzati geni appartenente al genoma umano conosciuti
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Cenni di genetica di popolazione delle specie native
 Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Differenze nella trascrizione tra procarioti ed eucarioti
 Differenze nella trascrizione tra procarioti ed eucarioti Gli RNA eucariotici vanno incontro ad un processo di maturazione nel nucleo modificazioni post-trascrizionali negli eucarioti l mrna nascente (trascritto
Differenze nella trascrizione tra procarioti ed eucarioti Gli RNA eucariotici vanno incontro ad un processo di maturazione nel nucleo modificazioni post-trascrizionali negli eucarioti l mrna nascente (trascritto
Elementi ripetuti del Genoma Umano. Milioni di anni
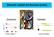 Elementi ripetuti del Genoma Umano Patologia Milioni di anni Evoluzione Un gene è duplicato Un gene è riarrangiato Genoma Il Genoma è l intero DNA contenuto in una Cellula Geni Sequenze Intergeniche Sequenze
Elementi ripetuti del Genoma Umano Patologia Milioni di anni Evoluzione Un gene è duplicato Un gene è riarrangiato Genoma Il Genoma è l intero DNA contenuto in una Cellula Geni Sequenze Intergeniche Sequenze
-85% del genoma é trascritto in RNA -Solo 1.1% codifica per proteine (c.a geni) -Le proteine sono milioni ( ogni gene più proteine) I GENI SONO
 Replication -85% del genoma é trascritto in RNA -Solo 1.1% codifica per proteine (c.a 20000 geni) -Le proteine sono milioni ( ogni gene più proteine) I GENI SONO QUINDI AMBIGUI I processi epigenetici,tutti
Replication -85% del genoma é trascritto in RNA -Solo 1.1% codifica per proteine (c.a 20000 geni) -Le proteine sono milioni ( ogni gene più proteine) I GENI SONO QUINDI AMBIGUI I processi epigenetici,tutti
Bioinformatica. Analisi del genoma
 Bioinformatica Analisi del genoma GABRIELLA TRUCCO CREMA, 5 APRILE 2017 Cosa è il genoma? Insieme delle informazioni biologiche, depositate nella sequenza di DNA, necessarie alla costruzione e mantenimento
Bioinformatica Analisi del genoma GABRIELLA TRUCCO CREMA, 5 APRILE 2017 Cosa è il genoma? Insieme delle informazioni biologiche, depositate nella sequenza di DNA, necessarie alla costruzione e mantenimento
Stesso DNA ma cellule diverse
 Stesso DNA ma cellule diverse Organismo (uomo) Il corpo umano è composto damiliardi di cellule Ogni nucleo cellulare è dotato di un identico corredo cromosomico I cromosomi sono in coppia Ogni cromosoma
Stesso DNA ma cellule diverse Organismo (uomo) Il corpo umano è composto damiliardi di cellule Ogni nucleo cellulare è dotato di un identico corredo cromosomico I cromosomi sono in coppia Ogni cromosoma
La Diagnostica molecolare non è mai stata così semplice. Idylla. Sistema di diagnostica molecolare completamente automatizzato
 La Diagnostica molecolare non è mai stata così semplice Idylla TM Sistema di diagnostica molecolare completamente automatizzato Idylla TM Passare al molecolare non è mai stato così facile Eccellente facilità
La Diagnostica molecolare non è mai stata così semplice Idylla TM Sistema di diagnostica molecolare completamente automatizzato Idylla TM Passare al molecolare non è mai stato così facile Eccellente facilità
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia. Numero di gara
 Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
ANNO 2008 I SESSIONE BIOLOGO. L eredità dei caratteri: i geni e i cromosomi L ambiente e i microrganismi Il sangue e la sua analisi
 ANNO 2008 L eredità dei caratteri: i geni e i cromosomi L ambiente e i microrganismi Il sangue e la sua analisi Igiene e laboratorio Legislazione professionale Certificazione di qualità Il candidato descriva
ANNO 2008 L eredità dei caratteri: i geni e i cromosomi L ambiente e i microrganismi Il sangue e la sua analisi Igiene e laboratorio Legislazione professionale Certificazione di qualità Il candidato descriva
Lezione 3. Genoma umano come esempio di genoma eucariote
 Lezione 3 Genoma umano come esempio di genoma eucariote Schema della lezione Sommario degli elementi contenuti in un genoma eucariote Variabilità: dove si trova e come si definisce I grandi progetti internazionali
Lezione 3 Genoma umano come esempio di genoma eucariote Schema della lezione Sommario degli elementi contenuti in un genoma eucariote Variabilità: dove si trova e come si definisce I grandi progetti internazionali
