DNA revolution. Microsatellites SNPs. Sequenziamento massivo (Next Generation Sequencing NGS)
|
|
|
- Leonardo Bernardini
- 5 anni fa
- Visualizzazioni
Transcript
1 DNA revolution Microsatellites SNPs Sequenziamento massivo (Next Generation Sequencing NGS)
2 NGS
3 NGS
4 NGS Non-Sanger-based high-throughput DNA sequencing technology Increasing the throughput Minimizing the need for the fragment-cloning method often used in Sanger sequencing Enabling the sequencing of biological codes at a very rapid pace with low cost per operation II-generation sequencing technology III-generation sequencing technology Rely on a pre-amplification step Can determine the base composition of single DNA molecules
5 Sequenziamento Chimico Dye-Terminator NGS con pre-amplificazione NGS su singola molecola
6
7
8
9
10
11 NGS NGS technologies are a combination of strategies for: Template preparation Sequencing and imaging Genome alignment Assembly methods Library preparation Major platforms for NGS: by Roche 2. Solexa by Illumina 3. SOLiD by Applied Biosystem
12 454 Genome Sequencing System (Roche) Pyrosequencing-based system (2005) reads
13 454 Genome Sequencing System (Roche) In sintesi: In pratica...
14 454 Genome Sequencing System (Roche) Emulsion PCR (empcr) Ø Variazione della PCR usata nelle tecnologie NGS per replicare le sequenze di DNA Ø Frammentazione della libreria di DNA: 1. SONICAZIONE 2. NEBULIZZAZIONE
15 454 Genome Sequencing System (Roche) Emulsion PCR (empcr) Ø Variazione della PCR usata nelle tecnologie NGS per replicare le sequenze di DNA Ø Frammentazione della libreria di DNA: 1. SONICAZIONE 2. NEBULIZZAZIONE Produzione di frammenti di DNA di dimensioni tra 300 e 800 bp Ø Ligazione di ADATTATORI alle estremità dei frammenti di DNA. Questo permetterà a sua volta il legame dei frammenti con gli adattatori alle beads magnetiche. Ø Legame dei frammenti con gli adattatori alle beads
16 454 Genome Sequencing System (Roche) Ø Denaturazione della doppia elica, amplificazione Ø Formazione di beads con popolazioni di frammenti CLONALI
17 454 Genome Sequencing System (Roche)
18 454 Genome Sequencing System (Roche)
19 454 Genome Sequencing System (Roche)
20 454 Genome Sequencing System (Roche)
21 454 Genome Sequencing System (Roche) In Pico trite tube reaction of fluorescence occurs with the addition of nucleotides
22 454 Genome Sequencing System (Roche) Principio generale alla base di tutti i sistemi di pirosequenziamento: Ø Incorporazione dei nucleotidi grazie alla DNA Polimerasi Ø Conversione del Pirofosfato (PPi) in ATP grazie alla ATP Sulfurilasi Ø Produzione del segnale luminoso grazie all ossidazione operata dalla Luciferasi di una molecola di luciferina
23 454 Genome Sequencing System (Roche) Principio generale alla base di tutti i sistemi di pirosequenziamento:
24 454 Genome Sequencing System (Roche) Possibile interferenza di segnale data dal residuo di dntp s ad ogni ciclo?? The four different nucleotides are added STEPWISE and incorporation is followed using the enzyme ATP sulfurylase and luciferase, The nucleotides are continuously degraded by nucleotidedegrading enzyme (Apyrase) allowing addition of subsequent nucleotide.
25 454 Genome Sequencing System (Roche) Principio generale alla base di tutti i sistemi di pirosequenziamento:
26 454 Genome Sequencing System (Roche) Pyrogram
27 454 Genome Sequencing System (Roche)... AGGGGTGGCTTTGGGGTTGCAGTTG... Pyrogram Sequenze ottenuta Ordine di aggiunta dei nucleotidi riportato sotto il pirogramma Proportional signals are obtained for one, two, three and four base incorporations.
28 454 Genome Sequencing System (Roche) SEE VIDEO watch?v=nffgwgfe0aa
29 Solexa (2007) Sequencing on-a-chip In sintesi: Ø Breaking up DNA Ø Adding ADAPTORS, but in this case attach not to a bead but to a slide Ø FOLD-BACK PCR is then used to amplify the fragmented DNA into a cluster
30 Solexa In pratica: 1) Frammentazione del DNA e ligazione dei frammenti ottenuti a degli adattatori 2) ANNEALING degli adattatori a singolo filamento alla superficie sulla quale avverrà la reazione di sequenziamento
31 Solexa 3-5) Amplificazione tramite BRIDGE-PCR
32 Solexa 6) Formazione di cluster densi e separati. Ogni cluster contiene una singlola sequenza 7) Aggiunta di nucleotidi fluorescenti grazie ad una polimerasi
33 Solexa 8) Lettura della fluorescenza del primo nucleotide 9) Utilizzo di terminatori reversibili (il fluoroforo può essere clivato)
34 Solexa Lunghezza dei frammenti raggiunta: 100 bp (in PAIR-END)
35 Solexa SEE VIDEO
36 SOLiD (2008) Ø Developed by George M. Church at Harvard University Ø Developed by Applied Biosystems, released in 2008 Ø Very cost effective ($0.13/million bases) Ø Has trouble with palindromic sequencing No DNA polimerasi Utilizzo dell enzima LIGASI (based on the oligonucleotide ligation tecnique)
37 SOLiD (2008) Ø Reads di 150 bp massimo (generalmente 35 bp) Ø Massima accuratezza Ø Vengono utilizzate sonde con due basi (two-based encoding system) Ø Ogni base della sequenza viene letta 2 volte
38 SOLiD (2008) In Sintesi: 1. LIBRARY PREPARATION: Pre-amplificazione con empcr su microsfere Ligazione delle microsfere su un vetrino 2. LIGATION AND IMAGING Ligation of the probes Different cycles
39 SOLiD (2008) 1) LIBRARY PREPARATION 1a)
40 SOLiD (2008) 1) LIBRARY PREPARATION 1b)
41 SOLiD (2008) 1) LIBRARY PREPARATION 1c) EmPCR/Bead Enrichment
42 SOLiD (2008) 1) LIBRARY PREPARATION 1c) EmPCR/Bead Enrichment empcr Millions of copies of template strand on each bead
43 SOLiD (2008) 1) LIBRARY PREPARATION
44 SOLiD (2008) 1) LIBRARY PREPARATION 1c) EmPCR/Bead Enrichment
45 SOLiD (2008) 1) LIBRARY PREPARATION 1c) EmPCR/Bead Enrichment
46 SOLiD (2008) 1) LIBRARY PREPARATION 1d) Deposit 3 modified beads onto a glass slide
47 SOLiD (2008) 2) LIGATION AND IMAGING Reagents: Template Strands (from PCR) Primers (to start reaction) 8 nucleotide long probes with dye Ligase to append probes to primers
48 SOLiD (2008) 2) LIGATION AND IMAGING Ligation Chemistry Process (Overview): 1. Primer binds to template strands 2. Probe hybridization and ligation 3. Fluorescent measured 4. Dye-end nucleotides cleaved 5. Steps 1-4 repeated 6+ times The entire process is completed n times. Each time the primer is offset by 1 base
49 SOLiD (2008) 2) LIGATION AND IMAGING
50 SOLiD (2008) 2) LIGATION AND IMAGING Ligazione del primer all adattatore Ligazione della prima sonda per compatibilità delle prime due basi
51 SOLiD (2008) 2) LIGATION AND IMAGING Lettura della fluorescenza Taglio della parte terminale della sonda
52 SOLiD (2008) 2) LIGATION AND IMAGING
53 SOLiD (2008) 2) LIGATION AND IMAGING
54 SOLiD (2008)
55 SOLiD (2008) Sonda arancio Adapter *
56 SOLiD (2008) Sonda rossa Adapter *
57 SOLiD (2008) SEE VIDEO
58 SOLiD (2008) Issues with palindromic sequences
59 SOLiD (2008) Issues with palindromic sequences
60 SOLiD and Solexa Generated much larger number of reads than the 454 system (30 and 100 million reads, respectively) But they were too short (only 35 bp long in the SOLiD system)
61 Ion Semiconductor Ion Torrent (2011) Does not rely on fluorescence Maximum read length of up to 400 bp
62 Ion Semiconductor Ion Torrent
63 Ion Semiconductor Ion Torrent
64 Application of NGS
65 Advantages of NGS Ø Do not require bacterial cloning of DNA fragments Ø Rely on the preparation of NGS libraries in a cell-free system Ø They can parallelize the thousands-to-many-millions of sequencing reactions Ø The sequencing output is directly detected with no need for electhroforesis Disvantages of NGS Ø Short read lengths which has resulted in difficulties in subsequent sequence splicing, assembly, annotation and bioinformatic analysis Ø Efficiency of the starter PCR can be affected by complex structures of the genome and the complete genomic sequencing cannot be represented in the library
66
67
68
69
70
71
72 SEE VIDEO watch?v=3uhw22hbpak
73 NGS NGS technologies are a combination of strategies for: Template preparation Sequencing and imaging Genome alignment Assembly methods
74 NGS NGS technologies are a combination of strategies for: Template preparation Sequencing and imaging Genome alignment Assembly methods
75 NGS
76 NGS
Applicazioni biotecnologiche in systems biology
 Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
Applicazioni biotecnologiche in systems biology Lezione #2 Dr. Marco Galardini AA 2012/2013 Contatti Dr. Marco Galardini Dip. Di Biologia Via Madonna del Piano 6, Polo Scientifico S. Fiorentino (c/o Incubatore
GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 9. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS)
 Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS) Per il sequenziamento di genomi la metodica principalmente utilizzata fino a pochi anni fa era basata su una strategia enzimatica, metodo
Dr. Tommaso Giordani METODI DI NEXT GENERATION SEQUENCING (NGS) Per il sequenziamento di genomi la metodica principalmente utilizzata fino a pochi anni fa era basata su una strategia enzimatica, metodo
NEXT- GENERATION DNA SEQUENCING
 NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
NEXT- GENERATION DNA SEQUENCING Introduzione Fino a pochi anni fa i metodi utilizzati per il sequenziamento si basavano sul metodo di Sanger, il quale però risulta avere notevoli svantaggi soprattutto
Polymerase chain reaction (PCR) qualche ora
 Lezione 3 Metodologie di sequenziamemento di DNA ed RNA. Da Sanger sequencing ad High Throughput (HTS) o di Next Generation (NGS) o di seconda generation Polymerase chain reaction (PCR) qualche ora 25
Lezione 3 Metodologie di sequenziamemento di DNA ed RNA. Da Sanger sequencing ad High Throughput (HTS) o di Next Generation (NGS) o di seconda generation Polymerase chain reaction (PCR) qualche ora 25
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health. NGS: tutti i segreti del laboratorio
 Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health NGS: tutti i segreti del laboratorio Francesco Cerutti S.S. Genetica e Immunobiochimica IstitutoZooprofilatticoSperimentaledel
Applicazioni del Sequenziamento di Nuova Generazione (NGS) nel contesto One Health NGS: tutti i segreti del laboratorio Francesco Cerutti S.S. Genetica e Immunobiochimica IstitutoZooprofilatticoSperimentaledel
Next Generation Sequencers: from the bacterial culture to raw data. Valeria Michelacci NGS course, June 2015
 Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
Next Generation Sequencers: from the bacterial culture to raw data Valeria Michelacci NGS course, June 2015 COSTS ASSOCIATED WITH DNA SEQUENCING 80-100 $ per Bacterial Genome!! Benefits from NGS Massive
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
HI-TECH IN SANITA'. MINI-INVASIVITA'.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale NGS: IMMUNOGENETICA E TRAPIANTI Michela Mazzocco S.C.Laboratorio di istocompatibilità
Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei
 ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
ANALISI DELLA SEQUENZA NUCLEOTIDICA DEL DNA Uno dei metodi più comunemente usati per questa analisi è quello di Sanger, detto anche metodo della terminazione della catena o dei didesossiribonucleotidi
Il DNA antico (adna)
 DNA antico Il DNA antico (adna) «Con Il termine DNA antico (ancient DNA = adna) si indica qualsiasi traccia di DNA proveniente da un organismo morto o da parte di esso, o anche DNA estratto da campioni
DNA antico Il DNA antico (adna) «Con Il termine DNA antico (ancient DNA = adna) si indica qualsiasi traccia di DNA proveniente da un organismo morto o da parte di esso, o anche DNA estratto da campioni
DNA Profiling e Genetica Forense. Silvia Fuselli Lezione 1
 DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
DNA Profiling e Genetica Forense Silvia Fuselli fss@unife.it Lezione 1 I geni negli individui Omozigote: lo stesso allele ad un locus in organismi diploidi (o poliplodi) E O E Eterozigote: Alleli diversi
Lezione 5. Next Generation Sequencing
 Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Lezione 5 Next Generation Sequencing Perchè Next Generation Sequencing Si possono generare centinaia di milioni di corte sequenze (35bp-250bp) in una sola corsa in un tempo breve con un basso prezzo per
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
20 anni di progressi nella conoscenza sul rischio neoplastico eredo-familiare
 20 anni di progressi nella conoscenza sul rischio neoplastico eredo-familiare 3.7% MO 2/12/2016 20 anni di progressi nella conoscenza sul rischio neoplastico eredo-familiare 7.4% MO 2/12/2016 Frederick
20 anni di progressi nella conoscenza sul rischio neoplastico eredo-familiare 3.7% MO 2/12/2016 20 anni di progressi nella conoscenza sul rischio neoplastico eredo-familiare 7.4% MO 2/12/2016 Frederick
Mutation screening mediante PCR
 Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
Mutation screening mediante PCR Denaturing High-Perfomance Liquid Chromatography (DHPLC) E una tecnica di analisi cromatografica ad alta pressione che consente di discriminare tra homoduplex ed heteroduplex
PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay)
 Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
Tecniche per l analisi di singole variazioni: PCR allele-specifica Ibridazione con sonde allele-specifiche (ASO) PCR Real-time con sonde TaqMan Saggio OLA (Oligo Ligation Assay) alcuni SNP (ca. 10%) sono
UNIVERSITA' DEGLI STUDI DI FOGGIA DOTTORATO DI RICERCA IN: INNOVAZIONE E MANAGEMENT DEGLI ALIMENTI AD ELEVATO IMPATTO SALUTISTICO -CICLO XXIX-
 UNIVERSITA' DEGLI STUDI DI FOGGIA DOTTORATO DI RICERCA IN: INNOVAZIONE E MANAGEMENT DEGLI ALIMENTI AD ELEVATO IMPATTO SALUTISTICO -CICLO XXIX- POSSIBILE INTERAZIONE TRA I LIVELLI PLASMATICI DI OMOCISTEINA
UNIVERSITA' DEGLI STUDI DI FOGGIA DOTTORATO DI RICERCA IN: INNOVAZIONE E MANAGEMENT DEGLI ALIMENTI AD ELEVATO IMPATTO SALUTISTICO -CICLO XXIX- POSSIBILE INTERAZIONE TRA I LIVELLI PLASMATICI DI OMOCISTEINA
UNIVERSITA DEGLI STUDI DI CAGLIARI Dipartimento di Scienze Mediche UNIVERSITA DEGLI STUDI DI CAGLIARI. Dipartimento di Scienze Mediche Genetica Medica
 AIBT Winter School Ravascletto (UD) 3-5 Dicembre 2015 NGS: NGS: vantaggi, vantaggi, ambiti ambiti e e ricadute. ricadute. Sandro Orrù Sandro Orrù UNIVERSITA DEGLI STUDI DI CAGLIARI UNIVERSITA DEGLI STUDI
AIBT Winter School Ravascletto (UD) 3-5 Dicembre 2015 NGS: NGS: vantaggi, vantaggi, ambiti ambiti e e ricadute. ricadute. Sandro Orrù Sandro Orrù UNIVERSITA DEGLI STUDI DI CAGLIARI UNIVERSITA DEGLI STUDI
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Tecniche di biologia molecolare. Utilizzi delle endonucleasi e polimerasi
 Tecniche di biologia molecolare Utilizzi delle endonucleasi e polimerasi 4 PRINCIPALI TIPI DI ENZIMI AGISCONO SUL DNA: - NUCLEASI (idrolisi del legame fosfodiestere in oligonucleotidi) - LIGASI (formazione
Tecniche di biologia molecolare Utilizzi delle endonucleasi e polimerasi 4 PRINCIPALI TIPI DI ENZIMI AGISCONO SUL DNA: - NUCLEASI (idrolisi del legame fosfodiestere in oligonucleotidi) - LIGASI (formazione
GENETICS AND MOLECULAR BIOLOGY FOR ENVIRONMENTAL ANALYSIS MOLECULAR ECOLOGY LESSON 6: DNA SEQUENCING. Prof. Alberto Pallavicini
 GENETICS AND MOLECULAR BIOLOGY FOR ENVIRONMENTAL ANALYSIS MOLECULAR ECOLOGY LESSON 6: DNA SEQUENCING Prof. Alberto Pallavicini pallavic@units.it GENOMICS ANALYSIS Genomics is highly technology driven.
GENETICS AND MOLECULAR BIOLOGY FOR ENVIRONMENTAL ANALYSIS MOLECULAR ECOLOGY LESSON 6: DNA SEQUENCING Prof. Alberto Pallavicini pallavic@units.it GENOMICS ANALYSIS Genomics is highly technology driven.
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
Whole genome SNPs comparison: SNPtree, NDtree, CSI Phylogeny and kmer-based analysis
 Whole genome SNPs comparison: SNPtree, NDtree, CSI Phylogeny and kmer-based analysis Valeria Michelacci NGS course, June 2016 Webserver-based free pipelines available NCBI pipeline FDA GenomeTrakr CGE/DTU
Whole genome SNPs comparison: SNPtree, NDtree, CSI Phylogeny and kmer-based analysis Valeria Michelacci NGS course, June 2016 Webserver-based free pipelines available NCBI pipeline FDA GenomeTrakr CGE/DTU
kind of data (Next Generation Sequencing and other available evidences), and to produce a well-defined genome assembly. Many efficient assembler have
 Abstract Next Generation Sequencing (NGS) technologies have a great impact both at economical and at research level, with the increasing of data production and the cost reduction and. This new kind of
Abstract Next Generation Sequencing (NGS) technologies have a great impact both at economical and at research level, with the increasing of data production and the cost reduction and. This new kind of
Tecniche di sequenziamento del DNA
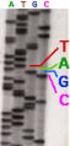 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Lezione Novembre 2009
 Lezione 9-10 10 Novembre 2009 corso di genomica a.a. 2009/10 aula 6a ore 14.00-16.00 corso di laurea specialistica magistrale Biotecnologia lezione 11 Dicembre sequenziamento shot-gun metodo pyrofosfato
Lezione 9-10 10 Novembre 2009 corso di genomica a.a. 2009/10 aula 6a ore 14.00-16.00 corso di laurea specialistica magistrale Biotecnologia lezione 11 Dicembre sequenziamento shot-gun metodo pyrofosfato
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Single-rate three-color marker (srtcm)
 3. Markers Pag. 1 The Single Rate Three Color Marker (srtcm) can be used as component in a Diffserv traffic conditioner The srtcm meters a traffic stream and marks its packets according to three traffic
3. Markers Pag. 1 The Single Rate Three Color Marker (srtcm) can be used as component in a Diffserv traffic conditioner The srtcm meters a traffic stream and marks its packets according to three traffic
Ai miei genitori, Love is caring enough not to hold on tightly. Con affetto, Fabrizio
 Ai miei genitori, Love is caring enough not to hold on tightly Con affetto, Fabrizio UNIVERSITA DI PISA Corso di Laurea Magistrale in Medicina Veterinaria DNA-FISH in cromosomi metafasici di Bos taurus
Ai miei genitori, Love is caring enough not to hold on tightly Con affetto, Fabrizio UNIVERSITA DI PISA Corso di Laurea Magistrale in Medicina Veterinaria DNA-FISH in cromosomi metafasici di Bos taurus
PCR. (Reazione a Catena della Polimerasi) Kary Mullis
 POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
POLYMERASE CHAIN REACTION - PCR PCR Polymerase Chain Reaction (Reazione a Catena della Polimerasi) Kary Mullis Premio Nobel per la Chimica nel 1993, Kary Mullis è divenuto una leggenda per la scoperta
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Summer School 2015. Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi
 Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
Summer School 2015 Alloreattività e trapianti nell uomo: le nuove metodiche di studio e i trapianti alternativi Applicazioni della Next-Generation Sequencing con tecnologia Illumina Roberto Cusano 04-06
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
Prof.ssa Tiziana Pepe Tecniche molecolari Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Tecniche molecolari
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
6.5 RNA Secondary Structure. 18 novembre 2014
 6.5 RNA Secondary Structure 18 novembre 2014 Calendario Oggi è la lezione 17/24: ultima lezione su Programmazione dinamica Metodo greedy: 18, 19 Grafi: 20, 21, 22, 23 Reti di flusso: 23, 24 (=mercoledì
6.5 RNA Secondary Structure 18 novembre 2014 Calendario Oggi è la lezione 17/24: ultima lezione su Programmazione dinamica Metodo greedy: 18, 19 Grafi: 20, 21, 22, 23 Reti di flusso: 23, 24 (=mercoledì
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Accesso Mul*plo - modelli
 Accesso Mul*plo - modelli Conceptual Model of Mul/ple Access A B C D Station A Station B Station C Station D Master Channel The Master does not know if and how many packets are present in each queue (i.e.,
Accesso Mul*plo - modelli Conceptual Model of Mul/ple Access A B C D Station A Station B Station C Station D Master Channel The Master does not know if and how many packets are present in each queue (i.e.,
Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari
 Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari Roberto Bresciani Dipartimento di Medicina Molecolare e Traslazionale Università degli Studi di Brescia Biologia cellulare Biologia
Tecnichedi laboratorioper lo studio di modelli cellulari e molecolari Roberto Bresciani Dipartimento di Medicina Molecolare e Traslazionale Università degli Studi di Brescia Biologia cellulare Biologia
6.5 RNA Secondary Structure
 6.5 RNA Secondary Structure Struttura di una biomolecola Biomolecola: DNA, RNA Struttura primaria: descrizione esatta della sua composizione atomica e dei legami presenti fra gli atomi Struttura secondaria:
6.5 RNA Secondary Structure Struttura di una biomolecola Biomolecola: DNA, RNA Struttura primaria: descrizione esatta della sua composizione atomica e dei legami presenti fra gli atomi Struttura secondaria:
lezione giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Lezione IX-X martedì 25-X-2011
 Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Genomica Servizio Sequenziamento DNA
 Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
BIOCHIMICA MOLECOLARE CLINICA
 Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
Corso di Laurea Magistrale in BIOLOGIA CELLULARE e MOLECOLARE E SCIENZE BIOMEDICHE A.A. 2016 2017 BIOCHIMICA MOLECOLARE CLINICA Principi e tecniche molecolari per la diagnosi di patologie umane Prof. Patrizia
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Tecniche biofisiche basate su nanopori
 Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Tecniche biofisiche basate su nanopori -Pori di dimensione nanometrica -Attraverso membrane lipidiche o inorganiche -Di natura proteica o scavati con tecnologie top-down Possono essere impiegati per -Studiare
Strumenti della Genetica Molecolare Umana (5)
 Strumenti della Genetica Molecolare Umana (5) Prof. Mario Ventura Capitoli 6-7-8 LEZIONE SUMMARY PER TECNICHE GIORNO 23 OTTOBRE LEZIONE AGGIUNTIVA GIORNO 30 NOVEMBRE ORE 8:30-11:00 Prof. Mario Ventura
Strumenti della Genetica Molecolare Umana (5) Prof. Mario Ventura Capitoli 6-7-8 LEZIONE SUMMARY PER TECNICHE GIORNO 23 OTTOBRE LEZIONE AGGIUNTIVA GIORNO 30 NOVEMBRE ORE 8:30-11:00 Prof. Mario Ventura
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
eaction A very efficient method for IN VITRO REPLICATION of DNA
 1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
1 olymerase hain What is it? eaction A very efficient method for IN VITRO REPLICATION of DNA What is for? Synthesis of several million copies of a specific DNA sequence within a few hours 2 PCR INGREDIENTS
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Quantitative real time Polymerase Chain Reaction
 Quantitative real time Polymerase Chain Reaction sonde TaqMan attività 5 3 esonucleasica della DNA polimerasi Un filamento di DNA polimerizzato = 1 emissione di fluorescenza Permette di vedere cosa succede
Quantitative real time Polymerase Chain Reaction sonde TaqMan attività 5 3 esonucleasica della DNA polimerasi Un filamento di DNA polimerizzato = 1 emissione di fluorescenza Permette di vedere cosa succede
La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria
 La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria Il 15% del DNA associato ai centromeri, costituito da sequenze altamente ripetute (DNA satellite e
La quantità di DNA umano (circa 3 miliardi di paia di basi) è in eccesso rispetto a quella necessaria Il 15% del DNA associato ai centromeri, costituito da sequenze altamente ripetute (DNA satellite e
Materiale del corso: http://bioinformatica.uniroma2.it/gc2014/
 Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Informazioni sul corso Fabrizio Ferrè fabrizioferre@uniroma2it Tel: 06 72594320 Dipartimento di Biologia, stanza 320 (dente di Genetica) Materiale del corso: http://bioinformaticauniroma2it/gc2014/ Username:
Diagnostica molecolare dei disordini genomici
 Diagnostica molecolare dei disordini genomici MLPA (Multiplex Ligation-dependent Probe Amplification) Riconoscimento di variazioni del numero di copie in più di 40 distinte sequenze genomiche mediante
Diagnostica molecolare dei disordini genomici MLPA (Multiplex Ligation-dependent Probe Amplification) Riconoscimento di variazioni del numero di copie in più di 40 distinte sequenze genomiche mediante
Quanto DNA è contenuto in una cellula?...
 Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
APPLIED GENOMICS DNA SEQUENCING
 APPLIED GENOMICS DNA SEQUENCING Prof. Alberto Pallavicini pallavic@units.it GENOMICS ANALYSIS Genomics is highly technology driven. The enormous impact of genomics research on medical and agrotechnological
APPLIED GENOMICS DNA SEQUENCING Prof. Alberto Pallavicini pallavic@units.it GENOMICS ANALYSIS Genomics is highly technology driven. The enormous impact of genomics research on medical and agrotechnological
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Principali classi di mutazioni
 Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
IBRIDAZIONE DNA:DNA. Tassonomia molecolare CHEMOTAXONOMY
 IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
IBRIDAZIONE DNA:DNA SOUTHERN blotting Hybridization Techniques Hybridization Techniques NOTHERN blotting FISH (Fluorescence In Situ Hybridization) FISH (Fluorescence In Situ Hybridization) The sample is
LA REAZIONE A CATENA DELLA POLIMERASI
 Polymerase Chain Reaction LA REAZIONE A CATENA DELLA POLIMERASI e LE SUE APPLICAZIONI Prima diagnosi molecolare sul DNA 1978 *Primo ormone umano ricombinante (insulina) *Premio Nobel agli scopritori degli
Polymerase Chain Reaction LA REAZIONE A CATENA DELLA POLIMERASI e LE SUE APPLICAZIONI Prima diagnosi molecolare sul DNA 1978 *Primo ormone umano ricombinante (insulina) *Premio Nobel agli scopritori degli
sottobasi per valvole a spola
 sottobasi per valvole a spola manifolds for spool valves Sottobasi modulari per valvole a spola /8 e /4 Multiple sub-bases for /8 and /4 spool valves Sottobasi a posti fissi per valvole a spola /8 e /4
sottobasi per valvole a spola manifolds for spool valves Sottobasi modulari per valvole a spola /8 e /4 Multiple sub-bases for /8 and /4 spool valves Sottobasi a posti fissi per valvole a spola /8 e /4
Scuola Sec. Secondo Grado FINALE IND. - 14/15 ESERCIZIO 1
 ESERCIZIO 1 Si ricordi che una regola di deduzione è un termine che ha la struttura: regola(,,). Tale termine indica una regola di nome che consente di dedurre
ESERCIZIO 1 Si ricordi che una regola di deduzione è un termine che ha la struttura: regola(,,). Tale termine indica una regola di nome che consente di dedurre
ALMA MATER STUDIORUM UNIVERSITA DI BOLOGNA
 ALMA MATER STUDIORUM UNIVERSITA DI BOLOGNA SCUOLA DI AGRARIA E MEDICINA VETERINARIA Corso di laurea magistrale in Scienze e Tecnologie Alimentari STUDIO DEL PROFILO MICROBICO E DI GENI FUNZIONALI IN CARCASSE
ALMA MATER STUDIORUM UNIVERSITA DI BOLOGNA SCUOLA DI AGRARIA E MEDICINA VETERINARIA Corso di laurea magistrale in Scienze e Tecnologie Alimentari STUDIO DEL PROFILO MICROBICO E DI GENI FUNZIONALI IN CARCASSE
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL ELETTROFORESI SU GEL Metodo per separare le molecole (DNA, RNA, proteine, etc.) sulla base di proprieta fisiche o chimiche quali: (1)dimensioni (2)forma (3)carica elettrica L'AGAROSIO
ELETTROFORESI SU GEL ELETTROFORESI SU GEL Metodo per separare le molecole (DNA, RNA, proteine, etc.) sulla base di proprieta fisiche o chimiche quali: (1)dimensioni (2)forma (3)carica elettrica L'AGAROSIO
REGISTRATION GUIDE TO RESHELL SOFTWARE
 REGISTRATION GUIDE TO RESHELL SOFTWARE INDEX: 1. GENERAL INFORMATION 2. REGISTRATION GUIDE 1. GENERAL INFORMATION This guide contains the correct procedure for entering the software page http://software.roenest.com/
REGISTRATION GUIDE TO RESHELL SOFTWARE INDEX: 1. GENERAL INFORMATION 2. REGISTRATION GUIDE 1. GENERAL INFORMATION This guide contains the correct procedure for entering the software page http://software.roenest.com/
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Laboratorio di Elementi di Bioinformatica
 Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2017/2018 ati in Bioinformatica ocente: Raffaella Rizzi 1 Outline ü Cos è un NA genomico e un RNA? Outline
Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E3101Q116) AA 2017/2018 ati in Bioinformatica ocente: Raffaella Rizzi 1 Outline ü Cos è un NA genomico e un RNA? Outline
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Lezione 8. DNA sequencing informatics
 Lezione 8 DNA sequencing informatics Il materiale di questa lezione è contenuto nel libro Next-generation DNA sequencing informatics Edited by Stuart M Brown Disponibile in biblioteca (CHIOSTRO 572.8633
Lezione 8 DNA sequencing informatics Il materiale di questa lezione è contenuto nel libro Next-generation DNA sequencing informatics Edited by Stuart M Brown Disponibile in biblioteca (CHIOSTRO 572.8633
EVOLUZIONE MOLECOLARE. Silvia Fuselli
 EVOLUZIONE MOLECOLARE Silvia Fuselli silvia.fuselli@unife.it TESTI Organizzazione del corso Graur and Li, Fundamentals of molecular evolution, Sinauer 2000 Michael Lynch, The Origins of Genome Architecture,
EVOLUZIONE MOLECOLARE Silvia Fuselli silvia.fuselli@unife.it TESTI Organizzazione del corso Graur and Li, Fundamentals of molecular evolution, Sinauer 2000 Michael Lynch, The Origins of Genome Architecture,
PCR - Polymerase Chain Reaction
 PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA,
Laboratorio di Elementi di Bioinformatica
 Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E30Q6) AA 205/206 Esempio di workflow Docente del laboratorio: Raffaella Rizzi Scopo del workflow Scopo: dato un insieme
Laboratorio di Elementi di Bioinformatica Laurea Triennale in Informatica (codice: E30Q6) AA 205/206 Esempio di workflow Docente del laboratorio: Raffaella Rizzi Scopo del workflow Scopo: dato un insieme
AIM OF THE LESSON: for the students to familiarise themselves with the language of cooking
 Lesson 1 Gli Gnocchi Date N of students AIM OF THE LESSON: for the students to familiarise themselves with the language of cooking The following activities are based on "Communicative method" which encourages
Lesson 1 Gli Gnocchi Date N of students AIM OF THE LESSON: for the students to familiarise themselves with the language of cooking The following activities are based on "Communicative method" which encourages
CORSO GENOPOM PRO. Marcatori molecolari. Docente: Pasquale Termolino
 CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI
 Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
Electronic Supplementary Information for: protein KLHL40 changes upon nemaline myopathy. associated mutation
 Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 2016 Electronic Supplementary Information for: The dynamics of the β-propeller domain in Kelch protein
Electronic Supplementary Material (ESI) for RSC Advances. This journal is The Royal Society of Chemistry 2016 Electronic Supplementary Information for: The dynamics of the β-propeller domain in Kelch protein
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI
 Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
Read Online and Download Ebook LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO TESTAMENTO VERSIONE RIVEDUTA BY GIOVANNI LUZZI DOWNLOAD EBOOK : LA SACRA BIBBIA: OSSIA L'ANTICO E IL NUOVO Click link bellow and
PCR. Polymerase Chain Reaction
 PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
Vieste (FG) maggio 2016 Centro Congressi Pugnochiuso
 Vincenzo Romano Spica Unità di Sanità Pubblica Laboratorio di Epidemiologia e Biotecnologie Università di Roma Foro Italico vincenzo.romanospica@uniroma4.it Vieste (FG) 27-28 maggio 2016 Centro Congressi
Vincenzo Romano Spica Unità di Sanità Pubblica Laboratorio di Epidemiologia e Biotecnologie Università di Roma Foro Italico vincenzo.romanospica@uniroma4.it Vieste (FG) 27-28 maggio 2016 Centro Congressi
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Acidi Nucleici: DNA = acido deossiribonucleico
 Acidi Nucleici: DNA = acido deossiribonucleico depositario dell informazione genetica RNA: acido ribonucleico trascrizione e traduzione dell informazione genetica dogma centrale della biologia molecolare
Acidi Nucleici: DNA = acido deossiribonucleico depositario dell informazione genetica RNA: acido ribonucleico trascrizione e traduzione dell informazione genetica dogma centrale della biologia molecolare
CAPITOLATO TECNICO. Kit di controllo qualità delle sfere ioniche template-positive prearricchite e post-arricchite mediante il fluorimetro Qubit (in
 REAGENTI 1 Kit di preparazione PCR in emulsione per sequenziamento Kit per la preparazione, mediante reazione di PCR in emulsione, di 120 test 240 test 163,1 39.144,00 77302685FD sfere ioniche template-positive
REAGENTI 1 Kit di preparazione PCR in emulsione per sequenziamento Kit per la preparazione, mediante reazione di PCR in emulsione, di 120 test 240 test 163,1 39.144,00 77302685FD sfere ioniche template-positive
PCR. Polymerase Chain Reaction
 PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
PCR Polymerase Chain Reaction PCR o Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) o Introdotta da Kary B. Mullis alla metà degli anni 80 ha rivoluzionato la genetica
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Decode NGS data: search for genetic features
 Decode NGS data: search for genetic features Valeria Michelacci NGS course, June 2015 Blast searches What we are used to: online querying NCBI database for the presence of a sequence of interest ONE SEQUENCE
Decode NGS data: search for genetic features Valeria Michelacci NGS course, June 2015 Blast searches What we are used to: online querying NCBI database for the presence of a sequence of interest ONE SEQUENCE
Constant Propagation. A More Complex Semilattice A Nondistributive Framework
 Constant Propagation A More Complex Semilattice A Nondistributive Framework 1 The Point Instead of doing constant folding by RD s, we can maintain information about what constant, if any, a variable has
Constant Propagation A More Complex Semilattice A Nondistributive Framework 1 The Point Instead of doing constant folding by RD s, we can maintain information about what constant, if any, a variable has
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
MODALITA DI REPLICAZIONE
 MODALITA DI REPLICAZIONE In teoria è ipotizzabile che il DNA possa duplicarsi con modalità: 1) semi-conservativa se alla generazione successiva passano due doppie eliche entrambi costituite da un'elica
MODALITA DI REPLICAZIONE In teoria è ipotizzabile che il DNA possa duplicarsi con modalità: 1) semi-conservativa se alla generazione successiva passano due doppie eliche entrambi costituite da un'elica
