Il sequenziamento del genoma umano
|
|
|
- Lino Colella
- 10 anni fa
- Visualizzazioni
Transcript
1 Il sequenziamento del genoma umano 1. storia e risultati dei due progetti: HGP e Celera. 2. innovazioni e problematiche di ricerca connesse al progetto genoma umano: (a) sequenziamento su larga scala (b) polimorfismi vs errori di sequenziamento (c) DNA ripetuto (d) Genome assembly 3. Mappe genetiche 4. Mappe fisiche: (a)mappe di restrizione (b) mappe FISH (c) le mappe di STS e l integrazione delle mappe genetiche con quelle fisiche 5. Il sequenziamento di singoli genomi: Venter e Watson
2 Cronologia 1985 progettazione UCSC (University of California Santa Cruz) progetto pubblico 1988 approvazione 1990 inizio finanziamento per 15 anni 1998 parte progetto Celera Genomics progetto privato 2001 primo annuncio completamento 2003 completamento (tasso di errore 1/10,000)
3 La Rivoluzione dello Human Genome Project (HGP) Assenza di ipotesi a priori Multidisciplinarità Accesso illimitato ai dati ELSI Ethical Legal and Social Implications
4 Obbiettivi dello HGP Produzione di mappe fisiche e mappe genetiche Sequenza completa del genoma Identificazione e localizzazione dei geni Identificazione e localizzazione dei polimorfismi
5 Risultati first draft of the human genome in February 2001 sequence of the entire genome's three billion base pairs some 90 percent complete. number of human genes significantly fewer than previous estimates (50, ,000) Full sequence completed and published in April 2003
6 Gli obbiettivi sono stati raggiunti e superati
7 The announcement June 26, 2000
8 Celera (Craig Venter) vs. HGP (Francis Collins) 2001 Venter CJ et al. The sequence of the human genome. Science Feb 16;291(5507): Lander E et al. Initial sequencing and analysis of the human genome. Nature Feb 15;409(6822):
9 polemiche Myers EW, Sutton GG, Smith HO, Adams MD, Venter JC. On the sequencing and assembly of the human genome.proc Natl Acad Sci U S A Apr;99(7): Waterston RH, Lander ES, Sulston JE.On the sequencing of the human genome.proc Natl Acad Sci U S A Mar 19;99(6):
10 Sito web
11 Il sequenziamento del genoma umano 2. innovazioni e problematiche di ricerca connesse al progetto genoma umano: (a) sequenziamento su larga scala (b) polimorfismi vs errori di sequenziamento (c) Genome assembly (d) DNA ripetuto
12 2. innovazioni e problematiche a. È possibile sequenziare al max 600 bp consecutive mentre il cromosoma più piccolo (cromosoma 21) è lungo 46,944,323 bp b. Errori di sequenziamento o polimorfismi? c. sequence assembly d. Tratti di sequenze ripetute e. DNA non clonabile
13 a. sequenziamento su larga scala (a) Top-down (hierarchical) Un singolo cromosoma viene spezzettato in larghi pezzi (cloni) che vengono clonati in vettori artificiali.i cloni vengono ordinati e ciascuno viene ulteriormente suddiviso in altri cloni ordinati. Quando i cloni raggiungono dimensioni appropiate vengono sequenziati Ogni clone è fingerprinted (pattern di restrizione o STSs) Il risultato è una mappa fisica di cloni ordinati e delle loro rispettive sequenze. La sequenza finale è ottenuta allineando le sequenze dei singoli cloni
14 a. sequenziamento su larga scala (b) Bottom-up (shotgun) Il DNA viene spezzettato in modo random in numerosi frammenti di dimesioni sequenziabili. Le sequenze si chiamano reads Questa procedura viene ripetuta più volte per ottenere reads parzialmente sovrapposte La sequenza finale viene ottenuta assemblando al calcolatore le diverse reads nessuna conoscenza a priori, nessuna mappa richiesta
15 Green ED. Strategies for the systematic sequencing of complex genomes.nat Rev Genet Aug;2(8): Review.
16 2. innovazioni e problematiche a. È possibile sequenziare al max 600 bp consecutive mentre il cromosoma più piccolo (cromosoma 21) è lungo 46,944,323 bp b. Errori di sequenziamento o polimorfismi? c. sequence assembly d. Tratti di sequenze ripetute e. DNA non clonabile
17 b. errori o polimorfismi? Polimorfismi: due sequenze differiscono in media di 1 base ogni 100 Errori: nella versione finale della sequenza del genoma umano è presente 1 errore ogni 10,000 basi
18 Read Coverage Average number of times a base is sequenced C Length of genomic segment: L Number of reads: n Length of each read: l C = n l L Lander-Waterman model: Assuming uniform distribution of reads, C=10 results in 1 gapped region per 1,000,000 nucleotides Lander ES, Waterman MS Genomic mapping by fingerprinting random clones: a mathematical analysis, Genomics 2(3): (1988)
19 2. innovazioni e problematiche a. È possibile sequenziare al max 600 bp consecutive mentre il cromosoma più piccolo (cromosoma 21) è lungo 46,944,323 bp b. Errori di sequenziamento o polimorfismi? c. sequence assembly d. Tratti di sequenze ripetute e. DNA non clonabile
20 Step in assembling the genome Given a set of (short) fragments from sequencing... find overlap between all pairs find the order of reads in DNA determine a consensus sequence
21 Contig continuous set of overlapping sequences Gap Contigs
22 Overlap-Layout-Consensus 1. Overlap: find potentially overlapping reads 2. Layout: merge reads into contigs and contigs into supercontings 3. Consensus: derive the DNA sequence and correct read errors..acgattacaataggtt.. Assemblers: ARACHNE, PHRAP, CAP, TIGR, CELERA
23 Filling in gaps Gap Contig Contig Contig Gap Probe libraries
24 Fragment Assembly Assembly was Computational Challenging Until late 1990s the shotgun fragment assembly of human genome was viewed as intractable problem
25 2. innovazioni e problematiche a. È possibile sequenziare al max 600 bp consecutive mentre il cromosoma più piccolo (cromosoma 21) è lungo 46,944,323 bp b. Errori di sequenziamento o polimorfismi? c. sequence assembly d. Tratti di sequenze ripetute e. DNA non clonabile
26 d. sequenze ripetute
27 Repeat Types Low-Complexity DNA: (e.g. ATATATATACATA ) Microsatellite repeats: (a 1 a k ) N where k ~ 3-6 (e.g. CAGCAGCAGCAG) Transposons/retrotransposons SINE (Short Interspersed Nuclear Elements): e.g., Alu: ~300 bp long, 10 6 copies Long Interspersed Nuclear Elements ~500-5,000 bp long, 200,000 copies LTR retroposons: Long Terminal Repeats (~700 bp) at each end Gene Families: genes duplicate & then diverge Segmental duplications: ~very long, very similar copies
28 Challenges in Fragment Assembly > 50% of human genome are repeats Repeat Repeat Repeat La sequenza verde-rosso-azzurro può essere assemblata in due modi diversi Repeat Repeat Repeat oppure Repeat Repeat Repeat
29 Paired-end reads help to resolve repeat order Raw sequence obtained from both ends of a cloned insert in any vector BAC Repeat Repeat Repeat
30 Il sequenziamento del genoma umano 3. Mappe genetiche 4. Mappe fisiche: (a)mappe di restrizione (b) mappe FISH (c) le mappe di STS e l integrazione delle mappe genetiche con quelle fisiche
31 Genetic marker segment of DNA with an identifiable physical location on a chromosome and whose inheritance can be followed. can be a gene, or it can be some section of DNA with no known function. when is in linkage with a gene whose location has not yet been identified, markers could be used as indirect ways of tracking the inheritance pattern of that gene
32 Genetic marker genetic markers properties: locus-specific polymorphic in the studied population easily genotyped The quality of a genetic marker is typically measured by its: Heterozygosity in the population of interest PIC (Botstein et al., 1980): Polymorphism Information Content (probability that the parent is heterozygous x probability that the offspring is informative)
33 mappe posizione di geni e/o marcatori nel genoma genetiche: ottenute mediante incrocio o analisi dei pedigrees basate sulle frequenze di ricombinazione fisiche: ottenute con tecniche di biologia molecolare. basate sulle distanze nucleotidiche
34 Mappe genetiche le prime costruite in Drosophila all inizio del secolo geni usati come marcatori (marcatori utilizzabili devono avere almeno 2 forme alleliche) utilizzati prima fenotipi morfologici, poi biochimici (nell uomo AB0, proteine del siero, antigeni HLA, malattie)
35 Morgan (Sturtevant, 1930) il crossing-over è un evento casuale, che avviene con (circa) uguale probabilità in ogni punto del cromosoma più due punti del cromosoma sono vicini, meno è frequente il crossing-over tra loro, ovvero più sono in linkage la frequenza di ricombinazione può esser presa a misura della distanza tra due geni combinando le distanze tra diverse coppie di geni, si può costruire una mappa genetica della loro posizione relativa sul cromosoma
36 In pratica: mappe di linkage Sistemi: - organismi modello incroci pianificati - uomo pedigrees Marcatori: - Geni, RFLPs, SSLPs, SNPs difficili da realizzare : tempi lunghi per gli incroci pianificati, pedigree non sempre sufficientemente grandi, fenotipi difficili da misurare oggi integrate nella costruzione delle mappe fisiche
37 mappe fisiche mappe di restrizione: indicano i siti di taglio degli enzimi di restrizione rapide e dettagliate, ma inapplicabili a genomi grandi FISH: la posizione dei marcatori è determinata ibridando sonde fluorescenti sui cromosomi interi poco accurata, ma lenta e tecnicamente difficile mappe STS: la posizione dei marcatori è determinata grazie a tecniche di PCR e/o di analisi di ibridazione di frammenti genomici
38 mappe di restrizione singole digestioni del DNA, unite a doppie digestioni e a digestioni parziali, permettono la ricostruzione della posizione relativa di tutti i siti di restrizione Lunghezza sequenza palindromica Probabilità di trovare il sito di restrizione 4 (1/4) 4 = 1 ogni 256 bp 5 (1/4) 5 = 1 ogni 1024 bp 6 (1/4) 6 = 1 ogni 4096 bp 8 (1/4) 8 = 1 ogni bp n (1/4) n
39 mappe di restrizione
40 Il risultato è mappa con le posizioni dei siti di restrizione
41 mappe FISH FISH = Fluorescent In Situ Hybridization la posizione dei marcatori è determinata ibridando sonde fluorescenti sui cromosomi interi (metafasici o anche meno condensati)
42 Mappe (FISH) Il principio delle mappe FISH è l ibridazione con sonde fuorescenti
43 stringenza La parametro più importante dell ibridazione è la stringenza. È regolata da temperatura e forza ionica
44 Stringenza alta stringenza richiede elevata omologia tra sonda e bersaglio elevata Temperatura bassa concenrtrazione salina presenza di denaturanti chimici bassa stringenza è sufficiente bassa omologia tra sonda e bersaglio bassa Temperatura elevata [ ] salina assenza di denaturanti chimici Problema con sequenze ripetute
45 Mappe STS: la mappa fisica del genoma umano secondo HGP a. Creazione di librerie PAC e BAC b. Mappaggio dei cloni c. fingerprinting dei cloni mediante STS d. Clone selection e. Clone sequencing f. Contig assembly
46 YAC: yeast artificial chromosome Vettori di clonaggio che permettono l inserimento di DNA >200 Kb inizialmente basati su mini crosmosomi di lievito (YAC) instabili Sito di clonaggio Telomeri N S Marcatore di inserimento Marcatori fenotipici centromero standard YACs fino a 600 kb special YACs fino a 1400 kb
47 Altri vettori batteriofago P1: può contenere fino a 125 kb BACs: basati sul plasmide F di E.coli; fino a > 300 kb PACs: commistione tra P1 e BAC; fino a > 300 kb Fosmidi: contengono ori di F e cos di ; simili a cosmidi ma + stabili
48 Libraries YAC library
49 Library hybridization YAC library Marked probes hybridization * *
50 STSs: Sequence Tagged Sites sequenze brevi ( bp) Facilmente amplificabile per PCR Unica nel genoma Localizzazione genomica nota Identifica inequivocabilmente la molecola di DNA più grande che lo contiene
51 STS-like ESTs (Expressed Sequence Tags): corte sequenze derivanti dall analisi di cdna SSLPs (Simple Sequence Lenght Polymorphisms) Microsatelliti (STRs, Short Tandem Repeats) Minisatelliti (VNTRs, Variable Number Tandem Repeats)
52 STS mapping STS vicini STS lontani cromosoma collezione di frammenti 4 volte su 6 nello stesso frammento 2 volte su 6 nello stesso frammento La frequenza con cui sono sullo stesso frammento è propozionale alla distanza
53
54 (a) clone libraries amplification Cromosome from donors pool of large number of anonymous males and females fragmentation cloning BAC
55 (b) clone maps Determinazione della posizione dei cloni rispetto al cromosoma sulla base dei (pochi) STSs esistenti exsisting maps of STS clone libraries
56 (c) fingerprinting Identificazione di nuovi STSs che permetteranno di collegare tra loro i cloni exsisting maps of STS new STSs Clone_1 Clone_13 Clone_2
57 (d) Clone selection minimal tiling path collezione di cloni che permette di coprire la regione con il minor grado di sovrapposizione Clone_1 Clone_6 Clone_17 Clone_4 Clone_13 Clone_16 Clone_10
58 (e) Clone sequencing Clone_1 Clone_4 Clone_6 Clone_10 Clone_13 Clone_16 Clone_17 Shotgun secuencing Contig_1 Contig_4 Contig assembly
59 (f) Contig assembly Contig_1 Contig_4 Contig_6 Contig_10 Contig_17 Contig_13 Contig_16
60 Il sequenziamento del genoma umano 5. Il sequenziamento di singoli genomi: Venter e Watson
61
62
63
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
Sequenziamento e analisi di genomi completi
 Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Sequenziamento e analisi di genomi completi Genoma L'insieme del materiale genetico di un organismo o cellula. (Hans Winkler, 1920) Un genoma è sequenziato quando viene stabilita interamente la successione
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Biologia Molecolare. CDLM in CTF 2010-2011 L analisi del genoma
 Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Biologia Molecolare CDLM in CTF 2010-2011 L analisi del genoma L analisi del genoma n La tipizzazione del DNA n La genomica e la bioinformatica n La genomica funzionale La tipizzazione del DNA DNA Fingerprinting
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Avanzamento dei sistemi di sequenziamento
 Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
Avanzamento dei sistemi di sequenziamento Sistemi di sequenziamento capillare basati su: Lunghezza delle read: 800 basi Poche sequenze prodotte in una singola corsa Second Generation Sequencing (SGS):
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20
 SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
Definizione di genoteca (o library) di DNA
 Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
Definizione di genoteca (o library) di DNA Collezione completa di frammenti di DNA, inseriti singolarmente in un vettore di clonaggio. Possono essere di DNA genomico o di cdna. Libreria genomica: collezione
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Genomica Servizio Sequenziamento DNA
 Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Genomica Servizio Sequenziamento DNA Listino prezzi 1 maggio 2005 Value Read Codice Descrizione Prezzo / Lettura 1001-000000 Tubi 13,50 1001-000010 Tubi con etichetta codice a barre 12,00 1094-000050 Etichette
Organizzazione del genoma umano II
 Organizzazione del genoma umano II Lezione 7 & Pseudogeni I Pseudogeni non processati : convenzionali ed espressi * Copie non funzionali del DNA genomico di un gene. Contengono esoni, introni e spesso
Organizzazione del genoma umano II Lezione 7 & Pseudogeni I Pseudogeni non processati : convenzionali ed espressi * Copie non funzionali del DNA genomico di un gene. Contengono esoni, introni e spesso
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Il genoma umano. Cosa significa genoma? Ditelo con parole vostre
 Il genoma umano Cosa significa genoma? Ditelo con parole vostre Cos è il genoma umano? 22 coppie di autosomi + una coppia di cromosomi sessuali (XX o XY) http://www.molecularstation.com/molecular-biology-images/data/502/human-genome-gene.png
Il genoma umano Cosa significa genoma? Ditelo con parole vostre Cos è il genoma umano? 22 coppie di autosomi + una coppia di cromosomi sessuali (XX o XY) http://www.molecularstation.com/molecular-biology-images/data/502/human-genome-gene.png
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
I marcatori genetici e loro applicazioni nelle produzioni animali. Dott.ssa Chiara Targhetta
 I marcatori genetici e loro applicazioni nelle produzioni animali Dott.ssa Chiara Targhetta LOCUS localizzazione genomica unica all interno di un cromosoma; permette di definire la posizione di un gene
I marcatori genetici e loro applicazioni nelle produzioni animali Dott.ssa Chiara Targhetta LOCUS localizzazione genomica unica all interno di un cromosoma; permette di definire la posizione di un gene
Lezione IX-X martedì 25-X-2011
 Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
Lezione IX-X martedì 25-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre
ANALISI DI LINKAGE (CONCATENAZIONE)
 ANALISI DI LINKAGE (CONCATENAZIONE) L analisi di linkage permette di determinare la posizione cromosomica di un locus responsabile di una determinata malattia/carattere genetico rispetto a marcatori polimorfici
ANALISI DI LINKAGE (CONCATENAZIONE) L analisi di linkage permette di determinare la posizione cromosomica di un locus responsabile di una determinata malattia/carattere genetico rispetto a marcatori polimorfici
Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010. Percorso nº 3: Clonaggio di segmenti di DNA
 Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010 Percorso nº 3: Clonaggio di segmenti di DNA 11-5-2010 I plasmidi: un mondo da esplorare. Elementi genetici capaci di replicarsi autonomamente
Corso di Laurea in Biotecnologie Anno-Accademico 2009-2010 Percorso nº 3: Clonaggio di segmenti di DNA 11-5-2010 I plasmidi: un mondo da esplorare. Elementi genetici capaci di replicarsi autonomamente
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
Come studiare un genoma complesso. 1. Costruirne la mappa
 Come studiare un genoma complesso 1. Costruirne la mappa Costruzione della mappa di un cromosoma Obiettivo: ordinare in modo reciproco lungo il cromosoma geni responsabili di fenotipi riconoscibili, o
Come studiare un genoma complesso 1. Costruirne la mappa Costruzione della mappa di un cromosoma Obiettivo: ordinare in modo reciproco lungo il cromosoma geni responsabili di fenotipi riconoscibili, o
Linkage. Lezione 4 (riprendere il testo di Genetica ) By NA
 Linkage Lezione (riprendere il testo di Genetica ) Tipi di mappe: mappe genetiche Mappe genetiche : si basano sulla frequenza di ricombinazione fra locus identificati attraverso marcatori di varia natura:
Linkage Lezione (riprendere il testo di Genetica ) Tipi di mappe: mappe genetiche Mappe genetiche : si basano sulla frequenza di ricombinazione fra locus identificati attraverso marcatori di varia natura:
a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA
 CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA Dott.ssa Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due
CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA Dott.ssa Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
CORSO INTEGRATO DI GENETICA
 CORSO INTEGRATO DI GENETICA a.a.2011-2012 11.10.2011 Lezioni N. 7 e 8 Ereditarietà Mendeliana Segregazione alleli, indipendenza geni, associazione, ricombinazione Dott.ssa Elisabetta Trabetti UN GENE =
CORSO INTEGRATO DI GENETICA a.a.2011-2012 11.10.2011 Lezioni N. 7 e 8 Ereditarietà Mendeliana Segregazione alleli, indipendenza geni, associazione, ricombinazione Dott.ssa Elisabetta Trabetti UN GENE =
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI a) sequenziamento gerarchico (clone by clone) b) sequenziamento shotgun
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI a) sequenziamento gerarchico (clone by clone) b) sequenziamento shotgun
GENOMA. c varia da pochi kb nei virus a milioni di kb in piante e animali
 GENOMA Insieme del materiale genetico presente in una cellula (DNA nucleare, plastidiale e mitocondriale) Contiene tutte le informazioni necessarie per consentire la vita alla cellula e all individuo Nei
GENOMA Insieme del materiale genetico presente in una cellula (DNA nucleare, plastidiale e mitocondriale) Contiene tutte le informazioni necessarie per consentire la vita alla cellula e all individuo Nei
Analisi di dati di sequenziamento del trascrittoma (RNA-Seq):
 Il vostro progetto Analisi di dati di sequenziamento del trascrittoma (RNA-Seq): 1. Analisi di qualità 2. Mappatura sul genoma 3. Calcolo dell espressione 4. Test di espressione differenziale 5. Visualizzazione
Il vostro progetto Analisi di dati di sequenziamento del trascrittoma (RNA-Seq): 1. Analisi di qualità 2. Mappatura sul genoma 3. Calcolo dell espressione 4. Test di espressione differenziale 5. Visualizzazione
Plasmidi come vettori di clonaggio
 Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Plasmidi come vettori di clonaggio Un vettore plasmidico di buona qualità deve possedere le seguenti proprietà: 1. Piccole dimensioni (
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Come identificare i Cromosomi
 Come identificare i Cromosomi Fino al 1970, i cromosomi sono stati classificati in base alle dimensioni ed alla posizione del centromero. A I più grandi (metacentrici) B Grandi (submetacentrici) C Medi
Come identificare i Cromosomi Fino al 1970, i cromosomi sono stati classificati in base alle dimensioni ed alla posizione del centromero. A I più grandi (metacentrici) B Grandi (submetacentrici) C Medi
Nel 1997 un gruppo di ricercatori dell Università di Monaco, guidato. Genomi e genomica. DOMANDE CHIAVE Come si ottengono
 Genomi e genomica 14 DOMANDE CHIAVE Come si ottengono le sequenze del DNA genomico? Come viene decifrata l informazione contenuta nel genoma? Che cosa può rivelare la genomica comparata sulla struttura
Genomi e genomica 14 DOMANDE CHIAVE Come si ottengono le sequenze del DNA genomico? Come viene decifrata l informazione contenuta nel genoma? Che cosa può rivelare la genomica comparata sulla struttura
Il sequenziamento del DNA
 Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
Il sequenziamento del DNA Si può ottenere la massima informazione sulla struttura di una molecola di DNA determinandone la sequenza nucleotidica completa Il sequenziamento del DNA è una componente irrinunciabile
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo Per animali transgenici
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo Per animali transgenici
Bioinformatica (modulo bioinf. dei genomi moderni )
 Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Bioinformatica (modulo bioinf. dei genomi moderni ) Dr. Marco Fondi Lezione # 5 Corso di Laurea in Scienze Biologiche, AA 2011-2012 giovedì 3 novembre 2011 1 Sequenziamento ed analisi di genomi: la genomica
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Alberto Viale I CROMOSOMI
 Alberto Viale I CROMOSOMI DA MENDEL ALLA GENETICA AL DNA ALLE MUTAZIONI I cromosomi sono dei particolari bastoncelli colorati situati nel nucleo delle cellule. Sono presenti nelle cellule di ogni organismo
Alberto Viale I CROMOSOMI DA MENDEL ALLA GENETICA AL DNA ALLE MUTAZIONI I cromosomi sono dei particolari bastoncelli colorati situati nel nucleo delle cellule. Sono presenti nelle cellule di ogni organismo
Sequenziamento del DNA: strutture dei nucleotidi
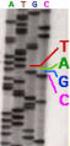 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
CORSO DI GENETICA. Roberto Piergentili. Università di Urbino Carlo Bo INGEGNERIA GENETICA
 CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
CORSO DI GENETICA INGEGNERIA GENETICA Tecniche di DNA ricombinante: gli enzimi di restrizione Le tecniche di base del DNA ricombinante fanno prevalentemente uso degli enzimi di restrizione. Gli enzimi
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY.
 Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
Carpire il segreto della vita con l informatica Giosuè Lo Bosco Dipartimento di Matematica e Informatica, Università di Palermo, ITALY. Lezioni Lincee Palermo, 26 Febbraio 2015 Alla base della vita degli
Nuovi ruoli dei telomeri e della telomerasi
 Nuovi ruoli dei telomeri e della telomerasi Marco Santagostino Tutor: Elena Giulotto Dipartimento di Genetica e Microbiologia, Università degli Studi di Pavia Argomenti trattati 1. I telomeri e la telomerasi
Nuovi ruoli dei telomeri e della telomerasi Marco Santagostino Tutor: Elena Giulotto Dipartimento di Genetica e Microbiologia, Università degli Studi di Pavia Argomenti trattati 1. I telomeri e la telomerasi
Dissezione del fenotipo
 Clinica molecolare (molecular medicine):come utilizzare le informazioni del genoma per studiare le patologie genetiche Clinica molecolare Nuova branca della medicina che nasce 2.400 anni fa con Democrito
Clinica molecolare (molecular medicine):come utilizzare le informazioni del genoma per studiare le patologie genetiche Clinica molecolare Nuova branca della medicina che nasce 2.400 anni fa con Democrito
I Genomi degli Eucarioti:
 I Genomi degli Eucarioti: Eucromatina ed Eterocromatina Eucromatina: regioni cromosomiche non condensate, attivamente trascritte e ad alta densità genica. Eterocromatina: (facoltativa o costitutiva): cromatina
I Genomi degli Eucarioti: Eucromatina ed Eterocromatina Eucromatina: regioni cromosomiche non condensate, attivamente trascritte e ad alta densità genica. Eterocromatina: (facoltativa o costitutiva): cromatina
Indice dell'opera. Prefazione. Capitolo 1 Introduzione alla genetica Genetica classica e moderna Genetisti e ricerca genetica Sommario
 Indice dell'opera Prefazione Capitolo 1 Introduzione alla genetica Genetica classica e moderna Genetisti e ricerca genetica Capitolo 2 DNA: il materiale genetico La ricerca del materiale genetico La composizione
Indice dell'opera Prefazione Capitolo 1 Introduzione alla genetica Genetica classica e moderna Genetisti e ricerca genetica Capitolo 2 DNA: il materiale genetico La ricerca del materiale genetico La composizione
Esperienza 3: clonaggio di un gene in un plasmide
 Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Esperienza 3: clonaggio di un gene in un plasmide Il clonaggio molecolare è una delle basi dell ingegneria genetica. Esso consiste nell inserire un frammento di DNA (chiamato inserto) in un vettore appropriato
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperienza 2: gli enzimi di restrizione
 Esperienza 2: gli enzimi di restrizione Gli enzimi di restrizione sono delle proteine sintetizzate dai batteri per proteggersi dalle infezioni virali (batteriofagi). Questi enzimi tagliano il DNA virale
Esperienza 2: gli enzimi di restrizione Gli enzimi di restrizione sono delle proteine sintetizzate dai batteri per proteggersi dalle infezioni virali (batteriofagi). Questi enzimi tagliano il DNA virale
Biotecnologie ed OGM. Prima parte: DNA ricombinante e microorganismi geneticamente modificati.
 Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
Biotecnologie ed OGM Prima parte: DNA ricombinante e microorganismi geneticamente modificati. COSA SONO LE BIOTECNOLOGIE? Si dicono Biotecnologie i metodi tecnici che permettono lo sfruttamento di sistemi
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Ingegneria Genetica e Microbiologia Applicata
 Corso di Laurea in Biotecnologie Anno Accademico 2009-2010 Ingegneria Genetica e Microbiologia Applicata Percorso n 3: Clonaggio di segmenti di DNA Settima esercitazione - 13 maggio 2010 F 1 1 1: taglio
Corso di Laurea in Biotecnologie Anno Accademico 2009-2010 Ingegneria Genetica e Microbiologia Applicata Percorso n 3: Clonaggio di segmenti di DNA Settima esercitazione - 13 maggio 2010 F 1 1 1: taglio
Esercitazioni di Genomica
 Esercitazioni di Genomica Bioinformatica ai tempi del NGS, PhD CRIBI Biotechnology Center, University of Padua BMR Genomics srl, Spin-Off Giovanni Birolo, PhD CRIBI Biotechnology Center, University of
Esercitazioni di Genomica Bioinformatica ai tempi del NGS, PhD CRIBI Biotechnology Center, University of Padua BMR Genomics srl, Spin-Off Giovanni Birolo, PhD CRIBI Biotechnology Center, University of
GENOMA. Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine CONTENUTO FUNZIONE. Progetti genoma in centinaia di organismi
 GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Genetica. Mendel e la genetica
 Genetica Le leggi dell ereditarietà di Mendel Ereditarietà e cromosomi Estensioni della genetica mendeliana Applicazioni della genetica Genoma umano Mendel e la genetica Mendel 81822-1884), un monaco di
Genetica Le leggi dell ereditarietà di Mendel Ereditarietà e cromosomi Estensioni della genetica mendeliana Applicazioni della genetica Genoma umano Mendel e la genetica Mendel 81822-1884), un monaco di
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
Agrobacterium è in grado di trasferire nel genoma vegetale qualsiasi sequenza si trovi delimitata dai right e left borders
 Agrobacterium è in grado di trasferire nel genoma vegetale qualsiasi sequenza si trovi delimitata dai right e left borders gene di interesse gene di interesse mcs multi cloning site Il gene di interesse
Agrobacterium è in grado di trasferire nel genoma vegetale qualsiasi sequenza si trovi delimitata dai right e left borders gene di interesse gene di interesse mcs multi cloning site Il gene di interesse
GENETICA... lessico. Genetica: studio dei geni e dell'ereditarietà
 GENETICA... lessico Genetica: studio dei geni e dell'ereditarietà Geni: porzioni di DNA contenenti un'informazione che permette di decodificare una certa proteina. Es: gene che determina il colore dei
GENETICA... lessico Genetica: studio dei geni e dell'ereditarietà Geni: porzioni di DNA contenenti un'informazione che permette di decodificare una certa proteina. Es: gene che determina il colore dei
TEORIA CROMOSOMICA : ALLEGATI
 TEORIA CROMOSOMICA : ALLEGATI FIG. 2 a pag. 1 FIG. 5 a pag. 3 FIG. 7 a pag. 5 FIG. 9 a pag. 7 FIG. 3 e 4 a pag. 2 FIG. 6 a pag. 4 FIG. 8 a pag. 6 FIG. 10 e 11 a pag. 8 1 FIGURA 2 Perché sono tutti maschi
TEORIA CROMOSOMICA : ALLEGATI FIG. 2 a pag. 1 FIG. 5 a pag. 3 FIG. 7 a pag. 5 FIG. 9 a pag. 7 FIG. 3 e 4 a pag. 2 FIG. 6 a pag. 4 FIG. 8 a pag. 6 FIG. 10 e 11 a pag. 8 1 FIGURA 2 Perché sono tutti maschi
Progetto Genoma Umano
 Progetto Genoma Umano Perché il Progetto Genoma? La determinazione e la conoscenza dell intera sequenza genomica sembrano essere la condizione necessaria per comprendere la completa biologia di un determinato
Progetto Genoma Umano Perché il Progetto Genoma? La determinazione e la conoscenza dell intera sequenza genomica sembrano essere la condizione necessaria per comprendere la completa biologia di un determinato
Il genoma dinamico: gli elementi trasponibili
 Il genoma dinamico: gli elementi trasponibili Anni trenta: studi sul mais ribaltano la visione classica secondo cui i geni si trovano solo in loci fissi sul cromosoma principale Esistono elementi genetici
Il genoma dinamico: gli elementi trasponibili Anni trenta: studi sul mais ribaltano la visione classica secondo cui i geni si trovano solo in loci fissi sul cromosoma principale Esistono elementi genetici
Introduzione al corso di bioinformatica e analisi dei genomi AA 2014-2015. Docente: Silvia Fuselli [email protected]
 Introduzione al corso di bioinformatica e analisi dei genomi AA 2014-2015 Docente: Silvia Fuselli [email protected] Fonti e testi di riferimento Dan Graur: http://nsmn1.uh.edu/dgraur/ >courses > bioinformatics
Introduzione al corso di bioinformatica e analisi dei genomi AA 2014-2015 Docente: Silvia Fuselli [email protected] Fonti e testi di riferimento Dan Graur: http://nsmn1.uh.edu/dgraur/ >courses > bioinformatics
Insegnamento di Genomica. Corsi di Laurea Specialistica in: Biotecnologie Agro-industriali Biologia Molecolare e cellulare
 Insegnamento di Genomica Corsi di Laurea Specialistica in: Biotecnologie Agro-industriali Biologia Molecolare e cellulare docenti: Silvia Fuselli ([email protected]) Vincenza Colonna Inga Prokopenko Morena
Insegnamento di Genomica Corsi di Laurea Specialistica in: Biotecnologie Agro-industriali Biologia Molecolare e cellulare docenti: Silvia Fuselli ([email protected]) Vincenza Colonna Inga Prokopenko Morena
PLASMIDI puc ALTRI TIPI DI VETTORI. VETTORI λ 17-06-2010
 PLASMIDI puc ALTRI TIPI DI VETTORI VETTORI λ (15-20 Kb) = vettori ottenuti apportando delle modifiche al genoma del batteriofago λ. COSMIDI (40-45 Kb) = plasmidi che contengono i siti cos di λ utili per
PLASMIDI puc ALTRI TIPI DI VETTORI VETTORI λ (15-20 Kb) = vettori ottenuti apportando delle modifiche al genoma del batteriofago λ. COSMIDI (40-45 Kb) = plasmidi che contengono i siti cos di λ utili per
IL MIGLIORAMENTO GENETICO ANIMALE E LA GENETICA MOLECOLARE
 IL MIGLIORAMENTO GENETICO ANIMALE E LA GENETICA MOLECOLARE Il miglioramento genetico delle produzioni animali, realizzato sino ad ora, è frutto dell elaborazione elaborazione e applicazioni della teoria
IL MIGLIORAMENTO GENETICO ANIMALE E LA GENETICA MOLECOLARE Il miglioramento genetico delle produzioni animali, realizzato sino ad ora, è frutto dell elaborazione elaborazione e applicazioni della teoria
Tappe della GENETICA UMANA. G. Mendel leggi dell ereditarietà 1865
 Tappe della GENETICA UMANA G. Mendel leggi dell ereditarietà 1865 Tappe della GENETICA UMANA G. Mendel leggi dell ereditarietà 1865 Watson & Crick DNA 1953 Tappe della GENETICA UMANA G. Mendel leggi dell
Tappe della GENETICA UMANA G. Mendel leggi dell ereditarietà 1865 Tappe della GENETICA UMANA G. Mendel leggi dell ereditarietà 1865 Watson & Crick DNA 1953 Tappe della GENETICA UMANA G. Mendel leggi dell
Sequenziamento ed analisi dell esoma intero (All Exon)
 Sequenziamento ed analisi dell esoma intero (All Exon) Obiettivi La procedura ha l obiettivo di sequenziare solo le regioni trascritte e codificanti del genoma che rappresentano, almeno nell uomo, circa
Sequenziamento ed analisi dell esoma intero (All Exon) Obiettivi La procedura ha l obiettivo di sequenziare solo le regioni trascritte e codificanti del genoma che rappresentano, almeno nell uomo, circa
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
Vettori plasmidici pbr322 puc18
 Plasmidi 0-10 kb Fago lambda (di inserzione) 0-15 kb Fago lambda (di sostituzione) 9-23 Kb Cosmidi 30-45 Kb Batteriofago P1 70-100 Kb PAC 130-150 kb BAC 70-200 kb YAC 200-2000 kb MAC ~ 1000 kb 1 Vettori
Plasmidi 0-10 kb Fago lambda (di inserzione) 0-15 kb Fago lambda (di sostituzione) 9-23 Kb Cosmidi 30-45 Kb Batteriofago P1 70-100 Kb PAC 130-150 kb BAC 70-200 kb YAC 200-2000 kb MAC ~ 1000 kb 1 Vettori
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
Elementi di genetica di base e marcatori molecolari. Dott.ssa Marica Soattin
 Elementi di genetica di base e marcatori molecolari Dott.ssa Marica Soattin Organismi animali costituiti da cellule di tipo eucariote Cellule con nucleo differenziato delimitato da membrana e organelli
Elementi di genetica di base e marcatori molecolari Dott.ssa Marica Soattin Organismi animali costituiti da cellule di tipo eucariote Cellule con nucleo differenziato delimitato da membrana e organelli
Come ordinare Geni sintetici Online
 Come ordinare Geni sintetici Online I geni sintetici di Eurofins MWG Operon possono essere ordinati online grazie all esclusivo software GENEius. Di seguito una guida dettagliata per effettuare la richiesta
Come ordinare Geni sintetici Online I geni sintetici di Eurofins MWG Operon possono essere ordinati online grazie all esclusivo software GENEius. Di seguito una guida dettagliata per effettuare la richiesta
MANIPOLAZIONE GENETICA DEGLI ANIMALI
 MANIPOLAZIONE GENETICA DEGLI ANIMALI Perché creare animali transgenici Per studiare la funzione e la regolazione di geni coinvolti in processi biologici complessi come lo sviluppo di un organismo e l insorgenza
MANIPOLAZIONE GENETICA DEGLI ANIMALI Perché creare animali transgenici Per studiare la funzione e la regolazione di geni coinvolti in processi biologici complessi come lo sviluppo di un organismo e l insorgenza
Analisi dei dati MLPA con il nuovo Coffalyser.NET. MRC-Holland
 Analisi dei dati MLPA con il nuovo Coffalyser.NET MRC-Holland Contenuti Che cos è il Coffalyser.NET Analisi dei frammenti e del Copy number Interpretazione dei dati Che cos è il Cofffalyser.NET Software
Analisi dei dati MLPA con il nuovo Coffalyser.NET MRC-Holland Contenuti Che cos è il Coffalyser.NET Analisi dei frammenti e del Copy number Interpretazione dei dati Che cos è il Cofffalyser.NET Software
Estendere Lean e Operational Excellence a tutta la Supply Chain
 Estendere Lean e Operational Excellence a tutta la Supply Chain Prof. Alberto Portioli Staudacher www.lean-excellence.it Dipartimento Ing. Gestionale Politecnico di Milano [email protected] Lean
Estendere Lean e Operational Excellence a tutta la Supply Chain Prof. Alberto Portioli Staudacher www.lean-excellence.it Dipartimento Ing. Gestionale Politecnico di Milano [email protected] Lean
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Corso di Biologia Molecolare
 Corso di Biologia Molecolare Dott.ssa Renata Tisi Dip. Biotecnologie e Bioscienze Ed. U4 Tel. 02 6448 3522 [email protected] Acidi nucleici Il ruolo degli acidi nucleici è quello di custodire e trasmettere
Corso di Biologia Molecolare Dott.ssa Renata Tisi Dip. Biotecnologie e Bioscienze Ed. U4 Tel. 02 6448 3522 [email protected] Acidi nucleici Il ruolo degli acidi nucleici è quello di custodire e trasmettere
Caratterizzazione dei geni coinvolti nelle nuove traslocazioni riguardanti la banda 14q32
 2 Workshop SIES di Ematologia traslazionale Verona, 21-22 Maggio 2009 Caratterizzazione dei geni coinvolti nelle nuove traslocazioni riguardanti la banda 14q32 Identificazione di una correlazione tra l'espressione
2 Workshop SIES di Ematologia traslazionale Verona, 21-22 Maggio 2009 Caratterizzazione dei geni coinvolti nelle nuove traslocazioni riguardanti la banda 14q32 Identificazione di una correlazione tra l'espressione
"Sotto l'azzurro fitto del cielo qualche uccello di mare se ne va, nè sosta mai perchè tutte le immagini portano scritto più in là".
 Lentamente muore chi diventa schiavo dell'abitudine, ripetendo ogni giorno gli stessi percorsi, chi non cambia la marca, chi non rischia e cambia colore dei vestiti, chi non parla a chi non conosce. Muore
Lentamente muore chi diventa schiavo dell'abitudine, ripetendo ogni giorno gli stessi percorsi, chi non cambia la marca, chi non rischia e cambia colore dei vestiti, chi non parla a chi non conosce. Muore
ESEMPI DI QUERY SQL. Esempi di Query SQL Michele Batocchi AS 2012/2013 Pagina 1 di 7
 ESEMPI DI QUERY SQL Dati di esempio... 2 Query su una sola tabella... 2 Esempio 1 (Ordinamento)... 2 Esempio 2 (Scelta di alcune colonne)... 3 Esempio 3 (Condizioni sui dati)... 3 Esempio 4 (Condizioni
ESEMPI DI QUERY SQL Dati di esempio... 2 Query su una sola tabella... 2 Esempio 1 (Ordinamento)... 2 Esempio 2 (Scelta di alcune colonne)... 3 Esempio 3 (Condizioni sui dati)... 3 Esempio 4 (Condizioni
Downloaded from www.immunologyhomepage.com. Riarrangiamento dei geni per le Immunoglobuline e sviluppo dei linfociti B
 Downloaded from www.immunologyhomepage.com Riarrangiamento dei geni per le Immunoglobuline e sviluppo dei linfociti B I geni che codificano i recettori per gli antigeni (BCR e TCR) sono presenti in uno
Downloaded from www.immunologyhomepage.com Riarrangiamento dei geni per le Immunoglobuline e sviluppo dei linfociti B I geni che codificano i recettori per gli antigeni (BCR e TCR) sono presenti in uno
Algoritmi di clustering
 Algoritmi di clustering Dato un insieme di dati sperimentali, vogliamo dividerli in clusters in modo che: I dati all interno di ciascun cluster siano simili tra loro Ciascun dato appartenga a uno e un
Algoritmi di clustering Dato un insieme di dati sperimentali, vogliamo dividerli in clusters in modo che: I dati all interno di ciascun cluster siano simili tra loro Ciascun dato appartenga a uno e un
Bioinformatica. Marin Vargas, Sergio Paul
 Bioinformatica Marin Vargas, Sergio Paul 2013 Wikipedia: La bioinformatica è una disciplina scientifica dedicata alla risoluzione di problemi biologici a livello molecolare con metodi informatici. La bioinformatica
Bioinformatica Marin Vargas, Sergio Paul 2013 Wikipedia: La bioinformatica è una disciplina scientifica dedicata alla risoluzione di problemi biologici a livello molecolare con metodi informatici. La bioinformatica
Vettori di espressione
 Vettori di espressione Vengono usati per: 1.Generare sonde di RNA 2.Produrre la proteina codificata Per fare questo viene utilizzato un promotore che risiede sul vettore, modificato per ottimizzare l interazione
Vettori di espressione Vengono usati per: 1.Generare sonde di RNA 2.Produrre la proteina codificata Per fare questo viene utilizzato un promotore che risiede sul vettore, modificato per ottimizzare l interazione
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
