Metodiche di tipizzazione molecolare di specie batteriche
|
|
|
- Raffaela Graziano
- 6 anni fa
- Visualizzazioni
Transcript
1 Metodiche di tipizzazione molecolare di specie batteriche Gherardi Giovanni Università Campus Bio-Medico Roma Corso di Laurea Tecniche di laboratorio Biomedico aa.2010/2011
2 Diversità genetica: specificità ospite Patogenicità resistenza antibiotica virulenza perché identificare i batteri a livello di sub specie/ceppo? Aumentata virulenza e trasmissibilità Resistenza multipla agli antibiotici Aumentato spettro d ospite Possibilità di manipolazione genetica con bioterrorismo Diversità intraspecie è dovuto a 3 eventi: trasferimento genico orizzontale perdita o acquisizione di geni ricombinazione
3 quindi, perché la tipizzazione molecolare : ORIGINE CLONALE DEI CEPPI (SE DERIVANO DA UN ORGANISMO PARENTALE) IDENTIFICAZIONE DEI BATTERI A LIVELLO DI CEPPO DALLA DIVERSITA GENETICA SPIEGARE IMPORTANTI CARATTERISTICHE FENOTIPICHE
4 APPLICAZIONI: - clinico, disseminazione di infezioni pazientepaziente, animale-paziente, ambiente-paziente) - ambientale (presenza e diffusione di organismi in ambienti inanimati) - industriale (identificazione di organismi di valore o minacciosi nel campo bio-industriale) Utile per identificare ceppi o cloni all interno di una specie di patogeni emergenti (bioterrorismo e in biologia forense)
5 Distinguiamo: - comparativa typing : di screening - definitiva typing (library): definisce definitivamente il clone Tipizzazione può essere eseguita a diversi livelli: 1.locale, per piccoli studi. 2.regionale o nazionale, in un reference laboratory. 3.Internazionale, networks internazionali, definendo e sorvegliando la disseminazione globale di particolari cloni.
6 PRIMA FASE: SELEZIONE DELLA COLLEZIONE DEI CEPPI Ceppi di particolare interesse: - ceppi non correlati epidemiologicamente - ceppi appartenenti ad outbreaks a - isolati con determinati criteri di selezione (particolari fattori di virulenza, particolari tratti di antibiotico-resistenza, ecc) Il numero dei ceppi e la complessità dipende dagli obiettivi della ricerca Gli organismi dovrebbero essere conservati in brodo con glicerolo a 80 C, secondo le linee guida. IMPORTANTE! Vi deve essere sempre un database con dati clinici, epidemiologici e demografici dei ceppi. a (outbreak: aumento temporale nella incidenza di infezione o colonizzazione da parte di certe specie batteriche, provocate da una aumentata trasmissione di un ceppo specifico).
7 RAGIONI PER LA TIPIZZAZIONE Sorveglianza delle malattie infettive Investigazione di outbreaks Studi di patogenesi e del corso di infezione. Quando riguarda tipizzazione di gruppi di ceppi con diverso grado di virulenza, in questo caso possono essere identificati markers di virulenza correlati alla patogenesi (presenza di determinati fattori di virulenza associati ad alcuni cloni o ceppi). Studio della genetica di popolazione batterica
8 CRITERI PER LA VALUTAZIONE E VALIDAZIONE DEI METODI DI TIPIZZAZIONE 1. Performance criteria - Stabilità - typeability - Potere discriminatorio - Concordanza (con il quadro epidemiologico) - Riproducibilità - test population 2. Convenience criteria - Flessibilità e spettro - Rapidità - Accessibilità - Facilità di esecuzione - Costo - Disponibilità per analisi computerizzata e di database elettronici
9 Performance criteria STABILITA Le caratteristiche testate dovrebbero rimanere stabili per ogni isolato dopo il suo isolamento primario e durante la conservazione e la subcultura in laboratorio. Per saggiare la stabilità, subculture multiple dello stesso ceppo, conservato in periodi diversi e in diverse condizioni, devono essere processate nella stessa corsa per rendere al minimo le variazioni introdotte in laboratorio TYPEABILITY Si riferisce alla capacità del metodo di assegnare un tipo a tutti gli isolati testati con quel metodo. Può essere espressa come percentuale di isolati tipizzabili sul numero totale di isolati tipizzati (tipizzabili e non-tipizzabili). POTERE DISCRIMINATORIO Si riferisce alla capacità di un metodo di assegnare diversi tipi a due ceppi non correlati campionati randomly all interno di una popolazione di una data specie. Può essere espresso come probabilità, mediante il Simpson index. Il Simpson index dovrebbe idealmente corrispondere a 1.00, ma in pratica dovrebbe essere > 0.95 perché un metodo di tipizzazione possa essere considerato più o meno ideale. Metodi di tipizzazione che indagano polimorfismi in siti multipli dell intero genoma hanno più probabilità di avere un maggiore potere discriminatorio rispetto ai metodi che indagano un singolo locus.
10 CONCORDANZA Concordanza epidemiologica. I risultati di tipizzazione dovrebbero riflettere, confermare e possibilmente chiarire l informazione epidemiologica disponibile sui casi in esame. RIPRODUCIBILITA Si riferisce alla capacità di un metodo di tipizzazione di assegnare lo spesso tipo ad un isolato testato in occasioni indipendenti, separate nel tempo e/o nello spazio. La riproducibilità può essere influenzata da vari step in una procedura, come risultato o del protocollo usato o del grado di stringenza: variazioni nella condizione di crescita, dei metodi usati nella estrazione del DNA, nei reagenti usati, diversi tipi di apparecchiature, diversità nella osservazione e registrazione dei risultati, come anche nela loro analisi ed interpretazione. Da ciò è importante sia la riproducibilità intra-laboratorio che quella inter-laboratorio; entrambe richiedono standardizzazione dei protocolli e personale adeguatamente attrezzato. TEST POPULATION Questo è il prerequisito per valutare la typeability, il potere discriminatorio e la concordanza epidemiologica dei metodi di tipizzazione. La natura della popolazione è definita dal contesto epidemiologico, cioè dalla specie coinvolta, se lo studio è locale, regionale o globale, e se la sorveglianza è long-term. Un sufficiente numero di isolati all interno di una popolazione test dovrebbe essere incluso (circa 100 isolati) e questi dovrebbero riflettere la diversità totale interno di quella specie. E anche importante coprire diverse nicchie ecologiche, come particolari popolazioni di pazienti (età, stato immunitario, tipo di ospedale e reparto, origine geografica, ecc) e importante serbatoi ambientali per le infezioni ad esempio foodborne e waterborne.
11 Convenience criteria FLESSIBILITA E SPETTRO Questo riflette il range di specie che sono tipizzabili con modificazioni minime del metodo (principio, apparecchiature, reagenti, ecc.). I metodi basati sul sequenziamento del DNA mostrano ottima flessibilità. RAPIDITA Si riferisce al tempo totale richiesto, a partire dagli isolati batterici ai risultati finali di tipizzazione (interpretazione dei risultati inclusa). I metodi più rapidi sono le cosiddette procedure coltureindipendenti. ACCESSIBILITA Dipende dalla disponibilità dei reagenti e delle apparecchiature, e dalle attitudini richieste dal personale. FACILITA DI ESECUZIONE Comprende semplicità tecnica, carico di lavoro, utile per processare un gran numero di isolati, facilità di interpretazione dei risultati. COSTO DISPONIBILITA PER ANALISI COMPUTERIZZATA E DI DATABASE ELETTRONICI Disponibilità di analisi computerizzata ed incorporazione dei risultati in database elettronici, fattori importanti per studi di confronti longitudinali tra numerosi isolati. Studi epidemiologici locali: analisi computerizzata e visiva. Studi epidemiologici regionali e globali, importante la creazione di database elettronici per valutare diffusione di specifici cloni. Importante esempio di database elettronici : MLST.
12 METODI DI TIPIZZAZIONE FENOTIPICI E GENOTIPICI
13 METODI FENOTIPICI - biotyping - pattern di sensibilità agli antibiotici (antibiogramma) - serotyping - tipizzazione fagica e batteriocina (saggia i profili litici esposti a diversi batteriofagi o batteriocine - tossine batteriche) - SDS-PAGE, di componenti cellulari e extracellulari - Multilocus enzyme electrophoresis (MLEE) - Spettrometria di massa (MS) diversi approcci -omics : proteomics, glycomics, MALDI - TOF
14 Biotyping Reazioni enzimatiche Fermentazioni di zuccheri Biotipo 1 Biotipo 2 Biotipo 3 Biotipo 4
15 Serotyping Antibiotyping
16 Quellung reaction per la sierotipizzazione capsulare dello Streptococcus pneumoniae (esempio antisiero per sierotipo capsulare 14)
17 Serotyping
18 Tipizzazione fagica
19 SDS-PAGE
20
21 MALDI TOF MS
22
23 Moura et al. FEMS Immunol Med Microbiol 2008, 53:
24 Moura et al. FEMS Immunol Med Microbiol 2008, 53:
25 METODI BASATI SULL IBRIDAZIONE - ibridazione diretta - ribotyping METODI BASATI SU FRAMMENTI - tipizzazione plasmidica METODI GENOTIPICI - metodi RFLP (restriction fragment length polymorphism): Restriction endonuclease analysis (REA); Ribotyping; Tipizzazione della sequenza IS6110 di M. tuberculosis PFGE - PCR fingerprinting (esempio sequenze BOX in S. pneumoniae o sequenze IS256 per S. aureus) Per aumentare la risoluzione della PCR fingerprinting la PCR è seguita da restrizione con un enzima di restrizione (PCR-RFLP analisi). - amplified fragment length polymorphism (AFLP) analisi, esempio di metodo di tipizzazione basato sulla PCR. - multilocus variable number tandem repeat (VNTR) analisi (MLVA) METODI BASATI SUL SEQUENZIAMENTO DEL DNA - single-locus sequence typing (SLST): emm typing per lo S. pyogenes, spa typing per S.aureus. - MLST - SNP genotyping
26 METODI BASATI SULL IBRIDAZIONE Direct (and reverse) hybridisation: Il DNA immobilizzato viene ibridizzato con sonde di DNA selettive; alcuni templates vengono riconosciuti, altri no. Essenzialmente è la tecnologia del Southern hybridisation. Esempi: Binary typing per lo S. aureus Spoligotyping per M.tuberculosis, amplificazione di locus con tandem repeat con variazioni nella sequenza interna. Tali varianti vengono evidenziate con sonde tandem repeat-specifiche
27 Usa sonde di DNA Ribotyping: Stima il numero dei loci genici ribosomiali e la loro localizzazione lungo il cromosoma Gli operoni rrna comprendono geni conservati, ciascuno dei quali è fiancheggiato da regioni di DNA variabili. Variazioni di sequenze in queste regioni porta a diversi profili RFLP quando si utilizzano sonde di DNA per i domini conservati dei geni 16S e 23S rrna.
28 Vantaggi: Automatizzata Riproducibile Operoni rrna sono universali poche bande prodotte economica Ribotyping: Svantaggi: Basso potere discriminatorio richiede grandi quantità di DNA genomico ad alta qualità labor e time consuming sistemi di rilevazione complessi, quali radioisotopi
29
30 METODI BASATI SUI FRAMMENTI Plasmid typing: Dipende dal numero, dalla dimensione e dai profili di restrizione con REs. Svantaggi: Mancanza di stabilità nel contenuto dei plasmidi rende tale tecnica poco utilizzata
31 REA: REstriction endonuclease analysis (REA), utilizzando enzimi ad alta frequenza di taglio. L analisi REA viene accoppiata a Southern blot e poi ad ibridazione, esempi: Rybotyping IS 6110 M.tuberculosis IS6110 typing- RFLP
32 PFGE SCELTA DELL ENZIMA DI RESTRIZIONE (RE con bassa frequenza di taglio) STANDARDIZZAZIONE DEI PROTOCOLLI, SVILUPPO DI DATABASE GLOBALI (PulseNet: per foodborne deseases quali Escherichia coli O157:H7, Salmonella non-typhi, Shigella, e Listeria monocytogenes) LIMITAZIONI PERSONALE SPECIALIZZATO (concentrazione del DNA nelle plugs, percentuale di agarosio, voltaggio, temperatura forza del tampone, attitudini tecniche, ecc) LABORIOSA E LUNGA SCARSA RIPRODUCIBILITA INTERLABORATORIO RICHIEDE DNA AD ALTA QUALITA BANDE QUASI IDENTICHE, MA DIVERSE: SCARSA RISOLUZIONE
33 PFGE
34
35
36
37
38
39
40
41 Dendogramma Dice coefficiente: no. bande A + no. bande B uguali no. bande A + no. bande B totali
42
43 Un evento mutazionale può differire da 0 a 4 bande (frammenti di DNA). Quando non vi sono bande di differenza osservabili, gli isolati devono essere chiamati indistinguishable e non identical, ed assegnati allo stesso tipo e subtipo. Quando gli isolati differiscono per 1-4 bande, questi devono essere assegnati allo stesso tipo, ma con diverso subtipo. Da 5 a 8 bande di differenza possono essere attribuibili ad almeno due eventi mutazionali. In generale: -fino a 4 bande di differenza: isolati correlati geneticamente. -da 5 a 6 bande di differenza: da considerare le informazioni cliniche ed epidemiologiche -sopra le 6 bande di differenza: isolati non correlati geneticamente Sempre: correlare i dati ottenuti con la PFGE con le notizie cliniche ed epidemiologiche. Interpretazione visiva + computer-assistita.
44 La PCR (reazione a catena della polimerasi) utilizzata per amplificare sequenze di DNA (a) Il DNA da amplificare (DNA bersaglio) viene prima riscaldato per separare le due eliche (DENATURATION), poi vengono aggiunti in eccesso due oligonucleotidi come inneschi (primers) (ANNEALING), uno complementare ad ogni elica, e la DNA polimerasi. (b) In seguito all appaiamento del primer l estensione del primer da parte della DNA polimerasi genera una copia della sequenza originale di DNA (EXTENSION). (c) Due cicli aggiuntivi di PCR generano rispettivamente 4 e 8 copie della sequenza originale di DNA. (d) Risultati ottenuti dopo 20 cicli di PCR su una preparazione di DNA contenente solo 10 copie del gene bersaglio. Utilizzo della PCR: 1.Clonaggio e sequenziamento 2.Studi comparativi ed evoluzionistici (gene del 16S rrna) 3.Amplificare piccole quantità di DNA 4.Microbiologia diagnostica 5.Fingerprinting in medicina legale
45 PCR
46 PCR fingerprinting: Utilizzo di primers conosciuti che vanno ad amplificare frammenti ben noti di specifiche specie batteriche (a differenza dei primers sconosciuti che vanno ad amplificare arbitrariamente frammenti di DNA) esempi: BOX-PCR per S. penumoniae IS256-PCR per S. aureus BOX-PCR Per aumentare la risoluzione della PCR fingerprinting la PCR è seguita da restrizione con un enzima di restrizione (PCR-RFLP analisi). Permette di evidenziare mutazioni puntiformi nel sito di taglio dell enzima: è una variante del metodo Single nucleotide polimorphislms (SNPs)
47 REP-PCR Sequenze di DNA ripetute in multiple copie sono presenti nei genomi batterici. La loro funzione non è nota, ma sono utili per il fingerprinting dei batteri. Utilizzati specifici primers che amplificano frammenti di DNA che si ritrovano tra le sequenze ripetute. 3 famiglie di sequenze ripetute sono state utilizzate nella REP-PCR: - le sequenze bp repetitive extragenic palindromic (REP) -le sequenze bp enterobacterial repetitive intergenic consensus (ERIC) -le sequenze 154-bp BOX element I frammenti si visualizzano mediante elettroforesi su gel agarosio o elettroforesi capillare. VANTAGGI Basso costo, rapidità, facilità, poco laboriosa, riproducibilità sia su eucarioti che su procarioti usata su: Acinetobacter spp., Streptococcus pneumoniae, M. tuberculosis, Clostridium difficile, L. monocytogenes Un kit commerciale: REP-PCR DiversiLab (Spectral Genomics Inc., Houston, TX), con il DIVERSILAB software che fornisce la interpretazione e la conservazione dei dati in un database. Inoltre permette di lavorare sulle cellule intere senza estrarre il DNA. SVANTAGGI rischi di contaminazioni, Artefatti, Necessità di controlli multipli
48 AP-PCR (Arbitrarily primed PCR) o RAPD E una PCR random di regioni genomiche sconosciute usando primers scelti arbitrariamente. Usando primers corti (generalmente di 10 bp) in combinazione con basse temperature di annealing, l AP-PCR permette l amplificazione di loci multipli, con ampliconi di diversa grandezza. VANTAGGI economica rapida Sensibile SVANTAGGI Riproducibilità inter-laboratorio Influenzata da diversi paramentri( temperatura di annealing e sequenza dei primers, qualità e concentrazione del DNA template, apparecchiatura di PCR e reagenti) Ottimizzazione Standardizzazione della procedura mediante Ready-To-Go RAPD analysis beads (Amersham Pharmacia Biotech), che fornisce Taq, dntps, buffers e un protocollo generale della RAPD
49 AP-PCR (Arbitrarily primed PCR) o RAPD
50 AP-PCR (Arbitrarily primed PCR) o RAPD I primers arbitrari devono legarsi al template secondo uno specifico ORIENTAMENTO I primers arbitrari devono legarsi al template entro una RAGIONEVOLE DISTANZA l uno dall altro
51 Amplified Fragment Length Polimorphism Analysis (AFLP):
52 AFLP consiste di 2 amplificazioni: una preselettiva ed una più selettiva, che genera frammenti che possono essere usati come markers discriminatori Il successo della AFLP dipende da tre fattori: reagenti ottimizzati, una robusta ed affidabile piattaforma elettroforetica, un efficacie analisi software (elettroforesi capillare). Vantaggi: possono essere amplificati più loci in una reazione di PCR, poco DNA richiesto, buon potere discriminatorio, riproducibile, economica, automatizzata Tranne che per pochissime specie bateriche (es Acinetobacter baumannii), la riproducibilità interlaboratorio è scarsa data la necessità assoluta di software dedicati per l interpretazione dei risultati specialmente utilizzando primers marcati e l utilizzo di apparecchiature di sequenziamento automatizzato)
53
54 MLVA (multilocus variable number tandem repteat analysis) Metodica introdotta nel 2000
55 MLVA Variable number tandem repeats (VNTR) sono sequenze di DNA ripetute in tandem ( testa-coda ), che variano nel numero di copie e sono ampiamente disperse nel genoma, sia in regioni non codificanti che nei geni, che evolvono rapidamente. Utilizzando primers specifici a sequenze altamente conservate si ottengono frammenti che variano per dimensione e per numero di unità ripetute, i cui profili si analizzano mediante software specifici. Marcando gli ampliconi di PCR con diversi coloranti e separandoli mediante elettroforesi capillare in un sequenziatore automatico, la MLVA può essere eseguita come multiplex PCR, aumentando l efficacia. Il primo passo è individuare i loci VNTR lungo il genoma (diversi programmi, come TANDEM REPEAT FINDER, il Tandem Repeat database, e il Microsatellite Repeats database). Tali programmi permettono anche di stabilire delle relazioni filogenetiche.
56 VANTAGGI Alto potere discriminatorio, Rapida, Facile, Economica, Riproducibile, Utilizzabile per le outbreaks SVANTAGGI VNTR evolvono rapidamente: non utilizzabile per gli studi epidemiologici long-term. In alcune specie (es E. faecium) potere discriminatorio più basso rispetto alla MLST e PFGE. Utilizzata su: Francisella tularensis, Bacillus anthracis, Yersinia pestis, e M. tuberculosis. Più recentemente su methicillin-resistant Staphylococcus aureus, Burkholderia pseudomallei, e C. difficile. La stabilità è principalmente associata alla lunghezza dell unità ripetuta, al numero di copie dell unità ripetuta, e alla purezza. Vi è un programma (SERV), che serve alla selezione e al confronto delle VNTR, che si basa proprio sulla lunghezza dell unità ripetuta, al numero di copie dell unità ripetuta, e alla purezza. Con la conoscenza delle sequenze genomiche di nuove specie batteriche sarà possibile migliorare la metodica MLVA, trovando nuove VNTRs e nuovi primers.
57
58 Metodi basati sul sequenziamento del DNA
59 Sequenziamento del DNA
60 I dideossinucleotidi e il sequenziamento con il metodo di Sanger (a) Un deossinucleotide normale ha un gruppo idrossilico sul carbonio al 3 mentre un dideossinucleotide ne è privo. (b) La terminazione della reazione di elongazione avviene a seguito all incorporazione del dideossinucleotide
61 Sequenziamento del DNA utilizzando il metodo di Sanger Si richiede un DNA a singolo filamento e la sequenza viene determinata in seguito a sintesi del filamento copia utilizzando una DNA polimerasi, che sintetizza a partire da un innesco specifico (primer), usando i 4 deossiribonucleotidi trifosfati. Nelle miscele di incubazione, costituite da 4 provette separate, vi sono piccole quantità di analoghi dideossi dei deossiribonucleotidi. Poiché lo zucchero nei dideossi manca la catena si interrompe e la reazione di sequenziamento termina. (a) Si devono correre quattro diverse reazioni, una per ogni dideossinucleotide. Si ottengono frammenti di lunghezza variabile in dipendenza dei tempi di incubazione. Analizzando i vari frammenti e confrontandoli è possibile leggere la sequenza copia del DNA. Utilizzo di dideossi radioattivi, rilevazione con sviluppo di lastre radiografiche (b) Utilizzo di dideossi fluorescenti, sequenziamento ottenuto con metodo automatico (c), bande riconosciute per spettroscopia a fluorescenza. Utilizzo di 4 diversi coloranti fluorescenti A:blu G:rosso C:verde T:giallo
62 Single-locus sequence typing (SLST) emm typing per lo Streptococcus pyogenes spa typing per lo Staphylococcus aureus
63 emm sequence typing
64 emm sequence typing Amplificazione di circa 1500 bp. From B. Beall Le sequenze della regione ipervariabile possono essere ottenute da tutti i sierotipi di Lancefield ceppi di riferimento. In generale, queste sequenze condividono soltanto circa 40 80% di identità di sequenza dei 50 codoni delle regioni determinanti il sequence type.
65 Sequence hypervariable (HV) typespecific region adjacent to primer 1 From B. Beall emm gene
66 Emm gene database From B. Beall Attualmente sono stai descritti> 180 sequence types che condividono > 95% identità di sequenza della regione determinante il tipo.
67 Shortcut per il sequence typing: D ice (O p t :1. 00 % ) ( T o l 1. 5 % % ) ( H > 0. 0 % S > 0. 0% ) [ 0.0 % % ] e m m e m m HindII + HaeIII emm1 emm12 emm3 D ic e (O p t :1. 00 % ) ( T o l 1. 5 % % ) ( H > 0. 0 % S > 0. 0 % ) [ 0.0 % % ] e m m e m m DdeI emm12 emm3 emm1 emm Ha Hi Dd Ha Dd From B. Beall
68 emm PCR-RFLP RFLP typing (hindii + haeiii restriction enzymes) emm 4 emm 87 emm 3 emm 75 st 448 emm 22 emm 14 emm 22 emm 12 emm 87 emm 9 emm 5 emm 1 emm 77 emm 80 emm 44/61 emm 87 emm 78 emm 89 emm 4 emm 5 emm 28 emm 59 emm 6 emm 11 emm 29 emm 2 emm 28 Dicuonzo G, Gherardi G et al, JCM 2001
69 spa sequence typing
70 MLST
71
72 Interpretation of MLST In the interpretation of results, strains with identical allelic profiles or differing at a single locus (single locus variant SLV) were considered to be genetically related, and strains differing in 2 of the 7 loci (double locus variant DLV) were assumed likely to be genetically related.
73 An example of population analysis by eburst
74
75
76 DNA microarrays Vetrini trattati (chips) che contengono numerose reazioni in un unica reazione.
77 Utilizzo di più metodiche di tipizzazione combinate
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92 Metodi di typing solo per la ricerca? NO!!!!!!!!!!!! anche per l attività clinica nelle misure di controllo dell infezione.
93
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
La variabilità genetica: tipi e metodi per studiarla e misurarla
 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
ANALISI MOLECOLARE DEL GENOMA
 ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in Italia
 Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in talia Progetto ricerca corrente MSRCTE0212 Patrizia Centorame Laboratorio Nazionale
Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in talia Progetto ricerca corrente MSRCTE0212 Patrizia Centorame Laboratorio Nazionale
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Nested
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Marcatori molecolari per l analisi genica, genetica e genomica
 Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
lezione giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM)
 lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
lezione 17-18 giovedì 8 aprile 2010 aula 2 ore 9:00 corso integrato di Biologia Applicata (BU) ed Ingegneria Genetica (BCM) il prodotto di PCR è sempre a lineare b solo al primo ciclo il templato è solo
La GENETICA DELLE POPOLAZIONI. studia con modelli matematici, a livello di gruppi di individui, variabilità genetica
 La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
La GENETICA DELLE POPOLAZIONI studia con modelli matematici, a livello di gruppi di individui, la variabilità genetica che è l unico tipo di variabilità rilevante per l evoluzione La variabilità genetica
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali tiziana.pepe@unina.it Metodologia
Le tecniche molecolari nell epidemiologia infettiva e nella sorveglianza delle Hospital Acquired Infections (HAI)
 Le tecniche molecolari nell epidemiologia infettiva e nella sorveglianza delle Hospital Acquired Infections (HAI) Dott.ssa G. Scalet Sezione Microbiologia-Dipartimento Di Patologia e Diagnostica Università
Le tecniche molecolari nell epidemiologia infettiva e nella sorveglianza delle Hospital Acquired Infections (HAI) Dott.ssa G. Scalet Sezione Microbiologia-Dipartimento Di Patologia e Diagnostica Università
Lo studio delle comunità microbiche. È stato reso possibile dalle Tecniche molecolari
 Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
Lo studio delle comunità microbiche È stato reso possibile dalle Tecniche molecolari Nella maggior parte dei casi le tecniche utilizzano l amplificazione di segmenti conservati Usando come «stampo» il
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
ORARIO LEZIONI del MASTER in Molecular Diagnostics A.A. 2016/2017 APRILE
 3 4 5 6 7 CHIOCCHETTI CHIOCCHETTI CHIOCCHETTI/D'ALFONSO CHIOCCHETTI CHIOCCHETTI 09.00-11.00 IL DNA il clonaggio la PCR/il sequenziamento e le banche L'RNA gel agarosio dati estrazione di DNA plasmidico
3 4 5 6 7 CHIOCCHETTI CHIOCCHETTI CHIOCCHETTI/D'ALFONSO CHIOCCHETTI CHIOCCHETTI 09.00-11.00 IL DNA il clonaggio la PCR/il sequenziamento e le banche L'RNA gel agarosio dati estrazione di DNA plasmidico
CORSO GENOPOM PRO. Marcatori molecolari. Docente: Pasquale Termolino
 CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
CORSO GENOPOM PRO Marcatori molecolari Docente: e-mail: Pasquale Termolino termolin@unina.it Ottobre 2013 Struttura del Corso! 24 ore totali! Introduzione! Marcatori fenotipici! Marcatori biochimici! Tecniche
GENOMA. Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine CONTENUTO FUNZIONE. Progetti genoma in centinaia di organismi
 GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
Cenni di genetica di popolazione delle specie native
 Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
Southern blot. !Per. determinare la presenza o assenza di specifici frammenti genici. !Studio. della struttura e dell organizzazione di un gene
 Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza. O.G.M. Vegetali
 Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
Progetto :TRIFOGLIO UNIVERSITA CATTOLICA DEL SACRO CUORE Piacenza O.G.M. Vegetali Identificazione di un O.G.M. e metodi di analisi del DNA transgenico Marco Nani 5BL Liceo Scientifico Mattei di Fiorenzuola
I marcatori molecolari. Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze
I marcatori molecolari Dipartimento di Agraria Sez. di Agronomia, Coltivazioni Erbacee e Genetica Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI
 VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
Lezioni di biotecnologie
 Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
Applicazione della biologia molecolare nella valutazione del benessere del cavallo
 UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
UNIVERSITA DEGLI STUDI DI PERUGIA FACOLTA DI MEDICINA VETERINARIA Centro di Studio del Cavallo Sportivo Applicazione della biologia molecolare nella valutazione del benessere del cavallo Andrea Verini
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Marcatori genetici delle piante
 Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
AZIENDA OSPEDALIERA UNIVERSITARIA INTEGRATA - VERONA (D.Lgs. n. 517/ Art. 3 L.R.Veneto n. 18/2009) D I M T
 1 Allegato 1 D I M T PROCEDURA APERTA PER LA FORNITURA QUINQUENNALE PER IL DIPARTIMENTO INTERAZIENDALE DI MEDICINA TRASFUSIONALE DI PRODOTTI PER LA TIPIZZAZIONE GENOMICA A BASSA E ALTA RISOLUZIONE SUDDIVISA
1 Allegato 1 D I M T PROCEDURA APERTA PER LA FORNITURA QUINQUENNALE PER IL DIPARTIMENTO INTERAZIENDALE DI MEDICINA TRASFUSIONALE DI PRODOTTI PER LA TIPIZZAZIONE GENOMICA A BASSA E ALTA RISOLUZIONE SUDDIVISA
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
Reg. (CE) 2073, punto 24 delle considerazioni preliminari
 I risultati delle analisi dipendono dal metodo analitico utilizzato; pertanto occorre associare ad ogni criterio microbiologico un metodo di riferimento specifico. Tuttavia, gli operatori del settore alimentare
I risultati delle analisi dipendono dal metodo analitico utilizzato; pertanto occorre associare ad ogni criterio microbiologico un metodo di riferimento specifico. Tuttavia, gli operatori del settore alimentare
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI
 LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Diagnosi Molecolare delle
 Diagnosi Molecolare delle Malattie infettive Diagnosimolecolare Biologiamolecolare Cosa è la Biologia molecolare? E una branca della biologia che studia gli esseri viventi a livello dei meccanismi molecolari
Diagnosi Molecolare delle Malattie infettive Diagnosimolecolare Biologiamolecolare Cosa è la Biologia molecolare? E una branca della biologia che studia gli esseri viventi a livello dei meccanismi molecolari
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola)
 Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
Corso STEM Il papilloma virus (Prof.ssa Gabriella Di Cola) Il papilloma virus (HPV) interessa maggiormente le donne intorno ai 25 anni di età; esso intacca la zona genitale, in particolare le cellule dell
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Caratterizzazione genetica ed assegnazione geografica di testuggini (Testudo hermanni) di origine ignota
 Caratterizzazione genetica ed assegnazione geografica di testuggini (Testudo hermanni) di origine ignota Scopo: caratterizzare geneticamente testuggini provenienti da sequestri per identificare animali
Caratterizzazione genetica ed assegnazione geografica di testuggini (Testudo hermanni) di origine ignota Scopo: caratterizzare geneticamente testuggini provenienti da sequestri per identificare animali
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
L informatizzazione dei laboratori di genetica. Simona Cavani Maria Isola Parodi Ornella Mazzetti
 L informatizzazione dei laboratori di genetica Simona Cavani Maria Isola Parodi Ornella Mazzetti Laboratorio di Genetica Struttura e patologie analizzate Segreteria / Accettazione Citogenetica -Cariotipo
L informatizzazione dei laboratori di genetica Simona Cavani Maria Isola Parodi Ornella Mazzetti Laboratorio di Genetica Struttura e patologie analizzate Segreteria / Accettazione Citogenetica -Cariotipo
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Importanza della genetica dei microrganismi
 Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Materiale genetico presente nella cellula batterica. Cromosoma batterico Plasmidi Elementi genetici trasponibili DNA fagico
 Genetica batterica Materiale genetico presente nella cellula batterica Cromosoma batterico Plasmidi Elementi genetici trasponibili DNA fagico Nucleoide (morfologia) È costituito da un unica unica molecola
Genetica batterica Materiale genetico presente nella cellula batterica Cromosoma batterico Plasmidi Elementi genetici trasponibili DNA fagico Nucleoide (morfologia) È costituito da un unica unica molecola
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
PATOGENI EMERGENTI NEL LATTE E PROSPETTIVE PER LA VENDITA DEL LATTE CRUDO
 5 Congresso annuale Mastitis Council Italia PATOGENI EMERGENTI NEL LATTE E PROSPETTIVE PER LA VENDITA DEL LATTE Daminelli P., Cosciani Cunico E., Losio M.N., Finazzi G., Bonometti E., Todeschi S., Boni
5 Congresso annuale Mastitis Council Italia PATOGENI EMERGENTI NEL LATTE E PROSPETTIVE PER LA VENDITA DEL LATTE Daminelli P., Cosciani Cunico E., Losio M.N., Finazzi G., Bonometti E., Todeschi S., Boni
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli. I nostri inquilini invisibili
 Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
La mappatura dei geni umani. SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione
 La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico
 Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico Perché identificare insetti con tecniche molecolari? Identificazione morfologica Identificazione
Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico Perché identificare insetti con tecniche molecolari? Identificazione morfologica Identificazione
Caratteristiche prestazionali
 Caratteristiche prestazionali Kit RNeasy DSP FFPE, Versione 1 73604 Gestione della versione Il presente è il documento Caratteristiche prestazionali del kit RNeasy DSP FFPE, Versione 1, R1. Prima di eseguire
Caratteristiche prestazionali Kit RNeasy DSP FFPE, Versione 1 73604 Gestione della versione Il presente è il documento Caratteristiche prestazionali del kit RNeasy DSP FFPE, Versione 1, R1. Prima di eseguire
7. Gruppi genici e sequenza ripetute
 7. Gruppi genici e sequenza ripetute contiene materiale protetto da copyright, ad esclusivo uso personale; non è consentita diffusione ed utilizzo di tipo commerciale La duplicazione e la delezione di
7. Gruppi genici e sequenza ripetute contiene materiale protetto da copyright, ad esclusivo uso personale; non è consentita diffusione ed utilizzo di tipo commerciale La duplicazione e la delezione di
UNIVERSITA DEGLI STUDI DI PALERMO FACOLTA DI MEDICINA E CHIRURGIA
 UNIVERSITA DEGLI STUDI DI PALERMO FACOLTA DI MEDICINA E CHIRURGIA CDL IN TECNICHE DI LABORATORIO BIOMEDICO (abilitante all esercizio della professione di Tecnico di Laboratorio Biomedico) Presidente: Prof.
UNIVERSITA DEGLI STUDI DI PALERMO FACOLTA DI MEDICINA E CHIRURGIA CDL IN TECNICHE DI LABORATORIO BIOMEDICO (abilitante all esercizio della professione di Tecnico di Laboratorio Biomedico) Presidente: Prof.
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
DGGE/TTGE. Le differenze nel punto di denaturazione, chimica o termica, dipendono dalla sequenza nucleotidica
 DGGE/TTGE Lungo gradiente di sostanze denaturanti (es. Urea) - DGGE Separazione degli amplimeri con elettroforesi Lungo gradiente temporale di temperatura (TTGE) Le differenze nel punto di denaturazione,
DGGE/TTGE Lungo gradiente di sostanze denaturanti (es. Urea) - DGGE Separazione degli amplimeri con elettroforesi Lungo gradiente temporale di temperatura (TTGE) Le differenze nel punto di denaturazione,
TASSONOMIA O SISTEMATICA
 TASSONOMIA O SISTEMATICA È la branca della batteriologia responsabile della caratterizzazione degli organismi ed organizzazione in gruppi affini (TAXA). NOMENCLATURA CLASSIFICAZIONE IDENTIFICAZIONE taxon
TASSONOMIA O SISTEMATICA È la branca della batteriologia responsabile della caratterizzazione degli organismi ed organizzazione in gruppi affini (TAXA). NOMENCLATURA CLASSIFICAZIONE IDENTIFICAZIONE taxon
IL RUOLO DEL LABORATORIO DI RIFERIMENTO REGIONALE Anna Barbui S.C. Microbiologia AOU San Giovanni Battista di Torino
 Azienda Ospedaliero-Universitaria San Giovanni Battista di Torino IL RUOLO DEL LABORATORIO DI RIFERIMENTO REGIONALE Anna Barbui S.C. Microbiologia AOU San Giovanni Battista di Torino Workshop Sorveglianza
Azienda Ospedaliero-Universitaria San Giovanni Battista di Torino IL RUOLO DEL LABORATORIO DI RIFERIMENTO REGIONALE Anna Barbui S.C. Microbiologia AOU San Giovanni Battista di Torino Workshop Sorveglianza
I VIRUS. Tutti i virus esistono in due stati: EXTRACELLULARE e INTRACELLULARE. Nel primo caso si parla comnemente di virioni o particelle virali.
 I VIRUS Un virus è definito come materiale nucleico (DNA o RNA) organizzato in una struttura di rivestimento proteico. Il materiale nucleico del virus contiene l informazione necessaria alla sua replicazione
I VIRUS Un virus è definito come materiale nucleico (DNA o RNA) organizzato in una struttura di rivestimento proteico. Il materiale nucleico del virus contiene l informazione necessaria alla sua replicazione
laboratorio biotecnologie zootecniche BAA/LM 3CFU
 laboratorio biotecnologie zootecniche BAA/LM 3CFU Calendario accademico Inizio delle Lezioni: 6 Marzo 2012 Fine delle Lezioni : 5 Giugno 2012 Inizio vacanze di Pasqua: 6 Aprile Fine vacanze di Pasqua:
laboratorio biotecnologie zootecniche BAA/LM 3CFU Calendario accademico Inizio delle Lezioni: 6 Marzo 2012 Fine delle Lezioni : 5 Giugno 2012 Inizio vacanze di Pasqua: 6 Aprile Fine vacanze di Pasqua:
04_biochimica. MFN0366-A1 (I. Perroteau) - Tecniche biochimiche-molecolari: Western blot
 1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
EPIDEMIOLOGIA MOLECOLARE DELLE INFEZIONI NOSOCOMIALI
 MANI PULITE E QUALITÀ NELL ASSISTENZA SANITARIA Governare il rischio infettivo. 2 a parte Pescara 28 marzo 2008 EPIDEMIOLOGIA MOLECOLARE DELLE INFEZIONI NOSOCOMIALI PRINCIPI ED APPLICAZIONI A SUPPORTO
MANI PULITE E QUALITÀ NELL ASSISTENZA SANITARIA Governare il rischio infettivo. 2 a parte Pescara 28 marzo 2008 EPIDEMIOLOGIA MOLECOLARE DELLE INFEZIONI NOSOCOMIALI PRINCIPI ED APPLICAZIONI A SUPPORTO
Genomica e Proteomica: presente e futuro nelle rilevazione e tipizzazione dei meccanismi di resistenza
 Genomica e Proteomica: presente e futuro nelle rilevazione e tipizzazione dei meccanismi di resistenza Mosè Favarato Azienda Ulss 12 Veneziana - Dipartimento di Patologia Clinica Laboratorio Analisi SS
Genomica e Proteomica: presente e futuro nelle rilevazione e tipizzazione dei meccanismi di resistenza Mosè Favarato Azienda Ulss 12 Veneziana - Dipartimento di Patologia Clinica Laboratorio Analisi SS
Percorso formativo di BIOLOGIA. I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico)
 Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
Percorso formativo di BIOLOGIA I Biennio (Lo studio scientifico della vita Dal macroscopico al microscopico) Principi di classificazione e filogenesi degli organismi viventi e basi dell evoluzione Le principali
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI
 DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
WORKLOAD molecolare. Valutazione del carico di lavoro tempo uomo laboratorio genetica molecolare
 WORKLOAD molecolare Valutazione del carico di lavoro tempo uomo laboratorio genetica molecolare Analisi valutate ESTRAZIONE DNA MANUALE/SEMIAUTOMATICA LAPUCCI ESTRAZIONE DNA AUTOMATICA-LAPUCCI reazione
WORKLOAD molecolare Valutazione del carico di lavoro tempo uomo laboratorio genetica molecolare Analisi valutate ESTRAZIONE DNA MANUALE/SEMIAUTOMATICA LAPUCCI ESTRAZIONE DNA AUTOMATICA-LAPUCCI reazione
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi. 8. Funzionamento dei genomi Il genoma dei procarioti
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del genoma Il genoma degli eucarioti
Genotipizzazione virale
 Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
Genotipizzazione virale Typing Separazione basata su maggiori differenze genetiche tra i membri di una singola specie virale Subtyping Individuazione di differenze stabili all interno di un gruppo di virus
GENOMICA. = conoscenza del DNA. Creazione banche banchedati datidel del DNA. Conoscenza di difenomeni biologici
 GENOMICA = conoscenza del DNA Conoscenza di difenomeni biologici Creazione banche banchedati datidel del DNA DNA Prevenzione e cura curadi dimalattie (screening del (screening DNA del per DNA malattie
GENOMICA = conoscenza del DNA Conoscenza di difenomeni biologici Creazione banche banchedati datidel del DNA DNA Prevenzione e cura curadi dimalattie (screening del (screening DNA del per DNA malattie
1. Quantificazione del genomico e determinazione della purezza
 ESERCITAZIONE 2 1. Quantificazione del genomico e determinazione della purezza Una volta estratto il DNA si procede con la quantificazione. Tale quantificazione viene fatta normalmente con l utilizzo di
ESERCITAZIONE 2 1. Quantificazione del genomico e determinazione della purezza Una volta estratto il DNA si procede con la quantificazione. Tale quantificazione viene fatta normalmente con l utilizzo di
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Sequenziamento del DNA: strutture dei nucleotidi
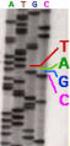 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Aggiornamenti di biologia molecolare nella diagnostica microbiologica. Pier Sandro Cocconcelli
 Aggiornamenti di biologia molecolare nella diagnostica microbiologica Pier Sandro Cocconcelli Istituto di Microbiologia Centro Ricerche Biotecnologiche Università Cattolica del Sacro Cuore Piacenza-Cremona
Aggiornamenti di biologia molecolare nella diagnostica microbiologica Pier Sandro Cocconcelli Istituto di Microbiologia Centro Ricerche Biotecnologiche Università Cattolica del Sacro Cuore Piacenza-Cremona
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Epidemiologia molecolare delle malattie infettive: metodi di analisi ed interpretazione dei risultati
 10 Ann Ig 2014; 26: 10-45 doi:10.7416/ai.2014.1956 M.L. Sammarco et al. Epidemiologia molecolare delle malattie infettive: metodi di analisi ed interpretazione dei risultati M.L. Sammarco*, G. Ripabelli*,
10 Ann Ig 2014; 26: 10-45 doi:10.7416/ai.2014.1956 M.L. Sammarco et al. Epidemiologia molecolare delle malattie infettive: metodi di analisi ed interpretazione dei risultati M.L. Sammarco*, G. Ripabelli*,
Analisi degli alimenti tramite PCR Real-Time. Introduzione ad una tecnologia all'avanguardia
 Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE:
 GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
GENOMICA STRUTTURALE: GENOMICA FUNZIONALE: 1. Anatomia dei genomi 8. Funzionamento dei genomi Il genoma dei procarioti Modificazioni della cromatina e l espressione del Il genoma degli eucarioti genoma
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
CORSO INTEGRATO DI GENETICA a.a. 2011-2012. Genetica molecolare in medicina: Analisi di Mutazioni. Cristina Bombieri 7 dicembre 2011
 CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico Padano Pietro Piffanelli, Pamela Abbruscato, barbara Menin
 Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
Progetto POLORISO Incontro di coordinamento della attività scientifica del progetto CRA - Unità di Ricerca per la risicoltura Vercelli, aprile 0 Genetica, genomica ed innovazione varietale UO9, Parco Tecnologico
Principali classi di mutazioni
 Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
Principali classi di mutazioni Regole per valutare la patogenicità di una mutazione I - Delezioni dell intero gene, mutazioni non senso e anomalie della cornice di lettura molto probabilmente distruggono
MICRORGANISMI PROBIOTICI: caratterizzazione fenotipica, genotipica e probiotica. Alfonsina Fiore, Maria Casale
 Workshop FOCUS SU SICUREZZA D USO E NUTRIZIONALE DEGLI ALIMENTI 21-22 22 Novembre 2005 MICRORGANISMI PROBIOTICI: caratterizzazione fenotipica, genotipica e probiotica Alfonsina Fiore, Maria Casale C.N.Q.R.A.
Workshop FOCUS SU SICUREZZA D USO E NUTRIZIONALE DEGLI ALIMENTI 21-22 22 Novembre 2005 MICRORGANISMI PROBIOTICI: caratterizzazione fenotipica, genotipica e probiotica Alfonsina Fiore, Maria Casale C.N.Q.R.A.
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
