Il Sequenziamento Tecniche e Strategie
|
|
|
- Gianluca Tommaso Mauri
- 8 anni fa
- Просмотров:
Транскрипт
1 Il Sequenziamento Tecniche e Strategie
2 Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento con reagenti chimici che tagliano la sequenza nelle posizioni di specifici nucleotidi La molecola di ds DNA viene marcata al 5 di ogni filamento (gdatp) e poi denaturata. Metodo a terminazione di catena (Sanger et al., 1977) La sequenza di una molecola di ss DNA viene determinata mediante sintesi enzimatica di catene polinucleotidiche complementari che terminano nelle posizioni di specifici nucleotidi
3 Sequenziamento secondo Maxam e Gilbert 1 Marcatura terminale al 5 del DNA a doppio filamento 2 Denaturazione e separazione dei due filamenti 3 Il DNA a singola elica viene suddiviso in quattro campioni, ognuno dei quali viene trattato con un reagente chimico che degrada una o due delle 4 basi del DNA G = DMS + piperidina G + A = DMS + piperidina + acido formico C + T = idrazina + piperidina C = idrazina + piperidina in NaCl 1,5 M Si utilizzano concentrazioni dei reagenti tali che ogni singola copia del filamento di DNA marcato venga degradata una sola volta; poichè il filamento è presente in molteplici copie, statisticamente, considerando tutte le copie, ogni base del filamento verrà degradata almeno una volta e quindi si produrranno tanti frammenti di lunghezza diversa a seconda del sito di taglio. 4 Separazione dei frammenti marcati mediante elettroforesi su gel di poliacrilammide e visualizzazione dei risultati mediante autoradiografia
4
5
6
7 Sequenziamento secondo Sanger 1 La reazione si basa sulla sintesi del filamento complementare al ssdna che serve da stampo (templato) La reazione è catalizzata da una DNA pol. con le seguenti caratteristiche: Elevato grado di processività Attività esonucleasica 5-3 trascurabile Attività esonucleasica 3-5 trascurabile La reazione richiede dntp (datp, dgtp, dctp,dttp) come substrato Inoltre alla reazione viene aggiunto un ddntp che viene incorporato nella catena dalla polimerasi che non distingue tra dntp e ddntp. All incorporazione del ddntp si arresta la sintesi perché manca il gruppo ossidrilico al 3 necessario per la formazione del legame con il nucleotide successivo Siccome è presente sia il dntp (es.datp), che il ddntp (es.ddatp), la sintesi non si interrompe alla prima T dello stampo ma prosegue per X nucleotidi fino alla casuale incorporazione del ddatp. Il risultato è un insieme di nuove catene di diversa lunghezza tutte terminanti con un ddntp Si effettuano 4 reazioni distinte ognuna addizionata di un ddntp (A,G,C,T)
8
9
10 Sequenziamento di DNA clonato Il primer (innesco) è necessario per l inizio della sintesi da parte della DNA pol. Il primer stabilisce la regione del DNA stampo che verrà sequenziata: Primer universale primer sequenza 3 vettore Inserto di DNA 5 Primer interno, sequenza-specifico primer sequenza 3 vettore Inserto di DNA 5
11 Sequenziamento automatico con dideossinucleotidi marcati per fluorescenza ddntp con marcatore fluorescente dda dda ddc ddg ddt Reazioni di sequenziamento Frazionamento dei prodotti ddt ddc ddg dda dda Rivelatore di fluorescenza dda Sistema di visualizzazione CACCGCATCGAAATTAACTTCCAAGTTAAGCTTGG
12 Dye Terminator Labeling Strategy Coniugando a ciascun ddntp un diverso marcatore fluorescente, è possibile effettuare le quattro reazioni di sequenziamento in un unica provetta I frammenti marcati vengono separati attraverso elettroforesi capillare e nella porzione terminale del capillare sono colpiti dal raggio laser che eccita le molecole fluorescenti ad essi legate. Le emissioni fluorescenti vengono captate da un rilevatore. Le informazioni vengono integrate e trasformate in picchi di colore diverso, con aree proporzionali all intensità di emissione.
13 Cycle Sequencing Utilizzo di una Taq Polimerasi Termostabile Possibilità di usare T. annealing dell oligo precise Possibilità di risolvere facilmente strutture complesse denaturation 96 C annealing 50 C rxn. mixture extension 60 C A C G T labeled products A C G T A A C G T A C G T G
14 Perché si sequenzia??? Informazioni su cloni, presenza e orientamento inserto Informazioni su amplificati di DNA, presenza di aspecifici. Presenza di siti di restrizione, inserzioni, delezioni, sostituzioni, mutazioni puntiformi, eterozigosi. Diagnosi di malattie genetiche. Presenza di regioni codificanti, regioni regolatrici, microsatelliti, ecc. E.S.T. project, creazione di banche dati. Sequenziamento su larga scala, sequenziamento genomi.
15
16
17
18
19 Mappe genetiche e fisiche Mappatura Fisica Fa uso di tecniche di biologia molecolare per costruire mappe basate su tratti distintivi della sequenza Le tecniche principali sono: le mappe di restrizione, che localizzano la posizione relativa dei siti di taglio delle endonucleasi di restrizione l ibridazione in situ con fluorescenza (FISH) la mappatura mediante STS (Sequence Tagged Site)
20 Sito a Sequenza Etichettata STS è una breve sequenza di DNA, bp, facilmente riconoscibile e presente come sequenza unica nel cromosoma/genoma Una sequenza di DNA per essere considerata una STS deve rispondere a 2 caratteristiche: la sua sequenza deve essere nota, così da verificarne la presenza nei frammenti di DNA mediante PCR deve avere localizzazione unica nel cromosoma/genoma (single copy) in esame Le fonti più comuni di STS sono: le EST (Expressed Sequence Tags), sequenze espresse corrispondenti a porzioni di cdna di geni presenti in singola copia gli SSLP (Simple Sequence Length Polymorphisms) rappresentano un collegamento diretto tra mappe genetiche e mappe fisiche elementi inserzionali stabili (trasposoni)
21 Mappe genetiche e fisiche Mappatura genetica è costruita con tecniche genetiche che mostrano la posizione di geni o altri marcatori. Le tecniche consistono in esperimenti di incrocio I marcatori genici sono utilizzabili quando sono noti alleli mutanti dotati di fenotipo. I fenotipi possono essere macroscopici (colore degli occhi in Drosophila) ma anche biochimici (gruppi sanguigni) I marcatori di DNA devono anch essi esser presenti in almeno 2 forme alleliche. Se ne conoscono 3 tipi: i Polimorfismi della Lunghezza dei Frammenti di Restrizione (RFLP) i Polimorfismi della Lunghezza di Sequenze Semplici (SSLP) i Polimorfismi di Singoli Nucleotidi (SNP)
22 La mappatura genetica è basata sull analisi di associazione genetica sulle leggi di Mendel La risoluzione di una mappa genetica dipende dal numero di crossing over che sono stati individuati. Ciò rappresenta un grosso limite per gli eucarioti superiori x cui si ottiene una progenie poco numerosa Le mappe genetiche hanno un accuratezza limitata, a causa dell esistenza di hot spots di ricombinazione
23 POLIMORFISMO La presenza nella popolazione di due o più varianti (alleli, fenotipi, varianti di sequenza, varianti di struttura cromosomica) con frequenze significative. Un locus è considerato polimorfico se presenta almeno due alleli dei quali il più raro ha una frequenza maggiore dell 1%.
24 Polimorfismi della Lunghezza dei Frammenti di Restrizione (RFLP) Gli RFLP sono conseguenza di una mutazione puntiforme ma in un sito di restrizione Per ogni RFLP ci sono solo 2 possibili alleli, con o senza sito di restrizione Vengono individuati sia mediante analisi Southern che mediante PCR Polimorfismi della Lunghezza di Sequenze Semplici (SSLP) Gli SSLP sono insiemi di sequenze ripetute che presentano variazioni di lunghezza Per ogni SSLP ci sono più possibili alleli, a seconda di quante unità ripetute presentano esistono 2 tipi di SSLP: minisatelliti o Ripetizioni in Tandem a Numero Variabile (VNTR), in cui la lunghezza dell unità ripetuta è di 25bp max. Principalmente localizzati nelle regioni telomeriche microsatelliti o Ripetizioni in Tandem Semplici (STR), le cui ripetizioni sono generalmente di unità di/tetranucleotidiche. Dispersi in tutto il genoma
25 VNTR Variable Number of Tandem Repeat Numero variabile di ripetizioni in tandem Gli alleli differiscono per il numero variabile delle unità ripetute in tandem Ereditarietà mendeliana Molti alleli Elevata eterozigosità MINISATELLITI Unità ripetuta: 20bp centinaia bp Dimensione alleli: poche centinaia kb Localizzazione subtelomerica MICROSATELLITI STR short tandem repeat Unità ripetuta: 2-4 bp Dimensione alleli: bp Ben distribuiti nel genoma Tri- e tetra-nucleotidici Multiplexing / automazione
26 Polimorfismi di Singoli Nucleotidi (SNP) Gli SNP sono mutazioni puntiformi che però non causano RFLP Per ogni SNP in genere sono presenti solo 2 alleli, anche se in teoria dovrebbero essere 4 alleli... Tra i vantaggi degli SNP ci sono sia la loro abbondanza (1.42 milioni nel genoma umano), che la loro rapida identificazione mediante ibridazione Tipizzati in grande scala con sistemi automatizzati (microchip)
27 RFLP SNP VNTR Sito restrizione un nucleotide n ripetizioni 2 alleli 2 alleli molti alleli E. Mendeliana E. mendeliana E. mendeliana PCR restrizione seq.automatici PCR microchip
28
29 RFLP PCR sito polimorfico Southern sito polimorfico R 1 R 2 R 3 mappa di restrizione R 1 R 2 R 3 primer mappa di restrizione sonda a DNA digestione con R, gel e trasferimento ibridazione PCR + digestione membrana di nylon elettroforesi su gel 2 3 bande di ibridazione Il campione 3 è polimorfico autoradiografia SSLP Caratterizzazione mediante PCR allele1 TCTGAGAGAGGC allele2 TCTGAGAGAGAGAGGC allele2 allele1
30
31
32
33 Analisi Paternità
34
35
36 Ibridazione in situ con fluorescenza FISH consente di visualizzare direttamente la posizione di un marcatore all interno di un cromosoma o su una moleocla di DNA distesa il marcatore è una sequenza di DNA che viene osservata ibridando con una sonda fluorescente non utilizza più sonde radioattive P 32 /H 3 ma fluorescenti che combinano un alta sensibilità con un alta risoluzione Applicazioni della FISH su cromosomi metafasici, in cui il segnale è misurato rispetto all estremità del braccio corto del cromosoma (valore FLpter). La risoluzione è pari ad 1Mb su cromosomi metafasici distesi meccanicamente, mediante un passaggio di centrifugazione. La risoluzione è pari a 2/300Kb su cromosomi in interfase, lo stadio del ciclo in cui i cromosomi sono meno condensati. La risoluzione è pari a 25Kb
37
38 A metaphase cell positive for the bcr/abl rearrangement (associated with chronic myelogenous leukemia) using FISH. The chromosomes can be seen in blue. The chromosome that is labeled with green and red spots (upper left) is the one where the rearrangement is present.
Mappe fisiche. Si basano sulla localizzazione fisica delle molecole di DNA
 Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
Mappe fisiche Si basano sulla localizzazione fisica delle molecole di DNA Costruzione di una mappa fisica diversi metodi - Mappe a bassa risoluzione - Mappe ad alta risoluzione Risoluzione= distanza a
ANALISI MOLECOLARE DEL GENOMA
 ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
ANALISI MOLECOLARE DEL GENOMA Come sono disposti i geni nel genoma? MAPPA GENETICA: un set ordinato di geni sul cromosoma, la distanza tra i quali è espressa in unità di ricombinazione genetica (centimorgan)
IL SEQUENZIAMENTO DEL DNA. Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA.
 IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
IL SEQUENZIAMENTO DEL DNA Obiettivo: ottenere la sequenza nucleotidica di un frammento di DNA. Il DNA può essere sequenziato generando frammenti di DNA la cui lunghezza dipende dall ultima base della sequenza.
GENOMA. Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine CONTENUTO FUNZIONE. Progetti genoma in centinaia di organismi
 GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
La variabilità genetica: tipi e metodi per studiarla e misurarla
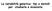 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
La mappatura dei geni umani. SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione
 La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
Tecniche di sequenziamento del DNA
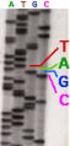 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Strumenti della Genetica Molecolare Umana (3) Capitoli 6-7-8 Piccolo test: trova al differenza Gel 2 Gel 1 Gel 1. digestione DNA genomico Gel 2. Digestione inserto cloni genomici BANCHE DI DNA RICOMBINANTE
Polimorfismi e la variazione genetica
 Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Polimorfismi e la variazione genetica Variazione individuale nel DNA (con frequenza 1% nella popolazione) SNP (single nucleotide polymorphism)è definito come una variazione a livello di un singolo paio
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Nested
7. Gruppi genici e sequenza ripetute
 7. Gruppi genici e sequenza ripetute contiene materiale protetto da copyright, ad esclusivo uso personale; non è consentita diffusione ed utilizzo di tipo commerciale La duplicazione e la delezione di
7. Gruppi genici e sequenza ripetute contiene materiale protetto da copyright, ad esclusivo uso personale; non è consentita diffusione ed utilizzo di tipo commerciale La duplicazione e la delezione di
Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin»
 Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin» E-mail: [email protected] Individuare in modo UNIVOCO un individuo (parentela, reati, ecc) da una data traccia
Beniamino Trombetta, PhD Dipartimento di Biologia e Biotecnologie «Charles Darwin» E-mail: [email protected] Individuare in modo UNIVOCO un individuo (parentela, reati, ecc) da una data traccia
Strumenti della Genetica Molecolare Umana (5)
 Strumenti della Genetica Molecolare Umana (5) Prof. Mario Ventura Capitoli 6-7-8 LEZIONE SUMMARY PER TECNICHE GIORNO 23 OTTOBRE LEZIONE AGGIUNTIVA GIORNO 30 NOVEMBRE ORE 8:30-11:00 Prof. Mario Ventura
Strumenti della Genetica Molecolare Umana (5) Prof. Mario Ventura Capitoli 6-7-8 LEZIONE SUMMARY PER TECNICHE GIORNO 23 OTTOBRE LEZIONE AGGIUNTIVA GIORNO 30 NOVEMBRE ORE 8:30-11:00 Prof. Mario Ventura
Sequenziamento del DNA: strutture dei nucleotidi
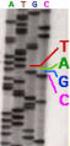 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp ( Mbp=150Gbp) di una
 Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp (150.000Mbp=150Gbp) di una Liliacea. Tra le graminacee il frumento ha un genoma
Genomi vegetali Da 7x10 7 bp per genoma aploide (130Mbp diploide, 5 cromosomi) di Arabidopsis thaliana alle 1,5x10 11 bp (150.000Mbp=150Gbp) di una Liliacea. Tra le graminacee il frumento ha un genoma
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Metodologia
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
CORSO INTEGRATO DI GENETICA a.a. 2011-2012. Genetica molecolare in medicina: Analisi di Mutazioni. Cristina Bombieri 7 dicembre 2011
 CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA
 CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA Dott.ssa Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due
CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2012-2013 31/10/2012 Lezioni 25 e 26 I polimorfismi del DNA Dott.ssa Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
REPLICAZIONE DEI GENOMI A DNA
 REPLICAZIONE DEI GENOMI A DNA LA REPLICAZIONE DEL DNA SERVE A PERPETUARE L INFORMAZIONE GENICA FIGURA 12.26 GENES VII FIGURA 13.1 GENOMI II FIGURA 13.2 GENOMI II NEL 1958 MESELSON E STAHL DIMOSTRANO CHE
REPLICAZIONE DEI GENOMI A DNA LA REPLICAZIONE DEL DNA SERVE A PERPETUARE L INFORMAZIONE GENICA FIGURA 12.26 GENES VII FIGURA 13.1 GENOMI II FIGURA 13.2 GENOMI II NEL 1958 MESELSON E STAHL DIMOSTRANO CHE
Marcatori genetici delle piante
 Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
Marcatori genetici delle piante Marcatore genetico: locus genico che identifica univocamente una regione cromosomica Polimorfismo genetico: Presenza di varianti alleliche per un dato locus (gene o marcatore)
DNA - DENATURAZIONE E RIASSOCIAZIONE
 DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
DNA - DENATURAZIONE E RIASSOCIAZIONE Il doppio strand della molecola di DNA può denaturarsi dando due singoli strand ad alte temperature (>90 C). Due strand di DNA complementari possono accoppiarsi dando
Contenuto di DNA aploide in alcune specie
 Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
Contenuto di DNA aploide in alcune specie 1-10 2 kb 10 3 kb 10 4 kb 10 5-10 8 kb Dimensioni del genoma Paradosso del valore C Non c è una correlazione tra la quantità di DNA e la complessità di un organismo
I marcatori genetici e loro applicazioni nelle produzioni animali. Dott.ssa Chiara Targhetta
 I marcatori genetici e loro applicazioni nelle produzioni animali Dott.ssa Chiara Targhetta LOCUS localizzazione genomica unica all interno di un cromosoma; permette di definire la posizione di un gene
I marcatori genetici e loro applicazioni nelle produzioni animali Dott.ssa Chiara Targhetta LOCUS localizzazione genomica unica all interno di un cromosoma; permette di definire la posizione di un gene
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Progetto Tandem Biologia saperi minimi Anno accademico Marzo 2012 COGNOME...
 Progetto Tandem Biologia saperi minimi Anno accademico 2011-2012 2 Marzo 2012 COGNOME... NOME 1) Quali delle seguenti affermazioni sulla struttura primaria delle proteine è falsa? a) può essere ramificata
Progetto Tandem Biologia saperi minimi Anno accademico 2011-2012 2 Marzo 2012 COGNOME... NOME 1) Quali delle seguenti affermazioni sulla struttura primaria delle proteine è falsa? a) può essere ramificata
Polimorfismi del DNA e loro applicazioni
 Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
Polimorfismi del DNA e loro applicazioni WWW.FISIOKINESITERAPIA.BIZ Lo sviluppo dei marcatori genetici nell uomo TIPO DI MARCATORE N DI LOCI CARATTERISTICHE Gruppi sanguigni ~ 20 Può esserci bisogno di
CORSO INTEGRATO DI GENETICA. a.a /11/2010. Analisi di linkage Analisi di mutazioni
 CORSO INTEGRATO DI GENETICA a.a. 2010-2011 04/11/2010 Lezioni 27_28 Analisi di linkage Analisi di mutazioni Dott.ssa Elisabetta Trabetti LINKAGE Geni in loci vicini sullo stesso cromosoma - concatenati
CORSO INTEGRATO DI GENETICA a.a. 2010-2011 04/11/2010 Lezioni 27_28 Analisi di linkage Analisi di mutazioni Dott.ssa Elisabetta Trabetti LINKAGE Geni in loci vicini sullo stesso cromosoma - concatenati
Lezione 4. Il sequenziamento del DNA, Sanger
 Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
Lezione 4 Il sequenziamento del DNA, Sanger Schema della lezione Polymerase chain reaction (PCR) Dal prodotto di PCR al sequenziamento di Sanger Lettura dei prodotti di sequenziamento con sequenziatori
IBRIDAZIONE MOLECOLARE E MAPPATURA DEI GENI
 IBRIDAZIONE MOLECOLARE E MAPPATURA DEI GENI La possibilità di visualizzare la posizione dei geni o di altre regioni non codificanti del genoma su un cromosoma (mappatura) consente anche l individuazione
IBRIDAZIONE MOLECOLARE E MAPPATURA DEI GENI La possibilità di visualizzare la posizione dei geni o di altre regioni non codificanti del genoma su un cromosoma (mappatura) consente anche l individuazione
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
Southern blot. !Per. determinare la presenza o assenza di specifici frammenti genici. !Studio. della struttura e dell organizzazione di un gene
 Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Marcatori molecolari per l analisi genica, genetica e genomica
 Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Prof. Mario Ventura. Mappatura fisica
 Mappatura fisica Tipi di mappe: mappe fisiche e genetiche Mappe fisiche: localizza i geni (o marcatori) lungo i cromosomi usando misurazioni che sono il riflesso di distanza fisica. Si distingue a bassa
Mappatura fisica Tipi di mappe: mappe fisiche e genetiche Mappe fisiche: localizza i geni (o marcatori) lungo i cromosomi usando misurazioni che sono il riflesso di distanza fisica. Si distingue a bassa
Duplicazione del DNA. 6 Dicembre 2007
 Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Duplicazione del DNA 6 Dicembre 2007 Duplicazione - Trascrizione - Traduzione DNA Trascrizione DNA - La DUPLICAZIONE è il processo che porta alla formazione di copie delle molecole di DNA ed al trasferimento
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico
 Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico Perché identificare insetti con tecniche molecolari? Identificazione morfologica Identificazione
Tecniche molecolari in uso per la caratterizzazione ed identificazione di artropodi di interesse economico Perché identificare insetti con tecniche molecolari? Identificazione morfologica Identificazione
Genomi dei procarioti
 Genomi dei procarioti Una molecola circolare di DNA E.coli circa 4 x 10 6 coppie di basi Il genoma è quasi tutto codificante Viene trascritto in mrna policistronici Il genoma eucariotico Il genoma eucariotico
Genomi dei procarioti Una molecola circolare di DNA E.coli circa 4 x 10 6 coppie di basi Il genoma è quasi tutto codificante Viene trascritto in mrna policistronici Il genoma eucariotico Il genoma eucariotico
Sommario. Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni
 Sommario Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni SNP: definizioni Polimorfismo al singolo nucleotide, in genere si intende la sostituzione
Sommario Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni SNP: definizioni Polimorfismo al singolo nucleotide, in genere si intende la sostituzione
TRASCRIZIONE DEL DNA. Formazione mrna
 TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
TRASCRIZIONE DEL DNA Formazione mrna Trascrizione Processo mediante il quale l informazione contenuta in una sequenza di DNA (gene) viene copiata in una sequenza complementare di RNA dall enzima RNA polimerasi
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Program ma Testi consigliati:
 Program ma Testi consigliati: 1. Genetica molecolare umana, Strachan &Read, Ed. UTET 2. Genetica in Medicina Thompson & Thompson Ed.Idelson-Gnocchi Comunicazioni e cambi orari Giorno 18 Ottobre non ci
Program ma Testi consigliati: 1. Genetica molecolare umana, Strachan &Read, Ed. UTET 2. Genetica in Medicina Thompson & Thompson Ed.Idelson-Gnocchi Comunicazioni e cambi orari Giorno 18 Ottobre non ci
a.a /10/2015 Lezioni 21 e 22 I polimorfismi del DNA
 CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2015-2016 29/10/2015 Lezioni 21 e 22 I polimorfismi del DNA Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due o piu varianti
CORSO INTEGRATO di Genetica e Biologia Molecolare GENETICA a.a. 2015-2016 29/10/2015 Lezioni 21 e 22 I polimorfismi del DNA Elisabetta Trabetti POLIMORFISMO La presenza nella popolazione di due o piu varianti
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
Ampliamenti delle leggi di Mendel
 Ampliamenti delle leggi di Mendel 1 La dominanza incompleta Nella dominanza incompleta gli eterozigoti mostrano un fenotipo intermedio a quello dei genitori, in accordo con le leggi di Mendel. Entrambi
Ampliamenti delle leggi di Mendel 1 La dominanza incompleta Nella dominanza incompleta gli eterozigoti mostrano un fenotipo intermedio a quello dei genitori, in accordo con le leggi di Mendel. Entrambi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ANALISI DI SEQUENZE DI DNA OBIETTIVI DELLA GENOMICA MAPPE GENETICHE e FISICHE sempre più accurate SEQUENZIAMENTO GENOMI GENOMICA COMPARATIVA confronto tra genomi
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20
 SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
SOLUZIONI AI PROBLEMI DEL CAPITOLO 20 Domande concettuali C1. Si potrebbe concludere che la donna porta una delezione nel gene che è riconosciuto dalla sonda. Per clonare questo gene, potresti iniziare
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona
 Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
Laboratorio di diagnostica Molecolare Dipartimento di Patologia Sezione di Anatomia Patologica Università di Verona Il tecnico di laboratorio biomedico: Applicazioni nel laboratorio di anatomia patologica
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
MICROARRAY Esempio generico
 MICROARRAY MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione
MICROARRAY MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
La genetica molecolare
 La genetica molecolare 1 Il materiale genetico Varia di quantità da specie a specie. Regola lo sviluppo della cellula. Ha la capacità di duplicarsi. Nome comune Numero di coppie di cromosomi zanzara 3
La genetica molecolare 1 Il materiale genetico Varia di quantità da specie a specie. Regola lo sviluppo della cellula. Ha la capacità di duplicarsi. Nome comune Numero di coppie di cromosomi zanzara 3
La trascrizione è simile alla replicazione ma esistono alcune importanti differenze
 LA TRASCRIZIONE processo nel quale il DNA stampo viene copiato in una molecola di RNA La molecola di RNA è identica in sequenza all elica codificante e complementare a quella stampo. La trascrizione è
LA TRASCRIZIONE processo nel quale il DNA stampo viene copiato in una molecola di RNA La molecola di RNA è identica in sequenza all elica codificante e complementare a quella stampo. La trascrizione è
Quanto DNA è contenuto in una cellula?...
 Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
Quanto DNA è contenuto in una cellula?... bp = Base Pair (coppie di basi) lungh DNA/lungh struttura ospitante E. coli: 4,6x10 6 bp (1,7 mm) 850 (unica molecola circolare) Cellula umana: 3,2x10 9 bp (~2
CONIUGAZIONE BATTERICA
 CONIUGAZIONE BATTERICA Come si coltivano batteri in laboratorio Terreno minimo: acqua, sali inorganici, fonte di carbonio Batteri prototrofici: crescono in terreno minimo Batteri auxotrofici: Incapaci
CONIUGAZIONE BATTERICA Come si coltivano batteri in laboratorio Terreno minimo: acqua, sali inorganici, fonte di carbonio Batteri prototrofici: crescono in terreno minimo Batteri auxotrofici: Incapaci
BIOTECNOLOGIE ANIMALI: LINKAGE E MARCATORI
 BIOTECNOLOGIE ANIMALI: LINKAGE E MARCATORI Associazione o Linkage Marcatori genetici Su ciascun cromosoma sono presenti più geni, ognuno ad un locus specifico Se i cromosomi venissero trasmessi da una
BIOTECNOLOGIE ANIMALI: LINKAGE E MARCATORI Associazione o Linkage Marcatori genetici Su ciascun cromosoma sono presenti più geni, ognuno ad un locus specifico Se i cromosomi venissero trasmessi da una
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI
 Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
Dr. Tommaso Giordani SEQUENZIAMENTO SANGER E ASSEMBLAGGIO DEI GENOMI Programma - Strategie di sequenziamento Sanger dei GENOMI COMPLETI sequenziamento gerarchico (clone by clone) sequenziamento shotgun
Il genoma dei batteri
 Il genoma dei batteri Trovare mutazioni nei geni batterici Mutazioni che colpiscono la morfologia della colonia, Mutazioni che conferiscono resistenza agli agenti battericidi Mutazioni che creano auxotrofi
Il genoma dei batteri Trovare mutazioni nei geni batterici Mutazioni che colpiscono la morfologia della colonia, Mutazioni che conferiscono resistenza agli agenti battericidi Mutazioni che creano auxotrofi
