MICROARRAY Esempio generico
|
|
|
- Alessio Mazza
- 9 anni fa
- Просмотров:
Транскрипт
1 MICROARRAY
2 MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione ordinata di migliaia di microscopici spot di DNA, contenenti ognuno poche picomoli di uno specifico frammento di DNA con una precisa sequenza (la sonda)
3 MICROARRAY Esempio generico Superficie di almeno 1 cm x 1 cm di spot o feature per array molecole di sonda (probe) per feature Tutte le molecole di sonda all interno di una feature hanno una sequenza identica Ogni feature ha una sequenza differente
4 MICROARRAY Le tecniche di DNA microarray sono basate sulla microibridazione contemporanea di migliaia di specifici frammenti di DNA Il microarray consiste in un supporto solido recante una successione ordinata di migliaia di microscopici spot di DNA, contenenti ognuno poche picomoli di uno specifico frammento di DNA con una precisa sequenza (la sonda) La marcatura del campione è in genere fluorescente Le diverse tecniche di microarray si possono dividere innanzitutto in base al tipo di applicazione e quindi di campione da analizzare I microarray si possono dividere in base alle tecniche con cui le sonde vengono ottenute e fissate sul supporto
5 MICROARRAY Supporto solido Deve essere di materiali duraturi, stabili ad alte temperature e a lavaggi ad alta concentrazione ionica Deve essere di materiali non porosi e consentire di ridurre al minimo il volume di ibridizzazione Il materiale non deve contribuire significativamente al background (es. deve essere scarsamente fluorescente) La superficie solida può essere plastica, vetro o silicio (gene chip) In genere la superficie è rivestita con una matrice chimica (es. epossi-silano, ammino-silano, poli-lisina, poliacrilammide) che consente il legame delle sonde
6 MICROARRAY Tipi di microarray (In base alle applicazioni ed al tipo di campione) Microarray per analisi di espressione genica: Il campione di partenza è RNA. Esso viene in genere retrotrascritto in cdna o trascritto in crna ed eventualmente amplificato. Microarray per individuazione di SNP, genotipizzazione, sequenziamento, ricerca di mutazioni: Il campione di partenza è DNA genomico, in genere frammentato ed eventualmente amplificato.
7 MICROARRAY Tipi di microarray (in base alle tecniche di sintesi delle sonde) Microarray Spottati: le sonde sono frammenti di DNA a singolo o doppio filamento pre-sintetizzati (in genere ottenuti per PCR) e successivamente depositati sul supporto e legati ad esso (in genere mediante UV-crosslinking). Nel caso di quelli per analisi di espressione, i geni sono in genere rappresentati da singoli frammenti, aventi dimensioni tra 50 e diverse centinaia di paia di basi Microarray di oligonucleotidi ad alta densità: le sonde sono set di oligomeri sintetizzati in situ sul supporto mediante un processo fotolitografico. Nel caso di quelli per analisi di espressione, I geni sono rappresentati da diversi oligonucleotidi di paia di basi
8 Microarray per l analisi dell espressione genica
9 Microarray spottato Produzione Il DNA spottato viene definito probe I probe vengono scelti sia da database (GenBank, dbest and UniGene), sia da librerie di cdna o EST, e collezioni di cloni Gli array a DNA possono essere fabbricati usando: ooligonucleotidi ( pb) ocdna amplificato I probe per array di cdna sono generalmente prodotti di PCR ( kb), ottenuti amplificando sequenze geniche mediante primers vettore specifici, e successivamente stampati come ds-cdna su vetrini in posizioni predefinite (spot)
10 Microarray spottato Produzione Gli spot (sia oligonucleotidi che cdna) sono di circa 150 mm, e sono tutti alla stessa distanza tra loro. Più di 30,000 cdnas possono essere posizionati sulla superficie di un comune vetrino da microscopio Prima dello spottaggio, il vetrino deve essere trattato in modo che esponga sulla superficie molecole con cariche positive (Es. polilisina) Le molecole di DNA che vengono depositate sul vetrino durante lo spottaggio, vengono successivamente UVcrosslinkate alle cariche positive del vetrino
11 Microarray spottato Produzione
12 Microarray spottato Produzione
13 Microarray spottato Legame competitivo L RNA estratto da tessuto o linea cellulare è utilizzato per generare il target marcato, che viene poi ibridizzato con i probe immobilizzati sul vetrino Questo tipo di tecnologia in genere prevede l ibridizzazione contemporanea su ogni vetrino di due cdna marcati, generati da due diversi campioni di RNA (confronto tra campione e controllo) L utilizzo di diverse molecole fluorescenti (Cy3 e Cy5) permette di marcare i due cdna con colori diversi; in questo modo è possibile miscelarli e ibridizzarli sullo stesso vetrino. Si parla di legame competitivo dei target ai probe
14 Microarray spottato RNA di controllo I diversi campioni di un set sperimentale devono essere ibridizzati con lo stesso controllo L RNA di controllo deve essere correlato al campione sperimentale Per esperimenti in vitro il controllo può essere RNA estratto da cellule non trattate, ecc Per campioni di pazienti, si può utilizzare l RNA del tessuto normale corrispondente Se viene analizzato un largo numero di campioni, il controllo può consistere in una miscela di tutti i campioni usati
15 Microarray spottato Numero di replicati e ibridizzazione La marcatura dei target con diverse molecole fluorescenti può produrre una differente efficienza di incorporazione. Quindi è importante avere: ola marcatura reciproca dello stesso campione con entrambi i fluorescenti orepliche dei campioni di esperimenti diversi, da utilizzare come controllo per la variabilità sperimentale opiù di una ibridizzazione: replicati su vetrini diversi Cy3- Cy5- Cy5- Cy3- Cy3- Cy5- Cy5- Cy3-
16 Microarray spottato Marcatura del target e ibridazione Sample RNA Wash Hybridisation Scan 1 Scan 2 Control RNA Image 1 Image 2 Combined image in software L RNA totale (circa 20 mg) dei due campioni da ibridizzare viene retrotrascritto e il cdna marcato rispettivamente con Cy3 (verde) e Cy5 (rosso) Controllo e campione vengono uniti in un unica miscela di ibridizzazione e il vetrino posto in camera di ibridazione overnight Il vetrino viene lavato e la fluorescenza emessa da ogni target è rilevata dallo scanner Il rapporto tra le intensità dei segnali rilevati nei due canali (R/V) rappresenta una misura dell ammontare relativo di specifici mrna in ogni campione
17 Microarray spottato Scannerizzazione del vetrino
18 Array di oligonucleotidi ad alta densità
19 Array di oligonucleotidi ad alta densità Fotolitografia Alcuni array di oligonucleotidi (GeneChip) sono costruiti utilizzando un processo di sintesi di luce-diretta chiamato fotolitografia Linker sintetici modificati con gruppi protettivi rimuovibili mediante la luce, sono legati alla superficie del vetrino. La luce viene diretta attraverso una maschera per deproteggere e attivare i gruppi selezionati. Nucleotidi protetti possono così accoppiarsi ai siti attivati
20 Array di oligonucleotidi ad alta densità Fotolitografia
21 Array di oligonucleotidi ad alta densità GeneChip
22 Array di oligonucleotidi ad alta densità Sintesi senza maschera
23 Array di oligonucleotidi ad alta densità Sintesi senza maschera
24 Array di oligonucleotidi ad alta densità
25 Array di oligonucleotidi ad alta densità
26 Array di oligonucleotidi ad alta densità Dimensioni dell array La fotolitografia permette la costruzione di array con alto contenuto di informazione E possibile ottenere feature di poche decine di μm 2 E possibile avere anche diversi milioni di feature (cioè migliaia di diverse probe) in uno spazio di meno di 2 cm 2 Ogni singolo probe è costituito da milioni di oligonucleotidi identici Un singolo array di meno di 2 cm 2 può consentire l analisi di circa 40,000 trascitti di geni umani
27 Array di oligonucleotidi ad alta densità Disegno degli array I geni sono rappresentati da diversi 25-meri: probe set Per ogni oligo che presenta un match perfetto (PM) con il trascritto, ne è presente uno identico con mismatch per una sola base in posizione centrale (MM) La presenza di un MM consente di stimare legami aspecifici e background locale e di sottrarli dal segnale di PM
28 Array di oligonucleotidi ad alta densità Marcatura del target
29 Array di oligonucleotidi ad alta densità Ibridizzazione A differenza degli array a DNA spottato, il sistema Affimetrix prevede l ibridizzazione di un singolo campione su ogni genechip
30 Array di oligonucleotidi ad alta densità La scannerizzazione del chip ci fornisce un immagine in cui il grado di ibridizzazione viene evidenziato da una diversa intensità di emissione nel rosso. La rilevazione dell immagine infatti è possibile grazie al legame alla biotina di un composto fluorescente (streptavidina-ficoeritrina) Rivelazione
31 Tecnologia Microarray Potenziali applicazioni La valutazione di profili di espressione genica, può trovare impiego in diverse applicazioni, quali: Caratterizzazione di marcatori diagnostici Classificazione molecolare di differenti sottotipi di malattia Identificazione di specifici target terapeutici Identificazione di vie di segnale regolate da geni chiave Drug discovery e studi tossicologici
32 Tecnologia Microarray Potenziali applicazioni La tecnologia degli array a DNA può essere utilizzata per due principali applicazioni: Profili di espressione genica da mrna: fornisce i livelli di espressione di determinati geni in un campione biologico Sequenziamento e genotipizzazione del DNA da DNA genomico: è possibile ottenere sequenze geniche e identificare polimorfismi
33 Tecnologia Microarray Potenziali applicazioni l utilizzo di microarray per la genotipizzazione ed il sequenziamento può trovare impiego in applicazioni, quali: Individuazione di mutazioni e riarrangiamenti genomici Ricerca di polimorfismi Ricerca di geni virali o batterici
34 Microarray per genotipizzazione e sequenziamento
35 Array per genotipizzazione e sequenziamento ANALISI IBRIDAZIONE SONDE TIPO ARRAY CNV (Copy Number Variation) SNP Sequenziam. di specifiche regioni Ricerca di specifiche mutazioni Competitiva Non compet. Non compet. Non compet. Non compet. Oligonucleotidi Cloni spottati Oligonucleotidi CGH-array GeneChip GeneChip (custom) Vari (es. APEX)
36 GeneChip Genotipizzazione e sequenziamento Ciascuna sequenza da analizzare è rappresentata da oligonucleotidi, ciascuno costituito da basi contenente tutte le possibili varianti di sequenza (sostituzioni, delezioni, inserzioni) La presenza di un polimorfismo è dato dalla presenza/assenza di segnale fluorescente in corrispondenza della sonda corrispondente
37 Analisi gain of hybridization signal Frammento (doppio filamento) da analizzare di lunghezza N Per coprire tutte le possibili sostituzioni su entrambi i filamenti saranno necessarie 8N sonde di nt Per coprire tutte le possibili delezioni su entrambi i filamenti saranno necessarie 2N sonde di nt Per coprire tutte le possibili inserzioni di lunghezza X su entrambi i filamenti saranno necessarie 2(4X)N sonde di nt
38 Analisi gain of hybridization signal Per analizzare una sequenza di 10 kb sono necessarie: 80,000 sonde per tutte le possibili sostituzioni Ulteriori 100,000 sonde per tutte le possibili delezioni di 1-5 bp Ulteriori 27,280,000 sonde per tutte le possibili inserzioni di 1-5 bp Limite del sistema: possono essere analizzate soprattutto delezioni e/o inserzioni di singoli nucleotidi.
39 Analisi loss of hybridization signal Idealmente ciascuna posizione nucleotidica bersaglio contribuisce nell ibridazione per un set di N sonde che si sovrappongono per N basi perfect match in unoligonucleotide array. In questo esempio il cambiamento nucleotidico in posizione A coinvolge nell ibridazione 25 sonde di 25bp. Sonde: oligonucleotidi perfect match
40 Analisi loss of hybridization signal SNP in omozigosi : completa perdita di segnale di ibridazione in corrispondenza delle sonde perfect match situate nell intorno dello SNP. SNP in eterozigosi: 50% perdita di segnale di ibridazione in corrispondenza delle sonde perfect match situate nell intorno dello SNP. Per coprire tutte le possibili sostituzioni su entrambi i filamenti (lunghezza N) saranno necessarie almeno 2N sonde di nt. Es. E necessario un array di 11,000 oligonucleotidi per analizzare il gene BRCA1 (5.5 kb)
41 Loss of signal Riferimento Omozigote WT A T A T PM Signal: 100% Omozigote MUT G C G C PM Signal: 0%
42 Loss of signal Riferimento Omozigote WT A T A T PM Signal: 100% Eterozigote MUT A T G C PM Signal: 50%
43 Gain of signal Omozigote WT A T A T (PM) A G T C Si No No No
44 Gain of signal Eterozigote MUT A T G C (PM) A G T C Si Si No No
45 Gain of signal Omozigote MUT G C G C (PM) A G T C Si Si No No
46 Riferimento Omozigote WT A T A T Gain + Loss (PM) A 100% G 0% T 0% C 0% Eterozigote MUT A T G C (PM) A G T C 50% 50% 0% 0% Omozigote MUT G C G C (PM) A 0% G 100% T 0% C 0%
47 Combinazione gain + loss of hybridization signal per l analisi del gene BRCA1
48 GeneChip specifico per SNPs di p53 (Combinazione gain + loss of hybridization signal )
49 Classificazione mutazioni p53 identificate mediante sequenziamento e mediante oligonucleotide microarray Mutation type Sequencing Microarray Total Single-bp substitutions Missense Nonsense Splice junction Deletion and/or insertion Single-bp deletion Multiple bp deletion Deletion and insertion Total Accuratezza singole sostituzioni: 94% con i microarray 87% con sequenziamento classico Accuratezza delezioni/inserzioni: 33% con i microarray 87% con sequenziamento classico
50 Sonde per mutazioni di CYP2D6 e CYP2C19 Genotipizzazione simultanea di 18 mutazioni conosciute che definiscono 10 alleli del gene CYP2D6 e 2 alleli del gene CYP2C19
51 Tecnologia Microarray Applicazioni cliniche Oncologia: in particolare nel settore delle leucemie questa tecnologia ha trovato buone possibilità di utilizzo in ambito diagnostico. La tecnologia Microarray è applicabile alla classificazione molecolare di leucemie e di diversi tipi di cancro. Sulla base dei differenti profili di espressione genica, si prospetta la possibilità di utilizzare gli array come strumento sia diagnostico (identificando anche nuovi sottotipi di malattia), sia prognostico (predizione risposta a terapie, rischi di recidiva, ecc ). La tecnologia microarray può trovare impiego anche nell identificazione di trattamenti individuali in accordo con il profilo di espressione genica del paziente e di nuovi target malattia-specifici per scopi di drug discovery.
52 Tecnologia Microarray Applicazioni cliniche Microbiologia: sono stati sviluppati array in grado di rilevare sequenze geniche di diversi tipi di patogeni. Array a oligonucleotidi, contenenti sequenze virali altamente conservate all interno della stessa famiglia consentono di rilevare e classificare nuove varianti virali. E proponibile l impiego di questa nuova tecnologia in diagnostica e nell identificazione di malattie di eziologia sconosciuta. Diversi studi hanno portato alla realizzazione di vari tipi di chip, il cui impiego può spaziare dalla diagnosi di infezioni batteriche (con rilevazione di geni per la resistenza ad antibiotici), agli studi sulle relazioni filogenetiche tra diversi microorganismi.
53 Tecnologia Microarray Applicazioni cliniche Farmacogenomica: studio della variazione individuale di risposta ai farmaci sulla base del corredo genetico. Alcuni polimorfismi genetici causano risposte differenti nei confronti di determinati farmaci. La tecnologia microarray può trovare impiego nello sviluppo di farmaci paziente-specifici. Microarray di SNP sono stati recentemente utilizzati anche per valutare il chimerismo dopo trapianti allogenici di cellule staminali
Biosensori 3: Biosensori a DNA e Bio- Gene Chip
 Biosensori 3: Biosensori a DNA e Bio- Gene Chip [email protected] (materiale parzialmente tratto da: http://nestor.med.unibo.it/shared_files/seminari/sartini_21-10-2004.ppt) Biosensori ad affinità basati
Biosensori 3: Biosensori a DNA e Bio- Gene Chip [email protected] (materiale parzialmente tratto da: http://nestor.med.unibo.it/shared_files/seminari/sartini_21-10-2004.ppt) Biosensori ad affinità basati
Differenti tipi di PCR
 Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Differenti tipi di PCR Colony PCR Nested PCR Multiplex PCR AFLP PCR Hot start PCR In situ PCR Inverse PCR Asymmetric PCR Long PCR Long accurate PCR Reverse transcriptase PCR Allele Specific PCR Real time
Metodologie citogenetiche. Metodologie molecolari. Formulare la domanda Utilizzare la metodica appropriata
 In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
In base al potere di risoluzione della tecnica Metodologie citogenetiche Metodologie molecolari Formulare la domanda Utilizzare la metodica appropriata 1 DNA RNA PROTEINE DNA Cromosomi (cariotipo, FISH,
Cenni storici. Tale tecnica trova largo impiego in tutte le aree di ricerca biologica:
 DNA Microarray: griglia di DNA costruita artificialmente, in cui ogni elemento della griglia riconosce una specifica sequenza target di RNA o cdna. Tale tecnica trova largo impiego in tutte le aree di
DNA Microarray: griglia di DNA costruita artificialmente, in cui ogni elemento della griglia riconosce una specifica sequenza target di RNA o cdna. Tale tecnica trova largo impiego in tutte le aree di
Southern blot. !Per. determinare la presenza o assenza di specifici frammenti genici. !Studio. della struttura e dell organizzazione di un gene
 Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
Southern blot!per determinare la presenza o assenza di di specifici frammenti genici!studio della struttura e dell organizzazione di di un gene Southern blot Procedura Isolare DNA genomico Digerirlo con
GENOMA. Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine CONTENUTO FUNZIONE. Progetti genoma in centinaia di organismi
 GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
GENOMA EVOLUZIONE CONTENUTO FUNZIONE STRUTTURA Analisi di sequenze -- Analisi di espressione -- Funzione delle proteine Progetti genoma in centinaia di organismi Importante la sintenia tra i genomi The
Biotecnologie. Screening delle genoteche con le sonde geniche
 Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
Biotecnologie Screening delle genoteche con le sonde geniche Giancarlo Dessì http://www.giand.it Licenza Creative Commons BY-NC-SA (BY: attribuzione, NC: uso non commerciale, SA: condividi allo stesso
I PROGETTI OMICI. Studiano gli elementi biologici nel loro insieme TRASCRITTOMICA PROTEOMICA
 1 I PROGETTI OMICI Studiano gli elementi biologici nel loro insieme TRASCRITTOMICA PROTEOMICA Studia il contenuto in mrna di una cellula (in differenti fasi di sviluppo, in risposta a vari stimoli o a
1 I PROGETTI OMICI Studiano gli elementi biologici nel loro insieme TRASCRITTOMICA PROTEOMICA Studia il contenuto in mrna di una cellula (in differenti fasi di sviluppo, in risposta a vari stimoli o a
SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica
 SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica Prof. Francesco Michelotti SAPIENZA Università di Roma Facoltà di Ingegneria
SAPIENZA Università di Roma Laurea magistrale in Ingegneria delle Nanotecnologie A.A. 2014-2015 Corso di Laboratorio di Biofotonica Prof. Francesco Michelotti SAPIENZA Università di Roma Facoltà di Ingegneria
Tecnologia del DNA ricombinante
 Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
Tecnologia del DNA ricombinante Scoperte rivoluzionarie che hanno permesso lo studio del genoma e della funzione dei singoli geni Implicazioni enormi nel progresso della medicina: comprensione malattie
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo
 Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Come facciamo ad isolare un gene da un organismo? Utilizziamo una libreria ovvero una collezione dei geni del genoma del cromosoma di un organismo GENOMA di alcuni organismi viventi raffigurato come libri
Vettori derivati dal fago λ: batteriofagi filamentosi M13
 Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Vettori derivati dal fago λ: batteriofagi filamentosi M13 A. Phage display: esibizione della proteina clonata in M13 ed esposta sulla superficie del batteriofago (facile screening dei ricombinanti) mediante
Le biotecnologie. Sadava et al. Biologia La scienza della vita Zanichelli editore 2010
 Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Le biotecnologie 1 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi viventi o da loro derivati.
Il progetto Genoma Umano è iniziato nel E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA.
 Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Il progetto Genoma Umano è iniziato nel 1990. E stato possibile perchè nel 1986 era stato sviluppato il sequenziamento automatizzato del DNA. Progetto internazionale finanziato da vari paesi, affidato
Genomica, proteomica, genomica strutturale, banche dati.
 Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Genomica, proteomica, genomica strutturale, banche dati. Alcune pietre miliari della biologia anno risultato 1866 Mendel scopre i geni 1944 il DNA è il materiale genetico 1951 prima sequenza di una proteina
Marcatori molecolari per l analisi genica, genetica e genomica
 Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
Marcatori molecolari per l analisi genica, genetica e genomica (RFLP e PCR-derivati, inclusi SSR e SNP) DNA fingerprinting DNA genotyping DNA haplotyping cp/mtdna barcoding MG/QTL mapping MAS breeding
LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI
 CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CONCETTI DI BASE LA BIOLOGIA MOLECOLARE E UNA BRANCA DELLA BIOLOGIA CHE STUDIA LE BASI MOLECOLARI DELLE FUNZIONI BIOLOGICHE, PONENDO UNA PARTICOLARE ATTENZIONE A QUEI PROCESSI CHE COINVOLGONO GLI ACIDI
CORSO INTEGRATO DI GENETICA a.a. 2011-2012. Genetica molecolare in medicina: Analisi di Mutazioni. Cristina Bombieri 7 dicembre 2011
 CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
CORSO INTEGRATO DI GENETICA a.a. 2011-2012 Genetica molecolare in medicina: Analisi di Mutazioni Cristina Bombieri 7 dicembre 2011 GENETICA MOLECOLARE IN MEDICINA SVILUPPI SCIENTIFICI Mappatura gene Identificazione
La variabilità genetica: tipi e metodi per studiarla e misurarla
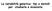 La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
La variabilità genetica: tipi e metodi per studiarla e misurarla La variabilità genetica (V.G.) 1. V.G. somatica 2. V.G. gametica intraindividuo 3. V.G. gametica intrapopolazione (polimorfismo) 4. V.G.
scaricato da www.sunhope.it LA TECNOLOGIA DEI MICROARRAYS
 LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale Analisi dell esoma e la medicina predittiva Domenico Coviello Direttore Medico
Analisi dei dati di espressione genica in esperimenti realizzati mediante microarray. Erika Melissari
 Analisi dei dati di espressione genica in esperimenti realizzati mediante microarray Erika Melissari ESPRESSIONE GENICA E un processo molto complesso e finemente regolato che permette ad una cellula di
Analisi dei dati di espressione genica in esperimenti realizzati mediante microarray Erika Melissari ESPRESSIONE GENICA E un processo molto complesso e finemente regolato che permette ad una cellula di
Marcatori molecolari
 Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
Marcatori molecolari Caratteristiche e applicazioni Luca Gianfranceschi e Rosanna Marino 1 I marcatori molecolari Strumento per l analisi genetica Strumento Molecolari Analisi genetica Marcatori non oggetto
La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse in Real Time PCR
 La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
La diagnostica molecolare delle malattie sessualmente trasmesse (non HIV): nuovi percorsi di appropriatezza analitica e clinica. La tecnologia Seegene per la diagnosi delle Malattie Sessualmente Trasmesse
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
A COSA SERVE il CLONAGGIO del DNA?? COME FUNZIONA il CLONAGGIO del DNA? COME FUNZIONA il CLONAGGIO del DNA?
 A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
A COSA SERVE il CLONAGGIO del DNA?? CREARE COPIE IDENTICHE (= CLONI) DI UN FRAMMENTO DI DNA DIFFERENZE con la PCR: è una reazione in vivo sfrutta l apparato di replicazione del DNA della cellula ospite
Array-CGH(Comparative Genomic Hybridisation) e diagnosi prenatale. Dalla citogenetica convenzionale alla citogenetica molecolare!
 Array-CGH(Comparative Genomic Hybridisation) e diagnosi prenatale Dalla citogenetica convenzionale alla citogenetica molecolare! CARIOTIPO STANDARD Identifica anomalie strutturali bilanciate e sbilanciate
Array-CGH(Comparative Genomic Hybridisation) e diagnosi prenatale Dalla citogenetica convenzionale alla citogenetica molecolare! CARIOTIPO STANDARD Identifica anomalie strutturali bilanciate e sbilanciate
Modificazione del 1 gennaio 2017
 Ornanza del DFI del 29 settembre 1995 sulle prestazioni dell assicurazione obbligatoria delle cure meco-sanitarie (Ornanza sulle prestazioni, OPre, allegato 3) RS 832.112.31 Moficazione del 1 gennaio 2017
Ornanza del DFI del 29 settembre 1995 sulle prestazioni dell assicurazione obbligatoria delle cure meco-sanitarie (Ornanza sulle prestazioni, OPre, allegato 3) RS 832.112.31 Moficazione del 1 gennaio 2017
La mappatura dei geni umani. SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione
 La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
La mappatura dei geni umani SCOPO conoscere la localizzazione dei geni per identificarne la struttura e la funzione Un grande impulso alla costruzione di mappe genetiche è stato dato da le tecniche della
Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA
 6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
6746 BROWN Iniziali I-XIV 13-04-2007 11:57 Pagina VII Indice Parte I I principi fondamentali della clonazione dei geni e dell analisi del DNA Capitolo 1 L importanza della clonazione dei geni e dell analisi
Sequenziamento del DNA: strutture dei nucleotidi
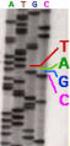 Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
Sequenziamento del DNA: strutture dei nucleotidi il metodo di Sanger 2-3 DIDEOSSINUCLEOTIDI Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI
 DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
DESCRIZIONE DEI DIFFERENTI METODI MOLECOLARI DISPONIBILI PER LA RILEVAZIONE DI HPV E L L IDENTIFICAZIONE DEI GENOTIPI Relatore:Paola Alberizzi-TLBM Responsabile:Dr.ssa Barbara Dal Bello INTRODUZIONE L
ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe
 ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe Z-2111-200 20 (0.2 ml) Per la rilevazione della translocazione t(9;22)(q34;q11)mediante ibridazione in situ fluorescente (FISH).... Dispositivo per uso
ZytoLight SPEC BCR/ABL Dual Color Dual Fusion Probe Z-2111-200 20 (0.2 ml) Per la rilevazione della translocazione t(9;22)(q34;q11)mediante ibridazione in situ fluorescente (FISH).... Dispositivo per uso
LA REAZIONE A CATENA DELLA POLIMERASI PCR
 LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
LA REAZIONE A CATENA DELLA POLIMERASI PCR INTRODUZIONE Tecnica ideata da Mullis e collaboratori nel 1984 La possibilita' di disporre di quantità virtualmente illimitate di un determinato frammento di DNA,
Importanza della genetica dei microrganismi
 Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
Importanza della genetica dei microrganismi 1.I microrganismi rappresentano un mezzo essenziale per comprendere la genetica di tutti gli organismi. 2.Vengono usati per isolare e duplicare specifici geni
NimbleGen ISCA Plus. la nuova soluzione Roche per la citogenetica. Enrico Bartolini, Donatella Guardione Roma, 18 aprile 2012
 NimbleGen ISCA Plus la nuova soluzione Roche per la citogenetica Enrico Bartolini, Donatella Guardione Roma, 18 aprile 2012 Il Gruppo Roche Informazioni principali Fondato nel 1896 a Basilea Le famiglie
NimbleGen ISCA Plus la nuova soluzione Roche per la citogenetica Enrico Bartolini, Donatella Guardione Roma, 18 aprile 2012 Il Gruppo Roche Informazioni principali Fondato nel 1896 a Basilea Le famiglie
La tecnologia dei microarray
 La tecnologia dei microarray Introduzione per l analisi dell espressione genica: Affymetrix GeneChip spotted arrays L analisi dei dati Campi di applicazione dei microarray Introduzione I progetti di sequenziamento
La tecnologia dei microarray Introduzione per l analisi dell espressione genica: Affymetrix GeneChip spotted arrays L analisi dei dati Campi di applicazione dei microarray Introduzione I progetti di sequenziamento
Lezioni di biotecnologie
 Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
Lezioni di biotecnologie Lezione 1 Il clonaggio 2 Cosa sono le biotecnologie? Le biotecnologie sono tutte quelle tecniche utilizzate (fin dall antichità) per produrre sostanze specifiche a partire da organismi
CORSO INTEGRATO DI GENETICA. a.a /11/2010. Analisi di linkage Analisi di mutazioni
 CORSO INTEGRATO DI GENETICA a.a. 2010-2011 04/11/2010 Lezioni 27_28 Analisi di linkage Analisi di mutazioni Dott.ssa Elisabetta Trabetti LINKAGE Geni in loci vicini sullo stesso cromosoma - concatenati
CORSO INTEGRATO DI GENETICA a.a. 2010-2011 04/11/2010 Lezioni 27_28 Analisi di linkage Analisi di mutazioni Dott.ssa Elisabetta Trabetti LINKAGE Geni in loci vicini sullo stesso cromosoma - concatenati
Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Nested
Prof.ssa Tiziana Pepe Evoluzioni della tecnica PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Nested
Diagnosi genetiche molecolari
 Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Polymerase chain reaction - PCR. Biotecnologie applicate all ispezione degli alimenti di origine animale
 Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Metodologia
Prof.ssa Tiziana Pepe Polymerase chain reaction - PCR Biotecnologie applicate all ispezione degli alimenti di origine animale Dip. di Medicina Veterinaria e Produzioni animali [email protected] Metodologia
ddpcr per la diagnosi prenatale di aneuploidie
 ddpcr per la diagnosi prenatale di aneuploidie Rilevazione della presenza di trisomia 21 per la diagnosi prenatale Il sangue materno contiene circa il 10% di DNA fetale Il grado di sovra-espressione dipende
ddpcr per la diagnosi prenatale di aneuploidie Rilevazione della presenza di trisomia 21 per la diagnosi prenatale Il sangue materno contiene circa il 10% di DNA fetale Il grado di sovra-espressione dipende
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Cenni di genetica di popolazione delle specie native
 Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Cenni di genetica di popolazione delle specie native prof. Pallavicini dott. Victoria Bertucci 27 gen 2012 1 Biodiversità L'espressione italiana è un calco linguistico derivante dal termine inglese biodiversity.
Retrovirus e DNA ricombinante. Lezioni d'autore di Paola Vinesi
 Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Retrovirus e DNA ricombinante Lezioni d'autore di Paola Vinesi I RETROVIRUS (I) Come tutti i virus, i retrovirus sono parassiti endocellulari obbligati che infettano le cellule rilasciando in esse il proprio
Il Sequenziamento Tecniche e Strategie
 Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
Il Sequenziamento Tecniche e Strategie Metodi per il sequenziamento del DNA Metodo della degradazione chimica (Maxam & Gilbert, 1977) La sequenza di una molecola di ds DNA viene determinata mediante trattamento
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
Ringraziamo per l elaborazione dei modelli di refertazione dello schema di Fibrosi Cistica. Ospedali Galliera, Laboratorio di Genetica, Genova
 Roma, 15/02/2011 Viale Regina Elena, 299 00161 Roma Tel: 0649902805 Fax: 0649902292 Email: [email protected] ; [email protected] Gentili colleghi, in fase di valutazione dei risultati delle analisi eseguite
Roma, 15/02/2011 Viale Regina Elena, 299 00161 Roma Tel: 0649902805 Fax: 0649902292 Email: [email protected] ; [email protected] Gentili colleghi, in fase di valutazione dei risultati delle analisi eseguite
I RECETTORI: Caratterizzazione e isolamento
 I RECETTORI: Caratterizzazione e isolamento Andrea Ferrigno Funzione dei recettori di membrana Secrezione dell ormone nel sangue L ormone è trasportato attraverso il circolo sanguigno L ormone raggiunge
I RECETTORI: Caratterizzazione e isolamento Andrea Ferrigno Funzione dei recettori di membrana Secrezione dell ormone nel sangue L ormone è trasportato attraverso il circolo sanguigno L ormone raggiunge
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI
 LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
LA TECNOLOGIA DEL DNA RICOMBINANTE RICHIEDE L USO DI ENZIMI SPECIFICI La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri
La tecnologia dei microarray
 La tecnologia dei microarray I microarray I progetti di sequenziamento (progetti permesso di identificare migliaia di geni genoma) ) hanno Migliaia di geni (ed i loro prodotti, le proteine) operano in
La tecnologia dei microarray I microarray I progetti di sequenziamento (progetti permesso di identificare migliaia di geni genoma) ) hanno Migliaia di geni (ed i loro prodotti, le proteine) operano in
Tecnologia del DNA e la genomica
 Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Tecnologia del DNA e la genomica DNA ricombinante La tecnologia del DNA ricombinante permette di unire in laboratorio il DNA di organismi diversi Permette di manipolare il DNA di un organismo allo scopo
Sommario. Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni
 Sommario Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni SNP: definizioni Polimorfismo al singolo nucleotide, in genere si intende la sostituzione
Sommario Diversità genetica a livello di sequenza Trovare gli SNP Genotipizzare gli SNP Principali applicazioni SNP: definizioni Polimorfismo al singolo nucleotide, in genere si intende la sostituzione
Ibridizzazione in situ
 ANALISI DELL RNA Northen blot E un metodo simile a quello di traferimento e di ibridizzazione del DNA (Southern blot) e si usa per sondare molecole di RNA. Gli mrna sono molecole brevi, in genere meno
ANALISI DELL RNA Northen blot E un metodo simile a quello di traferimento e di ibridizzazione del DNA (Southern blot) e si usa per sondare molecole di RNA. Gli mrna sono molecole brevi, in genere meno
frequente caratterizzato da fastidio/dolore addominale e alterazioni dell alvo (costipazione e/o diarrea). Questa sindrome si manifesta con un elevata
 RIASSUNTO I Introduzione La sindrome dell intestino irritabile (IBS) è un disturbo funzionale molto frequente caratterizzato da fastidio/dolore addominale e alterazioni dell alvo (costipazione e/o diarrea).
RIASSUNTO I Introduzione La sindrome dell intestino irritabile (IBS) è un disturbo funzionale molto frequente caratterizzato da fastidio/dolore addominale e alterazioni dell alvo (costipazione e/o diarrea).
DOGMA CENTRALE DELLA BIOLOGIA. Secondo il dogma centrale della biologia, il DNA dirige la. sintesi del RNA che a sua volta guida la sintesi delle
 DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
DOGMA CENTRALE DELLA BIOLOGIA Secondo il dogma centrale della biologia, il DNA dirige la sintesi del RNA che a sua volta guida la sintesi delle proteine. Tuttavia il flusso unidirezionale di informazioni
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Fondamenti di Chimica Farmaceutica. La scoperta dei farmaci
 Fondamenti di Chimica Farmaceutica La scoperta dei farmaci La scoperta di un farmaco è un processo che ha lo scopo di individuare un composto terapeuticamente utile per curare una malattia. Il punto di
Fondamenti di Chimica Farmaceutica La scoperta dei farmaci La scoperta di un farmaco è un processo che ha lo scopo di individuare un composto terapeuticamente utile per curare una malattia. Il punto di
 Cromosoma del batterio donatore Trasferimento della ORF nel vettore di espressione PRODUZIONE DELLA PROTEINA RICOMBINANTE Abbondante e facilmente purificabile Contiene una ORF continua!! 5 NT non tradotto
Cromosoma del batterio donatore Trasferimento della ORF nel vettore di espressione PRODUZIONE DELLA PROTEINA RICOMBINANTE Abbondante e facilmente purificabile Contiene una ORF continua!! 5 NT non tradotto
Analisi degli alimenti tramite PCR Real-Time. Introduzione ad una tecnologia all'avanguardia
 Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Analisi degli alimenti tramite PCR Real-Time Introduzione ad una tecnologia all'avanguardia Obiettivi Questa presentazione tratterà i seguenti argomenti: Quali sono le applicazioni della PCR Real-time?
Analisi bioinformatica di esperimenti di Esoma Intero in trio
 Analisi bioinformatica di esperimenti di Esoma Intero in trio L analisi dell esoma intero applicata ai trio (genitori e figlio/figlia affetto) è un approccio molto potente al fine di identificare mutazioni
Analisi bioinformatica di esperimenti di Esoma Intero in trio L analisi dell esoma intero applicata ai trio (genitori e figlio/figlia affetto) è un approccio molto potente al fine di identificare mutazioni
04_biochimica. MFN0366-A1 (I. Perroteau) - Tecniche biochimiche-molecolari: Western blot
 1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
1 2 1 - Gel di elettroforesi Trasferimento (blotting) + Membrana per western blot 3 4 2 Anticorpo primario ---> anticorpo secondario coniugato all enzima ---> reazione di biolimunescenza ---> luce che
DIPARTIMENTO DELL INNOVAZIONE DIREZIONE GENERALE DELLA RICERCA SCIENTIFICA E TECNOLOGICA UFFICIO IV DELL EX MINISTERO DELLA SALUTE ***
 DIPARTIMENTO DELL INNOVAZIONE DIREZIONE GENERALE DELLA RICERCA SCIENTIFICA E TECNOLOGICA UFFICIO IV DELL EX MINISTERO DELLA SALUTE *** LE BIOTECNOLOGIE CHE COSA SONO LE BIOTECNOLOGIE? Le biotecnologie
DIPARTIMENTO DELL INNOVAZIONE DIREZIONE GENERALE DELLA RICERCA SCIENTIFICA E TECNOLOGICA UFFICIO IV DELL EX MINISTERO DELLA SALUTE *** LE BIOTECNOLOGIE CHE COSA SONO LE BIOTECNOLOGIE? Le biotecnologie
Frontiere della Biologia Molecolare
 Prof. Giorgio DIECI Dipartimento di Bioscienze Università degli Studi di Parma Frontiere della Biologia Molecolare Milano, 4 marzo 2016 Fotografia al microscopio elettronico di una plasmacellula NUCLEO
Prof. Giorgio DIECI Dipartimento di Bioscienze Università degli Studi di Parma Frontiere della Biologia Molecolare Milano, 4 marzo 2016 Fotografia al microscopio elettronico di una plasmacellula NUCLEO
La Tecnologia dei Microarray. Tor Vergata Aprile 2011 ([email protected])
 La Tecnologia dei Microarray Tor Vergata Aprile 2011 ([email protected]) SH Friend and RB Stoughton, The Magic of Microarray, Scientific American, February. 2002, pp. 44-49 Vogliamo sapere velocemente
La Tecnologia dei Microarray Tor Vergata Aprile 2011 ([email protected]) SH Friend and RB Stoughton, The Magic of Microarray, Scientific American, February. 2002, pp. 44-49 Vogliamo sapere velocemente
Ricevimento Studenti: Lunedì previa prenotazione. Cenci lab
 Cenci lab Giovanni Cenci Dip.to Biologia e Biotecnologie C. Darwin Sezione Genetica Piano 2 -Citofono 3/4 0649912-655 (office) 0649912-843 (lab) [email protected] Ricevimento Studenti: Lunedì
Cenci lab Giovanni Cenci Dip.to Biologia e Biotecnologie C. Darwin Sezione Genetica Piano 2 -Citofono 3/4 0649912-655 (office) 0649912-843 (lab) [email protected] Ricevimento Studenti: Lunedì
U.O. Emato-oncologia Pediatrica, I.R.C.S.S Burlo Garofolo, Trieste Dr. Marco RABUSIN
 TITOLO Studio prospettico IRCSS Burlo Garofolo di Trieste S V I L U P P O D I S T R AT E G I E FA R M A C O L O G I C H E P E R L A PERSONALIZZAZIONE DEL TRATTAMENTO DELLA LEUCEMIA LINFOBLASTICA ACUTA
TITOLO Studio prospettico IRCSS Burlo Garofolo di Trieste S V I L U P P O D I S T R AT E G I E FA R M A C O L O G I C H E P E R L A PERSONALIZZAZIONE DEL TRATTAMENTO DELLA LEUCEMIA LINFOBLASTICA ACUTA
Mutazioni genetiche 2
 Mutazioni genetiche 2 Cosa sono le mutazioni? Le proteine sono in grado di svolgere la loro funzione solo se la loro sequenza amminoacidica è quella corretta. In caso contrario si possono generare delle
Mutazioni genetiche 2 Cosa sono le mutazioni? Le proteine sono in grado di svolgere la loro funzione solo se la loro sequenza amminoacidica è quella corretta. In caso contrario si possono generare delle
Lezione 1 secondo semestre mercoledi 3 Marzo
 Lezione 1 secondo semestre mercoledi 3 Marzo Prof: Marco Tripodi [email protected] BIOLOGIA e GENETICA a.a.2012-13 Sezione di Genetica Molecolare La cosa più importante di tutto il corso Capire,
Lezione 1 secondo semestre mercoledi 3 Marzo Prof: Marco Tripodi [email protected] BIOLOGIA e GENETICA a.a.2012-13 Sezione di Genetica Molecolare La cosa più importante di tutto il corso Capire,
La farmacogeneticanella gestione del paziente con tumore colorettale, l esperienza traslazionaleal CRO di Aviano
 SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in Italia
 Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in talia Progetto ricerca corrente MSRCTE0212 Patrizia Centorame Laboratorio Nazionale
Confronto dei metodi di tipizzazione molecolare per la caratterizzazione di ceppi di Listeria monocytogenes circolanti in talia Progetto ricerca corrente MSRCTE0212 Patrizia Centorame Laboratorio Nazionale
DELIBERAZIONE N X / 2512 Seduta del 17/10/2014
 DELIBERAZIONE N X / 2512 Seduta del 17/10/2014 Presidente ROBERTO MARONI Assessori regionali MARIO MANTOVANI Vice Presidente VALENTINA APREA VIVIANA BECCALOSSI SIMONA BORDONALI PAOLA BULBARELLI MARIA CRISTINA
DELIBERAZIONE N X / 2512 Seduta del 17/10/2014 Presidente ROBERTO MARONI Assessori regionali MARIO MANTOVANI Vice Presidente VALENTINA APREA VIVIANA BECCALOSSI SIMONA BORDONALI PAOLA BULBARELLI MARIA CRISTINA
ALLELI, forme alternative di un gene. Alleli, forme alternative di un gene. Alleli, forme alternative di un gene
 ALLELI, forme alternative di un gene Per ogni gene di un genoma possono esistere, in una popolazione di individui, una o più varianti. LE DIVERSE FORME ALTERNATIVE DI UNO STESSO GENE SI CHIAMANO ALLELI
ALLELI, forme alternative di un gene Per ogni gene di un genoma possono esistere, in una popolazione di individui, una o più varianti. LE DIVERSE FORME ALTERNATIVE DI UNO STESSO GENE SI CHIAMANO ALLELI
Metodi dell ecologia microbica
 Metodi dell ecologia microbica -Biodiversità: isolamento identificazione e quantizzazione dei microrganismi nei diversi habitat; -Attività microbica: misura di ciò che i microrganismi fanno nel loro ambiente
Metodi dell ecologia microbica -Biodiversità: isolamento identificazione e quantizzazione dei microrganismi nei diversi habitat; -Attività microbica: misura di ciò che i microrganismi fanno nel loro ambiente
LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara
 LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara Molti sono i virus che possono causare epatiti acute e
LE TECNICHE MOLECOLARI NELLA DIAGNOSI E MONITORAGGIO DELLE INFEZIONI DA HCV E HBV Paolo Ravanini Lab. di Microbiologia Az.Osp. Maggiore di Novara Molti sono i virus che possono causare epatiti acute e
METODI DI MARCATURA DEGLI ACIDI NUCLEICI
 METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia. Numero di gara
 Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
Procedura negoziata per la fornitura di diagnostici per Laboratorio di Genetica Molecolare settore Oncoematologia Numero di gara 6581966 CAPITOLATO TECNICO Item Voce Descrizione Quantità/anno 1.1 Sistema
Corso di Genetica -Lezione 12- Cenci
 Corso di Genetica -Lezione 12- Cenci Il codice genetico: Come triplette dei quattro nucleotidi specificano 20 aminoacidi, rendendo possibile la traduzione dell informazione da catena nucleotidica a sequenza
Corso di Genetica -Lezione 12- Cenci Il codice genetico: Come triplette dei quattro nucleotidi specificano 20 aminoacidi, rendendo possibile la traduzione dell informazione da catena nucleotidica a sequenza
Come disarmare i tumori
 Come disarmare i tumori Delia Mezzanzanica Unità di Terapie Molecolari Dipartimento di Oncologica Sperimentale e Medicina Molecolare Fondazione IRCCS Istituto Nazionale dei Tumori Il tumore ovarico è caratterizzato
Come disarmare i tumori Delia Mezzanzanica Unità di Terapie Molecolari Dipartimento di Oncologica Sperimentale e Medicina Molecolare Fondazione IRCCS Istituto Nazionale dei Tumori Il tumore ovarico è caratterizzato
FEBBRE MEDITERRANEA FAMILIARE: CENNI DI GENETICA
 DAVIDE MARTORANA FEBBRE MEDITERRANEA FAMILIARE: CENNI DI GENETICA PARMA, 23 OTTOBRE 2005 PROBLEMI DELL INQUADRAMENTO DIAGNOSTICO CARATTERISTICA RICORRENTE: EPISODI PERITONITICI ~70% DEI PAZIENTI SUBISCE
DAVIDE MARTORANA FEBBRE MEDITERRANEA FAMILIARE: CENNI DI GENETICA PARMA, 23 OTTOBRE 2005 PROBLEMI DELL INQUADRAMENTO DIAGNOSTICO CARATTERISTICA RICORRENTE: EPISODI PERITONITICI ~70% DEI PAZIENTI SUBISCE
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8
 Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
Strumenti della Genetica Molecolare Umana (2) Capitoli 6-7-8 BAC, Bacterial Artificial Chromosome Caratteristiche: 1. Lungo 7-8kb 2. Resistenti al cloramfenicolo (marcatore di trasformazione) 3. puc-link
FORMAZIONE DEL LEGAME PEPTIDICO
 AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
AMINOACIDI FORMAZIONE DEL LEGAME PEPTIDICO SEQUENZA AMINOACIDICA DELL INSULINA STRUTTURA SECONDARIA DELLE PROTEINE STRUTTURA TERZIARIA DELLE PROTEINE STRUTTURA QUATERNARIA DELLE PROTEINE Definizione Processi
Cosa sono i virus? Cosa sono i virus? Cosa sono i virus? I virus sono virus. dal punto di vista della cellula e dell organismo infettato
 I virus sono virus Cosa sono i virus? etimologia: veleno uso arcaico del termine: germe patogeno uso moderno del termine: riservato ai virus, e cioè Cosa sono i virus? dal punto di vista della cellula
I virus sono virus Cosa sono i virus? etimologia: veleno uso arcaico del termine: germe patogeno uso moderno del termine: riservato ai virus, e cioè Cosa sono i virus? dal punto di vista della cellula
Microbiologia clinica
 Microbiologia clinica Docente : Prof. Ubaldo Scardellato Testo: MICROBIOLOGIA CLINICA Eudes Lanciotti Cea ed. 2001 Temi cardine del programma 1. Lo sviluppo della microbiologia come scienza. 2. La natura
Microbiologia clinica Docente : Prof. Ubaldo Scardellato Testo: MICROBIOLOGIA CLINICA Eudes Lanciotti Cea ed. 2001 Temi cardine del programma 1. Lo sviluppo della microbiologia come scienza. 2. La natura
