ANALISI DI MUTAZIONI PUNTIFORMI NON NOTE ANALISI DI SEQUENZA.
|
|
|
- Floriano Ambrogio Clemente
- 8 anni fa
- Visualizzazioni
Transcript
1 ANALISI DI MUTAZIONI PUNTIFORMI NON NOTE Margherita Vinciguerra U.O. Ematologia II Laboratorio per lo Studio e la Diagnosi Molecolare Prenatale di Talassemia A.O. V. Cervello, Palermo. Analisi di sequenza Single Strand Conformation Polymorphisms Denaturant Gradient Gel Electrophoresis Temporal Temperature Gradient Gel Electrophoresis Denaturant High Performance Liquid Cromatography Microarray Real time ANALISI DI SEQUENZA. Sono stati sviluppati diversi metodi per determinare la sequenza di un frammento di DNA. Il metodo di Maxam-Gilbert si avvale di specifici reagenti chimici per modificare e tagliare la catena di DNA a livello di nucleotidi specifici; è, però, scarsamente automatizzabile. Il metodo dei didesossi o di Sanger, invece, si basa sulla sintesi enzimatica di una nuova catena di DNA su un elica stampo, utilizzando, oltre ai normali d-ntps, analoghi nucleotidi didesossi marcati con fluorocromi diversi: l assenza di un 3 -OH impedisce la formazione di un legame fosfo-diesterico con il successivo precursore e, quindi, l incorporazione di un nucleotide didesossi ( terminatore ) porta all arresto della reazione polimerasica. I prodotti che si ottengono sono frammenti che differiscono tra loro per una sola base e che vengono separati in funzione della loro lunghezza tramite elettroforesi capillare con sequenziatore automatico; le bande sono rilevate attraverso un sofisticato sistema ottico che permette l identificazione dell esatta successione delle basi nel segmento di DNA in esame. FASI DELL ANALISI DI SEQUENZA: 1. Amplificazione del DNA (PCR) 2. Purificazione 3. Reazione di sequenza 4. Purificazione 5. Denaturazione 6. Corsa elettroforetica 7. Lettura dei dati ottenuti Prima della reazione di sequenza, il prodotto di PCR deve essere purificato per rimuovere l eccesso di primers non dd A NT dd C NT BUFFER dntp + + PRIMER + DNA + MgCl dd G NT POLIMERASI dd T NT REAZIONE DI SEQUENZA PREMIX Unico tubo CICLI
2 incorporati ed i dntps; esistono diversi metodi per la purificazione. Uno di questi fa uso di enzimi di restrizione con attività esonucleasica che tagliano il DNA a singolo filamento ma non quello a doppio filamento; la fosfatasi alcalina catalizza, poi, la defosforilazione al 5 del DNA, impedendo l estensione del filamento da parte della polimerasi. Nella reazione di sequenza, la concentrazione dei ddntps deve essere circa 1/100 di quella dei dntps, in quanto l incorporazione dei terminatori deve essere del tutto casuale e garantire, comunque, una certa sintesi di DNA; la probabilità che venga incorporato il didesossi invece del normale precursore nell appropriata miscela di reazione deve essere, dunque, molto bassa. CCompletata la reazione di sequenza, si procede con la purificazione del prodotto per rimuovere l eccesso di ddntps marcati con fluorocromi. Esistono due metodi principali: uno chimico, attraverso l utilizzo dell enzima SAP (Shrimp Alkaline Phosphatase) e l altro fisico, il quale fa uso di colonnine a base di sephadex che trattiene i nucleotidi ma non i frammenti. I prodotti di reazione purificati sono risospesi in formamide deionizzata e si procede con la denaturazione (96 per 5 ); i campioni sono così pronti per la corsa elettroforetica con un sequenziatore automatico. Quest ultimo è un evoluzione dell elettroforesi classica: in questo caso, la migrazione avviene in un capillare ad una determinata corrente. Un sofisticato sistema ottico rileva la differenza tra le lunghezze d onda emesse dai quattro fluorocromi legati ai ddntps.. ANALISI DEI DATI: Ci sono tre modi per analizzare i dati di sequenza: 1. Ispezione visiva: possibile solo se il numero dei campioni da analizzare è ridotto; in tal caso, è necessario fare molta attenzione alle mutazioni in omozigosi che sfuggono ad una semplice ispezione visiva. 2. Base calling software: programmi che automaticamente riportano differenze tra la sequenza in esame e la sequenza standard. 3. Analisi comparativa di sequenza: forniscono comparazione diretta di tracciati di sequenza mutati e wilde-type. Per una corretta interpretazione dei dati, i picchi devono essere ben definiti, con fondo scarso o nullo; la qualità dei dati deve essere buona e la linea di base bassa per evitare artefatti e picchi di mutazione meno intensi del normale. RILEVATORE (CCD CAMERA) - + PROBLEMATICHE: Linea di base alta: se l altezza dei picchi è maggiore di 4000 unità di fluorescenza è preferibile diluire e far ricorrere i campioni. 75
3 Dye-blobs: indicano la presenza di tutte e quattro le fluorescenze anche in sequenze ottime; dovuti per lo più ad artefatti della corsa elettroforetica nel capillare, scompaiono nella maggior parte dei casi se si fanno ricorrere i campioni. PCR aspecifica: si traduce in un fondo nell elettroferogramma tanto maggiore quanto maggiore è la concentrazione dell amplificato aspecifico; in tal caso è necessario ripetere l amplificato, oppure ridisegnare i primers se si trovano all interno di zone ripetute, o ancora ridiluire i primers se contaminati oppure, infine, aumentare la specificità del templato (aumentando la temperatura di annealing o riducendo la concentrazione del magnesio). Incompleta purificazione dei primers: porta alla presenza di due popolazioni di primers, una completa e l altra popolazione N-1 (dovuta alla rimozione di un nucleotide al 5 ); pertanto, è preferibile richiedere primers di sequenza purificati dopo la sintesi con dhplc. L analisi di sequenza è per eccellenza la metodica di analisi diretta per la ricerca di mutazioni note e non note. I principali vantaggi di tale tecnica sono un informazione completa su posizione e natura della mutazione, l estrema sensibilità e l estrema accuratezza; gli svantaggi sono molto ridotti e riguardano soprattutto il costo iniziale dell acquisto e dell avvio del sequenziatore, la mole di dati da analizzare ed il tempo leggermente maggiore per avere i risultati. SSCP (Single strand conformation polymorphisms). Si basa sull alterazione della conformazione del DNA a singola elica, determinata da una sostituzione nucleotidica, che causa una diversa velocità di migrazione su gel di poliacrilammide del frammento di DNA in cui è avvenuta la mutazione. Il protocollo originario faceva uso di marcatura radioattiva, ma è stata messa a punto una metodologia basata sul riconoscimento delle bande di migrazione tramite colorazione con argento. Il DNA a doppia elica (PCR) è denaturato con calore e formamide e raffreddato rapidamente per evitare la rinaturazione. Il DNA a singola elica assume conformazioni diverse: la migrazione su gel di poliacrilammide non denaturante sarà determinata dalla conformazione piuttosto che dalle dimensioni. Il pattern di conformazioni è sequenza-dipendente: anche una singola sostituzione nucleotidica altera la conformazione. SSCP è una tecnica semplice da usare e l analisi dei dati è agevole; è, però, estremamente suscettibile a piccole variazioni nelle condizioni standard. SSCP FLUORESCENTE: Si avvale di un sistema di elettroforesi capillare automatico. i primers di PCR (forward e reverse) sono marcati con fluorocromi; 76
4 si denatura a ssdna e si raffredda rapidamente per evitare la rinaturazione; si procede con elettroforesi capillare con polimero non-denaturante; un rilevatore elaborerà le fluorescenze emesse dai fluorocromi colpiti dal raggio laser e rileverà la diversa mobilità di ogni filamento marcato. I vantaggi di tale metodica sono: la possibilità di usare temperature diverse, aumentando enormemente la sensibilità; il controllo preciso della temperatura; la grande riproducibilità; la possibilità di usare un controllo interno; la normalizzazione della mobilità con più accurata identificazione anche di mutazioni che causano solo una piccola alterazione della mobilità; la possibilità di usare anche prodotti di PCR più lunghi. DGGE (Denaturing gradient gel electrophoresis) E una tecnica basata su una migrazione elettroforetica su gel di poliacrilammide con gradiente di sostanze denaturanti (formamide e urea) di frammenti amplificati di DNA, i quali riducono la loro velocità di migrazione quando raggiungono una concentrazione di denaturante sul gel che corrisponda alla loro temperatura di fusione; la T m è strettamente dipendente dalla composizione nucleotidica del DNA e, quindi, la presenza di una sostituzione di una coppia di basi in un segmento di DNA ne modifica il valore. Di conseguenza, dopo corsa elettroforetica, colorazione con bromuro di etidio e rilevazione agli UV, si osserverà per il frammento recante la mutazione un pattern elettroforetico tanto più differente da quello normale quanto più la sostituzione nucleotidica avrà alterato la T m del frammento stesso. L L L incorporazione tramite PCR di GC-clamp (sequenza di coppie di basi ricche in GC ad altissima T m ) ad un estremità del frammento di DNA in esame consente di individuare, usando un unico tipo di gradiente di denaturante, eventuali mutazioni situate in qualunque punto del frammento in esame, anche in domini ad alta T m ; la GC clamp, infatti, costituendo sempre il dominio ad alta temperatura, altera il comportamento di denaturazione del frammento cui è stata unita, rendendolo di conseguenza tutto dominio a bassa temperatura. Il soggetto eterozigote per una mutazione o polimorfismo presenta quattro bande: a) 2 bande più veloci (omoduplex) alleli normale e mutato; b) 2 bande più lente (eteroduplex) alleli ibridi. E una tecnica laboriosa, ma utile soprattutto per geni molto lunghi, in quanto permette di restringere la ricerca della mutazione a pochi frammenti. TTGE (Temporal temperature gradient electrophoresis). È una tecnica derivata dal DGGE; per ottenere la temperatura di fusione del frammento di DNA in esame, impiega un gradiente lineare e temporale di energia termica, anziché un gradiente di denaturante chimico. 77
5 DHPLC (Denaturing high-performance liquid chromatography). Impiega il meccanismo di ripartizione in fase inversa ad accoppiamento ionico (IP-RP-HPLC ) e separa e distingue le molecole di DNA in base alla loro dimensione; l accoppiamento ionico è ottenuto con l ausilio dello ione TEAA (tri-etil-ammonio-acetato), molecola con doppia funzionalità in grado di interagire sia con il campione da analizzare sia con il supporto cromatografico (colonna costituita da un copolimero di polistirene-divinilbenzene). Durante l amplificazione genica: 1) se il DNA non contiene mutazioni, nella fase di re-annealing si formano solo omoduplex; 2) nel caso in cui ci sia una mutazione in eterozigosi, oltre agli omoduplex si generano gli eteroduplex. Nel caso dell omozigote per la stessa mutazione non c è la possibilità di formare direttamente gli eteroduplex; In tal caso si raggira l ostacolo miscelando una quantità analoga di campione normale. Come risultato cromatografico si avranno due situazioni: 1) il campione senza mutazione avrà un solo picco: 2) il campione con mutazione avrà due picchi dovuti alla formazione di omoduplex e due picchi dovuti alla formazione di eteroduplex. L eluizione dei frammenti di DNA dalla colonna è ottenuta mediante un gradiente di un agente organico, l acetonitrile, che rompe quest interazione. DHPLC parzialmente denaturante è capace di identificare singole sostituzioni nucleotidiche o piccole inserzioni/delezioni in regioni amplificate di un gene; si basa sul maggior tempo di ritenzione degli homoduplex rispetto agli eteroduplex all interno di una miscela di DNA amplificato, denaturato e rinaturato. Necessita di buoni amplificati e di ottime condizioni di temperatura. Le molecole di DNA eluite possono essere rilevate attraverso gli UV o con monitoraggio fluorescente dopo il caricamento. Presenta specificità e sensibilità del 100%, ma non si presta alla tipizzazione di campioni omozigoti. È un metodo di rilevamento indiretto cui deve seguire una caratterizzazione definitiva con un metodo diretto (ARMS, sequenza). DHPLC completamente denaturante è capace di separare corte molecole (70-80 bp) di DNA a singolo filamento che differiscono per una singola base e, quindi, può essere applicata per tipizzare campioni con mutazioni/polimorfismi noti; l unica sostituzione non rilevata è C G. DHPLC è, dunque, un metodo efficace per rilevare mutazioni puntiformi del DNA, soprattutto per geni molto lunghi; è molto sensibile, si offre ad un alto grado di automazione ed ha costi relativamente bassi, fatta eccezione per l acquisto e l avvio dell apparecchio. L accuratezza e la sensibilità dipendono, però dalla temperatura; inoltre, DHPLC è un metodo di rilevamento indiretto cui deve seguire una caratterizzazione definitiva con metodo diretto (ARMS, sequenza). 78
6 MICROARRAYS. Gli array sono dei supporti solidi (vetro o plastica) sui quali vengono fissate da centinaia a migliaia di molecole di acidi MICROARRAY Isolamento degli mrna cellulari e eventuale retrotrascrizione in cdna. Marcatura con sostanze fluorescenti. Ibridazione sul DNA microarray Rilevazione del segnale. I microarrays possono misurare solo modifiche relative di espressione ma non i livelli assoluti. nucleici di sequenza nota, disposte secondo una matrice di righe e colonne ordinata e definita; il principio biochimico alla base della tecnologia è il riconoscimento e l ibridazione tra il frammento di acido nucleico fissato (detto sonda ) ed il suo complementare (detto target ) eventualmente presente nel campione in esame, seguiti poi dalla rilevazione del segnale fluorescente d ibridazione. Esistono due tecnologie per la produzione di microarray: spotting ed in situ. I target, ovvero gli acidi nucleici da ibridare alle catene di cdna ancorate al supporto solido, sono normalmente ottenuti dalla marcatura dell mrna proveniente da un dato organismo per mezzo di molecole fluorescenti. Sonde e target sono messi a contatto per fare avvenire la reazione di ibridazione e, dopo alcuni lavaggi per rimuovere i prodotti aspecifici, l array viene passato attraverso uno scanner per la misura dei segnali fluorescenti. L intensità dei pixel di ciascuna immagine è proporzionale al numero di molecole di tracciante presenti sullo spot e cioè al numero di probes che hanno ibridato le sonde ancorate al supporto; livelli diversi di fluorescenza indicano, pertanto, livelli diversi di ibridazione e, quindi, di espressione genica. I microarray possono essere impiegati sia in studi di espressione genica per analizzare il profilo trascrizionale di un campione biologico sia in studi di analisi della variabilità genetica (identificazione di mutazioni o polimorfismi nella sequenza genomica target); possono, però, misurare solo modifiche relative di espressione e non i livelli assoluti. REAL-TIME PCR. La chimica di reazione di questa tecnica prevede l uso di sonde legate a molecole fluorescenti. Le sonde ad idrolisi hanno una molecola reporter al 5 ed una molecola quencer al 3 che impedisce al reporter di emettere liberamente il segnale; quando la polimerasi sintetizza il filamento complementare al DNA stacca la sonda da quest ultimo e la taglia; in questo modo il reporter passa in soluzione aumentando l intensità della fluorescenza che sarà direttamente proporzionale alla concentrazione di amplificato specifico. 79
7 I Light Upon Extension Primers (LUX) hanno una chimica a fluorescenza che consente di rilevare il segnale senza ricorrere a sonde; la fluorescenza del reporter al 3 è assorbita quando il primer non è esteso dalla presenza di una struttura secondaria a forcina introdotta artificialmente in 5 (il quencer è assente); l estensione del primer apre la forcina e si ha l emissione della fluorescenza. Le sonde LNA sono una variante delle sonde ad idrolisi; l introduzione di un legame fosfodiesterico intramolecolare a livello dello zucchero desossiribosio irrigidisce la struttura e si lega più stabilmente alla sequenza target. Ciò rende le sonde LNA fortemente indicate nell analisi di mutazioni puntiformi, ovvero nella discriminazione allelica in multiplex. La Real-time PCR può essere condotta in "multiplex, cioè in un singolo tubo di reazione può essere rilevato più di un prodotto di PCR; ogni prodotto di PCR è associato ad un particolare fluorocromo, la cui emissione è rilevata dall amplificatore per la real-time PCR. A differenza della PCR, consente di misurare la quantità di amplificato ad ogni ciclo; necessita, però, di una precisa determinazione di una curva standard di riferimento. 80
SEQUENZIAMENTO DEL DNA
 SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
SEQUENZIAMENTO DEL DNA Il metodo di Sanger per determinare la sequenza del DNA Il metodo manuale La reazione enzimatica Elettroforesi in gel denaturante di poliacrilammide Autoradiografia Il metodo automatico
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di
 Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
Dopo aver effettuato la PCR, all interno della soluzione oltre al tratto amplificato sono presenti: primers, dntps, Taq polimerasi e tampone di reazione Inizialmente i 20 µl dell amplificato vengono
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo HUMAN GENOME PROJECT
Isolamento e purificazione di DNA e RNA. -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine)
 Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
Isolamento e purificazione di DNA e RNA -Rompere la membrana cellulare -Separare gli acidi nucleici da altri componenti cellulari (lipidi e proteine) -Separare gli acidi nucleici tra loro -Rompere la membrana
PCR. PCR o reazione di polimerizzazione a catena. Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte
 PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
PCR Prof.ssa Flavia Frabetti PCR o reazione di polimerizzazione a catena Fine anni 80 Amplificazione esponenziale di DNA. Puo amplificare un tratto di DNA per piu di 1 milione di volte Permette di estrarre
Polimorfismi LEZIONE 6. By NA 1
 Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Polimorfismi LEZIONE 6 By NA 1 * Polimorfismo Variazione presente nella popolazione con una frequenza superiore a 1% Variazioni nell aspetto By NA 2 Polimorfismo proteico Variazione presente nella popolazione
Analisi Molecolare di sequenze di acidi nucleici
 Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Analisi Molecolare di sequenze di acidi nucleici 1. L Analisi di restrizione di frammenti o RFLP (Restriction Fragment Lenght Polymorphism) di DNA comporta lo studio delle dimensioni dei frammenti di DNA
Real Time PCR. La PCR Real Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico.
 eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
eal Time PC La PC eal Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PC eal Time, oltre a fungere da termociclatori,
REAZIONE A CATENA DELLA POLIMERASI. ( PCR =Polymerase Chain Reaction)
 REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
REAZIONE A CATENA DELLA POLIMERASI ( PCR =Polymerase Chain Reaction) Verso la metà degli anni 80, il biochimico Kary Mullis mise a punto un metodo estremamente rapido e semplice per produrre una quantità
Metodi di analisi mutazionale
 Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
Metodi di analisi mutazionale I metodi impiegati per l analisi di mutazioni o polimorfismi nel DNA genomico possono essere suddivise in due principali categorie: (1) metodi per individuare mutazioni note,
TECNICHE DI BIOLOGIA MOLECOLARE. LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici
 TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
TECNICHE DI BIOLOGIA MOLECOLARE LA REAZIONE POLIMERASICA A CATENA Principi teorici e aspetti pratici POLYMERASE CHAIN REACTION (PCR) 1955 A. Kronembreg e coll. (Stanford University) scoprono la DNA-polimerasi
PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b)
 PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
PRINCIPALI TIPI DI PCR a) RT-PCR: serve a valutare l espressione di un gene tramite l amplificazione dell mrna da esso trascritto PCR COMPETITIVA: serve a valutare la concentrazione iniziale di DNA o RNA
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro
 di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
di Milazzo Gabriele Mangiagli Graziella Paternò Valentina Lomagno Valeria Mangione Enza Prof. C. Di Pietro Polymerase Chain Reaction Inventata a metà degli anni 80 da Kary Mullis, è a tutt oggi uno strumento
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E
 Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
Si può ottenere un a grande quantità di copie di DNA a partire da una singola molecola di DNA stampo La reazione è semplice ed automatizzabile E possibile scegliere selettivamente cosa amplificare (specificità)
ELETTROFORESI SU GEL
 ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
ELETTROFORESI SU GEL Permette la separazione di frammenti di DNA/RNA da una miscela complessa E una tecnica fondamentale per: l analisi (elettroforesi analitica) la purificazione degli acidi nucleici (elettroforesi
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino
 La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
La reazione a catena della polimerasi (PCR) di Ofelia Leone e Vincenzo Mandarino La Polymerase Chain Reaction (PCR) o reazione di amplificazione a catena è una tecnica che permette di amplificare una specifica
Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo. alla realtà di ogni giorno
 Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
Editoriale n.10 Newsletter aprile 2013 Come si traccia un alimento di origine animale? Dalle lasagne con carne di cavallo alla realtà di ogni giorno Identificare la specie, un obiettivo fondamentale quando
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR)
 AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
AMPLIFICAZIONE IN VITRO DEL DNA REAZIONE A CATENA DELLA POLIMERASI (PCR) PCR: reazione polimerasica a catena Inventata da Kary Mullis negli anni 80 (premio Nobel 1993) Serve per ottenere una grande quantita
SAGE: Serial Analysis of Gene Expression
 SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
SAGE: Serial Analysis of Gene Expression L insieme di tutti gli mrna presenti in una cellula si definisce trascrittoma. Ogni trascrittoma ha una composizione complessa, con migliaia di mrna diversi, ciascuno
ANALISI POST-GENOMICHE TRASCRITTOMA: CONTENUTO DI RNA DI UNA CELLULA.
 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA RT-PCR REAL TIME PCR NORTHERN BLOTTING ANALISI POST-GENOMICHE Conoscere la sequenza genomica di un organismo non è che l inizio di una serie di esperimenti
Tecniche molecolari per lo studio degli acidi nucleici
 Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
Tecniche molecolari per lo studio degli acidi nucleici Prof.ssa Flavia Frabetti aa. 2010-11 Estrazione acidi nucleici (DNA o RNA) Verifica tramite elettroforesi su gel di agarosio Amplificazione o clonaggio
I marcatori molecolari. Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene
 I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
I marcatori molecolari Dipartimento di Scienze Agronomiche e Genetica Vegetale Agraria Corso di Genetica Agraria Giovanna Attene Marcatori molecolari del DNA I marcatori molecolari sono sequenze di DNA
PCR - Polymerase Chain Reaction. ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993).
 End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
End point PCR vs quantitative Real-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando
Metodiche Molecolari
 Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
Metodiche Molecolari La rivelazione degli acidi nucleici virali è un altro saggio che può essere utilizzato sia per verificare la presenza di un virus in un determinato campione biologico, sia per studiare
METODI DI MARCATURA DEGLI ACIDI NUCLEICI
 METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
METODI DI MARCATURA DEGLI ACIDI NUCLEICI Marcatura di acidi nucleici Una sonda per ibridazione è una molecola di DNA marcata, con una sequenza complementare al DNA bersaglio da individuare. Poiché la sonda
PCR. (Reazione a catena della polimerasi) ESTRAZIONE del DNA da gel di AGAROSIO. Corso di INGEGNERIA GENETICA, Prof. Renato Fani,
 PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
PCR (Reazione a catena della polimerasi) & ESTRAZIONE del DNA da gel di AGAROSIO Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.2 La PCR (Polymerase Chain Reaction) è una tecnica
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di. DNA umano. 22-26 giugno 2009
 Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Analisi di polimorfismi usati per la caratterizzazione di profili genetici in campioni di 22-26 giugno 2009 DNA umano 1 GENETICA FORENSE La genetica forense applica tecniche di biologia molecolare al fine
Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dt) oppure random esameri).
 Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
Retrotrascrizione l mrna viene convertito in cdna per mezzo dell enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di
8-06-2010. BLOTTING degli acidi nucleici: RT-PCR SOUTHERN BLOTTING NORTHERN BLOTTING. BLOTTING delle proteine: NORTHERN BLOTTING SOUTHERN BLOTTING
 8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
8-06-2010 TECNICHE PER L ANALISI DELL ESPRESSIONE GENICA BLOTTING degli acidi nucleici: RT-PCR REAL TIME PCR SOUTHERN BLOTTING NORTHERN BLOTTING NORTHERN BLOTTING BLOTTING delle proteine: MICROARRAYS MACROARRAYS
PROGETTO BIOFORM Corso didattico sperimentale. Esercizio. Tipizzazione del gene PV92
 PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
PROGETTO BIOFORM Corso didattico sperimentale Esercizio Tipizzazione del gene PV92 Elementi trasponibili Che cosa sono gli elementi trasponibili? Sono segmenti di DNA che sono in grado di trasferirsi in
Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta)
 Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta) E il termine generico che indica una serie di tecniche di separazione di molecole simili in
Cromatografia Dal greco chroma : colore, graphein : scrivere 1903 Mikhail Twsett (pigmenti vegetali su carta) E il termine generico che indica una serie di tecniche di separazione di molecole simili in
Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici
 "Focus su sicurezza d'uso e nutrizionale degli alimenti" 21-22 Novembre 2005 Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici Simona
"Focus su sicurezza d'uso e nutrizionale degli alimenti" 21-22 Novembre 2005 Applicazione dei metodi rapidi alla microbiologia alimentare: Real Time PCR per la determinazione dei virus enterici Simona
Immagine di una sequenza ben riuscita. Sequences troubleshooting
 Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
Immagine di una sequenza ben riuscita. Sequences troubleshooting Sequenza fallita Reazione fallita: non presenta picchi definiti e ha un alto rumore di fondo. il primer non trova sito di annealing il DNA
scaricato da www.sunhope.it LA TECNOLOGIA DEI MICROARRAYS
 LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
LA TECNOLOGIA DEI MICROARRAYS 1 8 anni dopo: 4162 riferimenti bibliografici sui microarrays I microarrays misurano il livello di espressione degli mrna trascritti dai geni del sistema biologico di interesse
PCR Polymerase Chain Reaction = Reazione a catena della polimerasi
 PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
PR Polymerase hain Reaction = Reazione a catena della polimerasi mplifica un frammento di D di cui si conosce almeno in parte la sequenza Utilizza un enzima, la D Polimerasi, per copiare una molecola di
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3. Criteri di identificazione
 Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Identificazione dei microrganismi mediante tecniche di biologia molecolare Esercitazione n 3 Criteri di identificazione Tradizionale (fenotipo) Tecniche di biologia molecolare Il livello di risoluzione
Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine
 Training Course 2010 Stem Cell Differentiation Napoli, 9-12 Novembre Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine Cristina D
Training Course 2010 Stem Cell Differentiation Napoli, 9-12 Novembre Analisi dei marcatori molecolari della pluripotenza e del differenziamento delle cellule embrionali staminali (ES) murine Cristina D
Diagnostica Biomolecolare: Tecnologie e Applicazioni
 Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Diagnostica Biomolecolare: Tecnologie e Applicazioni Preparazione dei campioni: (Estrazione del DNA o dell RNA dal tessuto di interesse) Analisi delle mutazioni: SSCP DHPLC Dot blot - Southern - PCR (ARMS
Analisi di sequenziamento degli acidi nucleici
 Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
Analisi di sequenziamento degli acidi nucleici In questa lezione darò qualche breve cenno sui metodi di sequenziamento del DNA e specialmente quelli con marcatura fluorescente che attualmente vengono effettuati
Analisi qualitativa con Agilent BioAnalyzer. cdna, RNA Totale e Messaggero e small RNA
 Il Servizio di Biologia Molecolare si è di recente dotato di uno strumento per l analisi qualitativa e quantitativa degli acidi nucleici Agilent Bioanalyzer 2100 e ha implementato tale tecnologia nell
Il Servizio di Biologia Molecolare si è di recente dotato di uno strumento per l analisi qualitativa e quantitativa degli acidi nucleici Agilent Bioanalyzer 2100 e ha implementato tale tecnologia nell
REAZIONI ORGANICHE Variazioni di energia e velocità di reazione
 REAZIONI ORGANICHE Variazioni di energia e velocità di reazione Abbiamo visto che i composti organici e le loro reazioni possono essere suddivisi in categorie omogenee. Per ottenere la massima razionalizzazione
REAZIONI ORGANICHE Variazioni di energia e velocità di reazione Abbiamo visto che i composti organici e le loro reazioni possono essere suddivisi in categorie omogenee. Per ottenere la massima razionalizzazione
La farmacogeneticanella gestione del paziente con tumore colorettale, l esperienza traslazionaleal CRO di Aviano
 SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
SOC di Farmacologia Sperimentale e Clinica VI CONGRESSO NAZIONALE FITeLab Slow Medicine Laboratory: IT s the Future 1-2-3 Ottobre 2015 Ospedale Borgo Roma (Verona) La farmacogeneticanella gestione del
METODI ALTERNATIVI. PCR digitale per la rilevazione e quantificazione. assoluta del DNA
 METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
METODI ALTERNATIVI PCR digitale per la rilevazione e quantificazione assoluta del DNA Roma, 25 settembre 2013 Giuseppina Buonincontro IZS PLV- S.S. Controllo Alimenti La prima generazione di PCR consente
LE MOLECOLE INFORMAZIONALI. Lezioni d'autore Treccani
 LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
LE MOLECOLE INFORMAZIONALI Lezioni d'autore Treccani Introduzione (I) I pionieri della biologia molecolare, scoperta la struttura degli acidi nucleici, pensarono di associare al DNA una sequenza di simboli,
Metodiche di analisi del DNA (cenni) (Ingegneria genetica)
 DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
DNA Metodiche di analisi del DNA (cenni) (Ingegneria genetica) Principali tecniche di base Enzimi di restrizione (Endonucleasi) Gel elettroforesi Ibridizzazione PCR (Polymerase Chain Reaction) Sequenziamento
PCR (Polymerase Chain Reaction)
 PCR (Polymerase Chain Reaction) Metodo enzimatico estremamente rapido e semplice per produrre una quantità illimitata di copie della sequenza di un singolo gene Sometime a good idea comes to yow when you
PCR (Polymerase Chain Reaction) Metodo enzimatico estremamente rapido e semplice per produrre una quantità illimitata di copie della sequenza di un singolo gene Sometime a good idea comes to yow when you
Elettroforesi. Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita.
 Elettroforesi Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita. A qualunque ph diverso dal pi le proteine hanno una carica netta quindi,
Elettroforesi Elettroforesi: processo per cui molecole cariche si separano in un campo elettrico a causa della loro diversa mobilita. A qualunque ph diverso dal pi le proteine hanno una carica netta quindi,
DNA - RNA. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi.
 DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
DNA - RNA Le unità fondamentali costituenti il DNA e l RNA sono i Nucleotidi. Nucleotide = Gruppo Fosforico + Zucchero Pentoso + Base Azotata. Esistono 4 basi azotate per il DNA e 4 per RNA Differenze
Protocollo Crime Scene Investigation
 Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
Protocollo Crime Scene Investigation Precauzioni da adottare in laboratorio: - non mangiare o bere - indossare sempre i guanti quando si maneggiano i tubini, i gel, le micropipette - nel dubbio, chiedere!
SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo
 C.R.A. Consiglio per la Ricerca in Agricoltura Centro di ricerca per la genomica Fiorenzuola d Arda (Piacenza) SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo Tutor: Dott. Gianni TACCONI Studente:
C.R.A. Consiglio per la Ricerca in Agricoltura Centro di ricerca per la genomica Fiorenzuola d Arda (Piacenza) SELEZIONE ASSISTITA PER LA RESISTENZA patogeni in orzo Tutor: Dott. Gianni TACCONI Studente:
Progetto della classe II C
 Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Progetto della classe II C Preparazione allo svolgimento dell esperienza La II C è preparata all esperienza presso il centro di ricerca E.B.R.I. iniziando un intenso lavoro di approfondimento sulla genetica
Biosintesi non ribosomiale di metaboliti peptidici bioattivi
 Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
Biosintesi non ribosomiale di metaboliti peptidici bioattivi Principali bersagli degli antibiotici Gli antibiotici derivano per la maggior parte da composti naturali Strutture di alcuni peptidi bioattivi
In anticipo sul futuro. La tecnica termografica come strumento di verifica e diagnosi di malfunzionamenti
 La tecnica termografica come strumento di verifica e diagnosi di malfunzionamenti La tecnica termografica genesi e definizioni Termografia: definizioni e principio di misura Dal greco: Scrittura del calore
La tecnica termografica come strumento di verifica e diagnosi di malfunzionamenti La tecnica termografica genesi e definizioni Termografia: definizioni e principio di misura Dal greco: Scrittura del calore
[ Analisi della. concentrazione] di Luca Vanzulli. Pag. 1 di 1
![[ Analisi della. concentrazione] di Luca Vanzulli. Pag. 1 di 1 [ Analisi della. concentrazione] di Luca Vanzulli. Pag. 1 di 1](/thumbs/30/14397601.jpg) [ Analisi della concentrazione] di Luca Vanzulli Pag. 1 di 1 LA CONCENTRAZIONE NELL ANALISI DELLE VENDITE L analisi periodica delle vendite rappresenta un preziosissimo indicatore per il monitoraggio del
[ Analisi della concentrazione] di Luca Vanzulli Pag. 1 di 1 LA CONCENTRAZIONE NELL ANALISI DELLE VENDITE L analisi periodica delle vendite rappresenta un preziosissimo indicatore per il monitoraggio del
Tecniche di sequenziamento del DNA
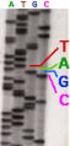 Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Tecniche di sequenziamento del DNA -Metodo di Maxam e Gilbert (della degradazione chimica del DNA) -Metodo di Sanger (a terminazione di catena) Metodo di Maxam-Gilbert Questo metodo, basato sulla degradazione
Quantificazione di. legionella pneumophila. mediante metodo biomolecolare
 Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
Quantificazione di legionella pneumophila mediante metodo biomolecolare Laboratorio di Epidemiologia Genetica e Genomica di Sanità Pubblica Istituto di Igiene LABORATORIO DI EPIDEMIOLOGIA GENETICA: ATTIVITA
XRF - Fluorescenza a raggi X
 XRF - Fluorescenza a raggi X La Fluorescenza a raggi X (X-Ray-Fluorescence) è una metodologia che consente di determinare la composizione qualitativa e quantitativa di solidi, liquidi e polveri, usando
XRF - Fluorescenza a raggi X La Fluorescenza a raggi X (X-Ray-Fluorescence) è una metodologia che consente di determinare la composizione qualitativa e quantitativa di solidi, liquidi e polveri, usando
DNA sequencing. Reading Genomes. Giovanni Bacci
 Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
Reading Genomes Giovanni Bacci Evoluzione del sequenziamento 1977 Frederick Sanger Prima tecnica di sequenziamento 1987 Applyed Biosystems Prima macchina automatica per il sequenziamento del DNA 1998 Phil
La mutazione è una modificazione della sequenza delle basi del DNA
 La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
La mutazione è una modificazione della sequenza delle basi del DNA Le mutazioni sono eventi rari e importanti in quanto sono alla base dell evoluzione biologica Le mutazioni possono essere spontanee (dovute
Strategie di purificazione di proteine
 Laurea Magistrale in Scienze e Biotecnologie degli Alimenti Strategie di purificazione di proteine Lezione n.xx-2-23 PRINCIPIO - ALIMENTI DA MATRICI SOLIDE E LIQUIDE MATRICI SOLIDE - ROMPERE LA STRUTTURA
Laurea Magistrale in Scienze e Biotecnologie degli Alimenti Strategie di purificazione di proteine Lezione n.xx-2-23 PRINCIPIO - ALIMENTI DA MATRICI SOLIDE E LIQUIDE MATRICI SOLIDE - ROMPERE LA STRUTTURA
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR. Dott. Paolo Cascio
 LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
LEZIONE X FUNZIONAMENTO E APPLICAZIONI DELLA PCR Dott. Paolo Cascio Tecnica della reazione a catena della DNA polimerasi o PCR (Polymerase Chain Reaction) 1) Introdotta da Kary Mullis alla metà degli anni
PRINCIPALI APPLICAZIONI DELLA PCR. PRINCIPALI TIPI DI PCR a) PRINCIPALI TIPI DI PCR b) PRINCIPALI TIPI DI PCR a)
 EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
EAZIONE A CATENA DELLA POLIMEASI (PC) PINCIPALI APPLICAZIONI DELLA PC UPPE primer LOWE primer GENE X 1. Identificazione di agenti patogeni nell uomo, negli animali o negli alimenti (batteri e virus) 2.
Classificazione dei Sensori. (raccolta di lucidi)
 Classificazione dei Sensori (raccolta di lucidi) 1 Le grandezze fisiche da rilevare nei processi industriali possono essere di varia natura; generalmente queste quantità sono difficili da trasmettere e
Classificazione dei Sensori (raccolta di lucidi) 1 Le grandezze fisiche da rilevare nei processi industriali possono essere di varia natura; generalmente queste quantità sono difficili da trasmettere e
La possibilita di conoscere i geni deriva dalla capacita di manipolarli:
 La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
La possibilita di conoscere i geni deriva dalla capacita di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione Il gene o la sequenza
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE
 LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
LA DIAGNOSTICA MOLECOLARE E I TUMORI DEL SANGUE UNA NUOVA FRONTIERA NELLA DIAGNOSI DI ALCUNI TUMORI La diagnostica molecolare ha l obiettivo di accertare un ampia varietà di patologie (infettive, oncologiche
Generazione Automatica di Asserzioni da Modelli di Specifica
 UNIVERSITÀ DEGLI STUDI DI MILANO BICOCCA FACOLTÀ DI SCIENZE MATEMATICHE FISICHE E NATURALI Corso di Laurea Magistrale in Informatica Generazione Automatica di Asserzioni da Modelli di Specifica Relatore:
UNIVERSITÀ DEGLI STUDI DI MILANO BICOCCA FACOLTÀ DI SCIENZE MATEMATICHE FISICHE E NATURALI Corso di Laurea Magistrale in Informatica Generazione Automatica di Asserzioni da Modelli di Specifica Relatore:
Lezioni di biotecnologie
 Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Lezioni di biotecnologie 2 Lezione 2 Analisi del DNA e delle proteine 3 Analizzare DNA e proteine Per le applicazioni delle biotecnologie è di fondamentale importanza: 1. essere in grado di identificare
Piano di gestione della qualità
 Piano di gestione della qualità Pianificazione della qualità Politica ed obiettivi della qualità Riferimento ad un eventuale modello di qualità adottato Controllo della qualità Procedure di controllo.
Piano di gestione della qualità Pianificazione della qualità Politica ed obiettivi della qualità Riferimento ad un eventuale modello di qualità adottato Controllo della qualità Procedure di controllo.
Diagnosi genetiche molecolari
 Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Diagnosi genetiche molecolari INDICAZIONI per la diagnosi genetica Ricorrenza nella famiglia di una malattia genetica - identificazione dei portatori - confermare la diagnosi basata sul quadro clinico
Verifiche qualitative dell RNA e cdna in ambito dei trials BCR/ABL e AML translocation programme. Massimo Degan, CRO Aviano (PN)
 e cdna in ambito dei trials BCR/ABL e AML translocation programme Massimo Degan, CRO Aviano (PN) perchè è importante verificare l RNA La verifica della qualità dell RNA nei saggi diagnostico-molecolari
e cdna in ambito dei trials BCR/ABL e AML translocation programme Massimo Degan, CRO Aviano (PN) perchè è importante verificare l RNA La verifica della qualità dell RNA nei saggi diagnostico-molecolari
Maria Antonietta Lepore. Principali tecniche di biologia molecolare clinica
 Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
Maria Antonietta Lepore Principali tecniche di biologia molecolare clinica Copyright MMIX ARACNE editrice S.r.l. www.aracneeditrice.it info@aracneeditrice.it via Raffaele Garofalo, 133 a/b 00173 Roma (06)
MISURE DI POTERE CALORIFICO E COMPOSIZIONE
 MISURE DI POTERE CALORIFICO E COMPOSIZIONE Potere calorifico dei combustibili: bomba calorimetrica e calorimetro di Junkers Composizione: gascromatografia Composizione dei gas combusti: o Sonda λ o Strumenti
MISURE DI POTERE CALORIFICO E COMPOSIZIONE Potere calorifico dei combustibili: bomba calorimetrica e calorimetro di Junkers Composizione: gascromatografia Composizione dei gas combusti: o Sonda λ o Strumenti
Sistemi di tracciabilità per un attestato di identità molecolare. FEM 2 - Ambiente S.r.l. Spin-off dell Università degli Studi di Milano-Bicocca
 Sistemi di tracciabilità per un attestato di identità molecolare FEM 2 - Ambiente S.r.l. Spin-off dell Università degli Studi di Milano-Bicocca PROBLEMA L identificazione e il controllo della filiera delle
Sistemi di tracciabilità per un attestato di identità molecolare FEM 2 - Ambiente S.r.l. Spin-off dell Università degli Studi di Milano-Bicocca PROBLEMA L identificazione e il controllo della filiera delle
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale
 HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
HI-TECH IN SANITA'. MINI-INVASIVITA' 2.0: nuove tecnologie al servizio dell'appropriatezza e della bioetica professionale L evoluzione della professione tecnica tra robotica, automazione e nuove tecnologie
Usando il pendolo reversibile di Kater
 Usando il pendolo reversibile di Kater Scopo dell esperienza è la misurazione dell accelerazione di gravità g attraverso il periodo di oscillazione di un pendolo reversibile L accelerazione di gravità
Usando il pendolo reversibile di Kater Scopo dell esperienza è la misurazione dell accelerazione di gravità g attraverso il periodo di oscillazione di un pendolo reversibile L accelerazione di gravità
Energia nelle reazioni chimiche. Lezioni d'autore di Giorgio Benedetti
 Energia nelle reazioni chimiche Lezioni d'autore di Giorgio Benedetti VIDEO Introduzione (I) L energia chimica è dovuta al particolare arrangiamento degli atomi nei composti chimici e le varie forme di
Energia nelle reazioni chimiche Lezioni d'autore di Giorgio Benedetti VIDEO Introduzione (I) L energia chimica è dovuta al particolare arrangiamento degli atomi nei composti chimici e le varie forme di
Automazione Industriale (scheduling+mms) scheduling+mms. adacher@dia.uniroma3.it
 Automazione Industriale (scheduling+mms) scheduling+mms adacher@dia.uniroma3.it Introduzione Sistemi e Modelli Lo studio e l analisi di sistemi tramite una rappresentazione astratta o una sua formalizzazione
Automazione Industriale (scheduling+mms) scheduling+mms adacher@dia.uniroma3.it Introduzione Sistemi e Modelli Lo studio e l analisi di sistemi tramite una rappresentazione astratta o una sua formalizzazione
Capitolo 2 Distribuzioni di frequenza
 Edizioni Simone - Vol. 43/1 Compendio di statistica Capitolo 2 Distribuzioni di frequenza Sommario 1. Distribuzioni semplici. - 2. Distribuzioni doppie. - 3. Distribuzioni parziali: condizionate e marginali.
Edizioni Simone - Vol. 43/1 Compendio di statistica Capitolo 2 Distribuzioni di frequenza Sommario 1. Distribuzioni semplici. - 2. Distribuzioni doppie. - 3. Distribuzioni parziali: condizionate e marginali.
Lezione XLI-XLII martedì 17-1-2012
 Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
Lezione XLI-XLII martedì 17-1-2012 corso di genomica aula 8 orario : Martedì ore 14.00-16.00 Giovedì ore 13.00-15.00 Esami 31- gennaio 2012 7- febbraio 2012 28 - febbraio 2012 D. Frezza Esercitazione II
Le Biomolecole I parte. Lezioni d'autore di Giorgio Benedetti
 Le Biomolecole I parte Lezioni d'autore di Giorgio Benedetti LE BIOMOLECOLE Le biomolecole, presenti in tutti gli esseri viventi, sono molecole composte principalmente da carbonio, idrogeno, azoto e ossigeno.
Le Biomolecole I parte Lezioni d'autore di Giorgio Benedetti LE BIOMOLECOLE Le biomolecole, presenti in tutti gli esseri viventi, sono molecole composte principalmente da carbonio, idrogeno, azoto e ossigeno.
Perché il logaritmo è così importante?
 Esempio 1. Perché il logaritmo è così importante? (concentrazione di ioni di idrogeno in una soluzione, il ph) Un sistema solido o liquido, costituito da due o più componenti, (sale disciolto nell'acqua),
Esempio 1. Perché il logaritmo è così importante? (concentrazione di ioni di idrogeno in una soluzione, il ph) Un sistema solido o liquido, costituito da due o più componenti, (sale disciolto nell'acqua),
NUCLEOTIDI e ACIDI NUCLEICI
 NUCLEOTIDI e ACIDI NUCLEICI Struttura dei nucleotidi Il gruppo fosfato conferisce carica negativa e proprietà acide FUNZIONI DEI NUCLEOTIDI MOLECOLE DI RISERVA DI ENERGIA L idrolisi dei nucleosidi trifosfato
NUCLEOTIDI e ACIDI NUCLEICI Struttura dei nucleotidi Il gruppo fosfato conferisce carica negativa e proprietà acide FUNZIONI DEI NUCLEOTIDI MOLECOLE DI RISERVA DI ENERGIA L idrolisi dei nucleosidi trifosfato
La trascrizione nei procarioti. Prof. Savino; dispense di Biologia Molecolare, Corso di Laurea in Biotecnologie
 La trascrizione nei procarioti Concetti base Nucleoside base purinica o pirimidinica legata alla posizione 1 dell anello pentoso Nucleotide base azotata-pentoso-fosfato Concetti base La trascrizione comporta
La trascrizione nei procarioti Concetti base Nucleoside base purinica o pirimidinica legata alla posizione 1 dell anello pentoso Nucleotide base azotata-pentoso-fosfato Concetti base La trascrizione comporta
Parte I. Prima Parte
 Parte I Prima Parte Capitolo 1 Introduzione generale 1.1 Il problema dell assegnazione Corsi-Borsisti Il problema dell assegnazione delle borse dei corsi ai vari studenti può essere riassunto nei punti
Parte I Prima Parte Capitolo 1 Introduzione generale 1.1 Il problema dell assegnazione Corsi-Borsisti Il problema dell assegnazione delle borse dei corsi ai vari studenti può essere riassunto nei punti
A cura di Giorgio Mezzasalma
 GUIDA METODOLOGICA PER IL MONITORAGGIO E VALUTAZIONE DEL PIANO DI COMUNICAZIONE E INFORMAZIONE FSE P.O.R. 2007-2013 E DEI RELATIVI PIANI OPERATIVI DI COMUNICAZIONE ANNUALI A cura di Giorgio Mezzasalma
GUIDA METODOLOGICA PER IL MONITORAGGIO E VALUTAZIONE DEL PIANO DI COMUNICAZIONE E INFORMAZIONE FSE P.O.R. 2007-2013 E DEI RELATIVI PIANI OPERATIVI DI COMUNICAZIONE ANNUALI A cura di Giorgio Mezzasalma
Università degli Studi di Torino Dipartimento di Anatomia, Farmacologia e Medicina Legale Laboratorio di Scienze Criminalistiche.
 Università degli Studi di Torino Dipartimento di Anatomia, Farmacologia e Medicina Legale Laboratorio di Scienze Criminalistiche Genetica Forense Sarah Gino SAL (STATO AVANZAMENTO LAVORI) Informazioni
Università degli Studi di Torino Dipartimento di Anatomia, Farmacologia e Medicina Legale Laboratorio di Scienze Criminalistiche Genetica Forense Sarah Gino SAL (STATO AVANZAMENTO LAVORI) Informazioni
I processi di tempra sono condotti sul manufatto finito per generare sforzi residui di compressione in superficie. Vengono sfruttate allo scopo
 I processi di tempra sono condotti sul manufatto finito per generare sforzi residui di compressione in superficie. Vengono sfruttate allo scopo diverse metodologie. 1 La tempra termica (o fisica) si basa
I processi di tempra sono condotti sul manufatto finito per generare sforzi residui di compressione in superficie. Vengono sfruttate allo scopo diverse metodologie. 1 La tempra termica (o fisica) si basa
Sommario Ottica geometrica... 2 Principio di Huygens-Fresnel... 4 Oggetto e immagine... 6 Immagine reale... 7 Immagine virtuale...
 IMMAGINI Sommario Ottica geometrica... 2 Principio di Huygens-Fresnel... 4 Oggetto e immagine... 6 Immagine reale... 7 Immagine virtuale... 9 Immagini - 1/11 Ottica geometrica È la branca dell ottica che
IMMAGINI Sommario Ottica geometrica... 2 Principio di Huygens-Fresnel... 4 Oggetto e immagine... 6 Immagine reale... 7 Immagine virtuale... 9 Immagini - 1/11 Ottica geometrica È la branca dell ottica che
Anno 2014. Rapporto ambientale
 Anno 2014 Rapporto ambientale 1 ANNO 2014 Nell anno 2005 la SITI TARGHE S.r.l. ha ottenuto la certificazione ambientale secondo la norma internazionale ISO 14001:2004, rinnovata nel 2008, nel 2011 e nel
Anno 2014 Rapporto ambientale 1 ANNO 2014 Nell anno 2005 la SITI TARGHE S.r.l. ha ottenuto la certificazione ambientale secondo la norma internazionale ISO 14001:2004, rinnovata nel 2008, nel 2011 e nel
Corso di. Dott.ssa Donatella Cocca
 Corso di Statistica medica e applicata Dott.ssa Donatella Cocca 1 a Lezione Cos'è la statistica? Come in tutta la ricerca scientifica sperimentale, anche nelle scienze mediche e biologiche è indispensabile
Corso di Statistica medica e applicata Dott.ssa Donatella Cocca 1 a Lezione Cos'è la statistica? Come in tutta la ricerca scientifica sperimentale, anche nelle scienze mediche e biologiche è indispensabile
Tecniche di microscopia
 Tecniche di microscopia I microscopi permettono di vedere l estremamente piccolo I microscopi ottici utilizzano lenti di vetro in grado di deflettere e focalizzare i raggi luminosi per riprodurre le immagini
Tecniche di microscopia I microscopi permettono di vedere l estremamente piccolo I microscopi ottici utilizzano lenti di vetro in grado di deflettere e focalizzare i raggi luminosi per riprodurre le immagini
Figura 1. Rappresentazione della doppia elica di DNA e struttura delle differenti basi.
 Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
Sommario La molecola di DNA è deputata a conservare le informazioni genetiche necessarie per lo sviluppo ed il funzionamento degli organismi viventi. Poiché contiene le istruzioni per la costruzione delle
Macchine a stati finiti G. MARSELLA UNIVERSITÀ DEL SALENTO
 Macchine a stati finiti 1 G. MARSELLA UNIVERSITÀ DEL SALENTO Introduzione Al più alto livello di astrazione il progetto logico impiega un modello, la cosiddetta macchina a stati finiti, per descrivere
Macchine a stati finiti 1 G. MARSELLA UNIVERSITÀ DEL SALENTO Introduzione Al più alto livello di astrazione il progetto logico impiega un modello, la cosiddetta macchina a stati finiti, per descrivere
Macromolecole Biologiche. I domini (III)
 I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
I domini (III) Domini α/β La cross over connection è l unità costitutiva su cui si basa la topologia di 3 tipi di domini α/β osservati nelle proteine: - α/β barrel - motivi ricchi di Leu (fold a ferro
Le strumentazioni laser scanning oriented per i processi di censimento anagrafico dei patrimoni
 CONVEGNO FACILITY MANAGEMENT: LA GESTIONE INTEGRATA DEI PATRIMONI PUBBLICI GENOVA FACOLTA DI ARCHITETTURA 06.07.2010 Le strumentazioni laser scanning oriented per i processi di censimento anagrafico dei
CONVEGNO FACILITY MANAGEMENT: LA GESTIONE INTEGRATA DEI PATRIMONI PUBBLICI GENOVA FACOLTA DI ARCHITETTURA 06.07.2010 Le strumentazioni laser scanning oriented per i processi di censimento anagrafico dei
immagine Biologia applicata alla ricerca bio-medica Materiale Didattico Docente: Di Bernardo
 Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
Esperto in processi innovativi di sintesi biomolecolare applicata a tecniche di epigenetica Materiale Didattico Biologia applicata alla ricerca bio-medica immagine Docente: Di Bernardo TECNICHE PER L ANALISI
Gestione Turni. Introduzione
 Gestione Turni Introduzione La gestione dei turni di lavoro si rende necessaria quando, per garantire la continuità del servizio di una determinata struttura, è necessario che tutto il personale afferente
Gestione Turni Introduzione La gestione dei turni di lavoro si rende necessaria quando, per garantire la continuità del servizio di una determinata struttura, è necessario che tutto il personale afferente
Librerie digitali. Video. Gestione di video. Caratteristiche dei video. Video. Metadati associati ai video. Metadati associati ai video
 Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
Video Librerie digitali Gestione di video Ogni filmato è composto da più parti Video Audio Gestito come visto in precedenza Trascrizione del testo, identificazione di informazioni di interesse Testo Utile
La base di partenza per la maggior parte dei processi produttivi di materiali ceramici sono le sospensioni. Queste si ottengono dalla miscelazione di
 La base di partenza per la maggior parte dei processi produttivi di materiali ceramici sono le sospensioni. Queste si ottengono dalla miscelazione di un solido (polvere) che diverrà il ceramico, con un
La base di partenza per la maggior parte dei processi produttivi di materiali ceramici sono le sospensioni. Queste si ottengono dalla miscelazione di un solido (polvere) che diverrà il ceramico, con un
Analisi molecolare dei geni
 Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100 C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni
