PURIFICAZIONE E CARATTERIZZAZIONE BIOCHIMICA DI UNA TREALASI DI INSETTO ESPRESSA COME PROTEINA ETEROLOGA IN Escherichia coli.
|
|
|
- Natalia Cecchini
- 5 anni fa
- Visualizzazioni
Transcript
1 PURIFICAZIONE E CARATTERIZZAZIONE BIOCHIMICA DI UNA TREALASI DI INSETTO ESPRESSA COME PROTEINA ETEROLOGA IN Escherichia coli. La trealasi (α,α-trealosio glucoidrolasi EC ) è un enzima che catalizza l idrolisi del disaccaride trealosio in due molecole di glucosio che può così essere disponibile sia come fonte energetica sia come precursore della chitina. Negli insetti la trealasi svolge un ruolo fondamentale, essendo il trealosio e non il glucosio il principale zucchero circolante nell emolinfa. Il cdna codificante la trealasi del moscerino Chironomus riparius è stato inserito nel plasmide pmal-c5x e il ceppo di E. coli BL21 è stato trasformato con il costrutto così ottenuto. La produzione della proteina di interesse, fusa con la maltose binding protein (MBP), avviene a seguito dell induzione con IPTG. L obiettivo di questa esperienza è quello di purificare la proteina chimerica trealasi- MBP e di procedere alla caratterizzazione biochimica della trealasi. I GIORNO Preparazione dei campioni analitici I campioni analitici sono in larga parte destinati ad elettroforesi in condizioni denaturanti. - prelevare un aliquota (15 µl) di estratto totale e trasferirlo in provetta da 1,5 ml, da tenere in ghiaccio. Questo campione ( proteine totali ) sarà sottoposto ad analisi SDS-PAGE (v. tabella a pag. 5). - prelevare un aliquota (150 µl) di estratto totale e centrifugare per 5 min a rpm. Con questo passaggio ( chiarificazione dell estratto) si ottiene la separazione della frazione proteica solubile e di quella insolubile. - trasferire il surnatante chiarificato in una provetta da 1,5 ml, da tenere in ghiaccio. Nei prossimi giorni, questo campione ( proteine solubili ) sarà 1
2 sottoposto ad analisi SDS-PAGE (v. tabella a pag. 5). a saggi di attività e del contenuto proteico - dopo aver rimosso tutto il surnatante, porre la provetta con il pellet ( frazione insolubile ) in ghiaccio. Questo campione ( proteine insolubili ) sarà sottoposto ad analisi SDS-PAGE (v. tabella a pag. 5). Preparazione dei campioni preparativi - trasferire l estratto totale in 2 provette da 2 ml e centrifugare per 5 min a rpm. - trasferire il surnatante (frazione di proteine solubili) in una provetta da 4 ml che viene posta in ghiaccio (campione preparativo proteine solubili ). Questo campione sarà sottoposto a cromatografia di affinità. Cromatografia di affinità La trealasi-mbp viene purificata mediante cromatografia di affinità utilizzando una resina coniugata con amilosio. Equilibrazione della resina in tampone A (operazione già eseguita) - Aggiungere 0,5 ml di tampone A nella provetta contenente 0.4 ml di resina - Mescolare per inversione e centrifugare a 2000 rpm per 3 minuti - Allontanare il surnatante ed eseguire altri 4 lavaggi (volume totale: 2 ml, 4 volumi di colonna). Binding alla resina - Aggiungere alla resina il campione preparativo proteine solubili - Incubare a temperatura ambiente per 1 h in agitazione su ruota - Trasferire in colonna la sospensione di resina e campione preparativo, lasciare sedimentare e raccogliere il picco escluso, ossia tutto ciò che non si lega alla resina (conservare in ghiaccio). Washing - Lavare con 15 volumi di colonna (6 ml) di tampone A (scartare) 2
3 Eluizione - Eluire in 5 frazioni da 1 ml di tampone B (frazioni di eluizione = nuovo campione preparativo). Porre in ghiaccio. Tamponi per la purificazione - Preparare 10 ml di Tampone A (Tris- HCl 20 mm, NaCl 200 mm, EDTA 1mM, DTT 1mM, ph 7.4) partendo dalle seguenti soluzioni madre: Tris- HCl 200 mm ph 7.4 NaCl 1 M EDTA 100 mm DTT 100 mm Portare al volume prescritto aggiungendo acqua q.b. - Preparare 10 ml di Tampone B (Tris- HCl 20 mm, NaCl 200 mm, EDTA 1mM, DTT 1mM, maltosio 50 mm ph 7.4) partendo dalle seguenti soluzioni madre: Tris- HCl 200 mm ph 7.4 NaCl 1 M EDTA 100 mm DTT 100 mm Maltosio 500 mm Portare al volume prescritto aggiungendo acqua q.b. 3
4 Preparazione del gel di poliacrilamide in presenza di SDS (SDS-PAGE) L andamento della purificazione viene monitorato mediante analisi SDS-PAGE. In questo tipo di elettroforesi, condotta in condizioni denaturanti, si utilizza un sistema discontinuo di gel di poliacrilamide: si ha un running gel a ph 8.8, con una percentuale di poliacrilamide che può variare tra il 7 e il 15% (in questo caso il 10%) e uno stacking gel a ph 6.8, con una percentuale di poliacrilamide del 4%. La presenza dello stacking gel consente una compattazione delle bande proteiche che entrano sotto forma di bande molto sottili nel running gel, dove verranno separate in base alla loro massa molecolare. Il gel necessario alla separazione elettroforetica viene allestito come indicato di seguito. I campioni sono stati preparati per aggiunta di SDS-sample buffer. Running gel 10% (10 ml) H 2 O distillata 4.1 ml 1,5 M Tris/HCl, ph 8.8, 0.4% SDS 2.5 ml Acrilamide/Bisacrilamide 30% 3.3 ml Stacking gel 4% (3 ml) Ammonio persolfato (100 mg/ml) 90 µl TEMED 10 µl H 2 O distillata 1.8 ml 0.5 M Tris/HCl, ph 6.8, 0.4% SDS 750 µl Acrilamide/Bisacrilamide 30% 400 µl Ammonio persolfato (100 mg/ml) 30 µl TEMED 10 µl N.B: Ammonio persolfato (iniziatore) e TEMED (catalizzatore) vengono aggiunti alle soluzioni pochi attimi prima del caricamento nel gel caster. 4
5 II GIORNO Preparazione dei campioni per SDS-PAGE I campioni da caricare sul gel vengono preparati aggiungendo un opportuno volume di sample buffer 5X, contenente blu di bromofenolo (BBF), utile per seguire la corsa elettroforetica, glicerolo, che rende più denso il campione consentendone la stratificazione sul fondo del pozzetto, beta-mercaptoetanolo, che riduce i ponti disolfuro delle proteine favorendone la denaturazione, sodio dodecilsofato (SDS), un detergente anfipatico denaturante che conferisce alle proteine una carica negativa. Sample buffer 5X 0.25 M Tris/HCl ph % SDS + 20% glicerolo M -mercaptoetanolo + blu di bromofenolo (BBF). I campioni da sottoporre ad analisi SDS-PAGE vengono allestiti in base alla seguente tabella: Estratto totale Frazione solubile Picco escluso campione 8 µl 8 µl 8 µl 24 µl 24 µl 24 µl 24 µl 24 µl Sample 6 µl 6 µl 6 µl 6 µl 6 µl 6 µl 6 µl 6 µl Buffer 5X H 2 O 16 µl 16 µl 16 µl V totale 30 µl 30 µl 30 µl 30 µl 30 µl 30 µl 30 µl 30 µl Risospendere accuratamente la frazione insolubile (pellet) in 600 µl di sample buffer 1X. Una volta allestiti i campioni, si denaturano le proteine incubando a 100 C per 5 minuti. 5
6 Corsa elettroforetica Caricare sul gel 25 µl di ciascuno dei campioni precedentemente preparati e 3 µl dello standard di peso molecolare Estratto totale Fraz. solubile Fraz. insolubile Picco escluso Ordine di caricamento Tampone di corsa Versare il tampone di corsa (25 mm Tris, 250 mm glicina, ph 8.3, 0.1% SDS) negli apparati elettroforetici Collegare gli apparati per elettroforesi ad un generatore di corrente, applicando una corrente costante di 20 ma per lastrina (amperaggio costante). Fermare la corsa quando il colorante tracciante (blu di bromofenolo) raggiunge il fondo del running gel. Il gel viene lavato in H 2 O distillata e colorato con Gel-CODE Blue, un colorante commerciale contenente blu Coomassie e successivamente decolorato in acqua. Dosaggio della concentrazione proteica mediante il metodo di Bradford (Bradford M., Anal. Biochem., 1976, 72, 248) La concentrazione proteica dei campioni della purificazione viene determinata mediante il metodo di Bradford. Questo tipo di dosaggio rappresenta uno dei metodi più sensibili e rapidi per la determinazione della concentrazione dei campioni biologici. Il reattivo contiene il Coomassie Brilliant Blue G-250, un colorante anionico. In ambiente acido le proteine reagiscono con il colorante carico negativamente formando un complesso di un colore che varia da rosso-bruno a blu che ha un massimo di assorbimento a una lunghezza d onda di 595 nm. Il numero di molecole di colorante legato è approssimativamente proporzionale al numero di proteine presenti. La misura ottenuta non è, tuttavia, assoluta, pertanto per quantificare le proteine è necessario calibrare il metodo. Viene preparata pertanto una retta di taratura 6
7 utilizzando quantità note di albumina di siero bovino (BSA) come proteina di riferimento. Miscele di reazione per saggio di Bradford Campioni per retta di taratura Bianco 2X BSA 0.5 µg BSA 1 µg BSA 2 µg BSA 4 µg BSA 6 µg BSA 0.1 µg/µl 5 µl 10 µl 20 µl 40 µl 60 µl H 2 O 500 µl 495 µl 490 µl 480 µl 460 µl 440 µl Reattivo di Bradford 500 µl 500 µl 500 µl 500 µl 500 µl 500 µl Dosaggio dei campioni di interesse Frazione Picco solubile escluso (1:10) (1:10) campione 5 µl 5 µl 20 µl 20 µl 20 µl 20 µl 20 µl H 2 O 495 µl 495 µl 480 µl 480 µl 480 µl 480 µl 480 µl Reattivo di Bradford 500 µl 500 µl 500 µl 500 µl 500 µl 500 µl 500 µl - Allestire contemporaneamente le miscele della retta di taratura e dei campioni, direttamente nelle cuvette. - Tappare le provette, mescolare per agitazione ed incubare per 5 minuti a temperatura ambiente. - Leggere l'estinzione a 595 nm per ciascun campione. - Allestire una retta di taratura riportando in ascisse i µg di proteina e in ordinate i corrispondenti valori di estinzione (ΔE). - Determinare i µg di proteina presenti in cuvetta dalla retta di taratura; dividendo i µg ottenuti per il volume in µl utilizzato per il dosaggio, si ricava la concentrazione proteica dei campioni analizzati. Se è stata effettuata una diluizione del campione, si dovrà tenere conto del fattore di diluizione. 7
8 - Riportare i valori nella seguente tabella: Frazione solubile Picco escluso ΔE μg μl mg/ml Dialisi Riunire le frazioni di eluizione più ricche in proteine e dializzare con una membrana da dialisi con cut off Dalton utilizzando 1 L di tampone Na 2 HPO 4 25 mm, NaCl 75 mm ph 7.4 al fine di eliminare il maltosio presente. La dialisi viene condotta a 4 C over night. 8
9 III GIORNO Recupero campione dializzato Trasferire in una provetta pulita i campioni di proteina dializzati. Dosaggio della concentrazione proteica di campioni dializzati (metodo di Bradford) Dosare il contenuto proteico del campione dializzato seguendo la tabella riportata Bianco 2X Campione dializzato campione 20 µl H 2 O 500 µl 480 µl Reattivo Bradford 500 µl 500 µl Calcolare la concentrazione del campione utilizzando la retta di taratura già ottenuta in precedenza Frazione purificata dializzata ΔE μg μl mg/ml Dosaggio di attività dell enzima trealasi Gli enzimi sono proteine in grado di catalizzare determinate reazioni biologiche, con un alta specificità per i loro substrati. La concentrazione enzimatica viene determinata in base alla velocità della reazione catalizzata, quindi in base alla velocità con cui il substrato viene trasformato in condizioni definite. La velocità della reazione catalizzata è direttamente proporzionale alla concentrazione dell enzima. La velocità della reazione viene seguita nel tempo misurando la scomparsa di substrato o la comparsa di prodotto. Per la misura dell attività, quindi, si seguono nel tempo le variazioni di un opportuna proprietà del substrato o del prodotto. 9
10 Il ph, la temperatura, cofattori o altri componenti della miscela di reazione possono influire sulla velocità della reazione catalizzata. L attività enzimatica, ovvero la velocità della reazione catalizzata, viene espressa in unità. Per convenzione generale e per tutti gli enzimi, si definisce attività internazionale (U) l attività enzimatica che, in condizioni definite di temperatura e di ph, trasforma una μmole di substrato al minuto. Le unità misurate vengono riferite ad un volume unitario del campione in esame o ai mg di proteine presenti nel campione. Il rapporto U/mL di campione viene detto attività relativa, mentre il rapporto U/mg di proteine viene indicato come attività specifica dell enzima. Molti substrati e prodotti assorbono la luce nel visibile o nell ultravioletto e molti dosaggi si basano sull interconversione NAD(P)+/NAD(P)H. Sia la forma ossidata sia la forma ridotta di questo nucleotide assorbono la luce a 260 nm, ma solo la forma ridotta assorbe a 340 nm. Questo diverso spettro di assorbimento permette di utilizzare questo nucleotide per dosaggi basati su metodi spettrofotometrici. Quegli enzimi che non utilizzano in modo diretto NAD(P)+/NAD(P)H possono essere comunque dosati sfruttando questo tipo di interconversione mediante reazioni accoppiate. In tali reazioni, l enzima che deve essere dosato è accoppiato ad un altro enzima che utilizza il sistema NAD+/NADH mediante un intermedio comune. Il dosaggio dell attività trealasica viene effettuato allo spettrofotometro mediante l accoppiamento della reazione catalizzata dalla trealasi con due reazioni catalizzate da enzimi ausiliari, Glucosio-6-P deidrogenasi (G6PDH) e Esochinasi (HK). Valutando l aumento di assorbanza di NADP in forma ridotta a una lunghezza d onda di 340 nm, l attività relativa dell enzima viene calcolata in base alla seguente formula: 10
11 U E min V ml d 2 V dove: ΔE/min= variazione di estinzione al minuto V tot = volume finale della miscela di dosaggio (µl) V c = volume di campione da dosare aggiunto (µl) ε 340 = coefficiente di estinzione molare del NAD(P)H (6.3 mm -1 cm -1 ) d= cammino ottico della cuvetta (1 cm) Il valore di U/ml va diviso per 2 in base alla stechiometria della reazione. 340 tot c Allestire le miscele di dosaggio dell attività trealasica come mostrato in tabella [Wegener et al., 2003]: Reagenti Concentrazione iniziale Concentrazione finale Volume Na + -Acetato, ph mm 120 mm 240 µl MgCl mm 10 mm 100 µl NADP + 10 mm 0.6 mm 60 µl ATP 40 mm 1 mm 25 µl Trealosio 200 mm 20 mm 100 µl G6PDH 115 U/mL 0.84 U/mL 8 µl HK 133 U/mL 1.34 U/mL 10 µl campione x µl Acqua 457-x µl Volume totale 1000 µl N.B.: i dosaggi enzimatici vengono condotti ad una temperatura costante (T ambiente) e in condizioni definite di ph, utilizzando concentrazioni saturanti del substrato, il trealosio. Calcolare un volume di campione contenente 4 μg di proteina trealasi in base alla concentrazione proteica del campione purificato e dializzato: il volume calcolato 11
12 viene utilizzato per il dosaggio di attività enzimatica sia per la frazione solubile sia per la frazione purificata e dializzata. La reazione parte con l aggiunta del trealosio, che quindi viene aggiunto per ultimo alle miscele di reazione. - Registrare la variazione di assorbanza nel tempo (2-3 minuti). - Calcolare il ΔE/min e utilizzare la formula riportata sopra - Calcolare U/mL e completare la seguente tabella: Frazione solubile Frazione purificata e dializzata ΔE/min U/mL Completamento della tabella di purificazione Vol. [Prot] Proteine Attività Attività Attività Resa Indice Campione (ml) mg/ml totali U/mL totale specifica % di mg tot U tot U tot /mg tot purificazione Frazione solubile Enzima puro 12
13 IV GIORNO Cinetica enzimatica Sull enzima purificato viene effettuata la determinazione dei parametri cinetici K m e V max. Vengono condotte diverse misure di attività enzimatica come descritto precedentemente, ma a diverse concentrazioni di substrato (0.125, 0.25, 0.5, 1, 5, 10 mm). La miscela di dosaggio dell attività trealasica viene allestita come mostrato in tabella: Reagenti Concentrazione iniziale Concentrazione finale Volume Na + -Acetato, ph mm 120 mm 240 µl MgCl mm 10 mm 100 µl NADP + 10 mm 0.6 mm 60 µl ATP 40 mm 1 mm 25 µl Trealosio 1.25, 2.5, 5, 10, 50, 100 mm 0.125, 0.25, 0.5, 1, 5, 10 mm 100 µl G6PDH 115 U/mL 0.84 U/mL 8 µl HK 133 U/mL 1.34 U/mL 10 µl Enzima x µl Acqua 437-x µl Volume totale 1000 µl La quantità di enzima per condurre l esperimento di cinetica enzimatica viene calcolata sulla base dell attività enzimatica del campione purificato e dializzato, espressa in U/mL, in modo tale che contenga 10 mu. La reazione parte con l aggiunta del trealosio. - Registrare la variazione di assorbanza nel tempo (2-3 minuti). - Calcolare il ΔE/min e utilizzare la formula riportata sopra - Calcolare U/mL e completare la seguente tabella: reazioni [trealosio] E/min U/mL (v o ) 1/[S] 1/v o (min -1 ) (mm min -1 ) (mm -1 ) (min mm -1 ) 13
14 mm mm mm 4 1 mm 5 5 mm 6 10 mm I punti sperimentali possono essere interpolati con l equazione di un iperbole, secondo l equazione di Michaelis-Menten: v 0 V K max m S S Per il calcolo di Km e Vmax si allestisce il grafico dei doppi reciproci, riportando i valori dei reciproci delle velocità iniziali in funzione dei reciproci delle corrispondenti concentrazioni di substrato. L'equazione dei doppi reciproci ha la seguente forma: 1 Km 1 1 v V V S 0 max max Ricavare i valori dei parametri cinetici K M e V MAX. 14
15 Determinazione della massa molecolare della trealasi di C. riparius mediante SDS-PAGE Sul gel colorato e decolorato, si misura la distanza di migrazione di ciascuna delle proteine marker caricate. Si riporta in grafico il logaritmo della massa molecolare delle proteine standard in funzione della migrazione relativa, allestendo una retta di taratura. Da essa si ricava la massa molecolare della proteina di interesse, dopo aver misurato la sua distanza di migrazione. L analisi del gel permette anche di valutare qualitativamente l andamento della purificazione e il grado di purezza della proteina. Calcolo della concentrazione molare per il calcolo K cat Sulla base del peso molecolare calcolare la concentrazione molare della trealasi. Una volta ottenuta la concentrazione molare, procedere con il calcolo della costante catalitica in base alla relazione: V max = [E] tot K cat La k cat rappresenta una misura della formazione di prodotto per mezzo della catalisi enzimatica: rappresenta il numero di molecole di substrato trasformate da una molecola di enzima in un minuto (min -1 ). 15
16 V GIORNO Effetti del ph sull attività enzimatica Il ph è un parametro che influenza l attività enzimatica. Si procede pertanto ad effettuare dosaggi di attività trealasica sul campione purificato in presenza di tamponi a ph differenti. Si allestiscono tre cuvette secondo lo schema di dosaggio seguito il terzo giorno utilizzando i seguenti tamponi: - MES 500 mm ph 5 - Na + -acetato 500 mm ph Tris-HCl 500 mm ph 8 Si procede alla rilevazione dell assorbanza allo spettrofotometro e si calcolano U/mL. Con i dati attenuti si costruisce una curva di attività in relazione al ph esprimendo i dati di attività in percentuale rispetto all attività riscontrata all optimum di ph pari a 6.5. Saggi di denaturazione termica La temperatura è un fattore che influenza l attività enzimatica. Si procede pertanto ad effettuare dosaggi di attività trealasica sul campione purificato in seguito al trattamento a differenti temperature (30 C, 40 C, 50 C, 60 C). Dopo aver incubato il campione, si effettua il dosaggio secondo lo schema seguito il terzo giorno. Con i dati ottenuti si costruisce una curva di attività in relazione alla temperatura, riportando i dati in percentuale rispetto all attività monitorata senza incubazione alle differenti temperature stabilite. 16
CORSO DI LAUREA IN BIOLOGIA GENERALE ED APPLICATA ESERCITAZIONE DEL CORSO DI ANALISI DI MACROMOLECOLE
 CORSO DI LAUREA IN BIOLOGIA GENERALE ED APPLICATA ESERCITAZIONE DEL CORSO DI ANALISI DI MACROMOLECOLE Anno accademico 2016-2017 L esperimento consiste nella analisi di due estratti proteici frazionati
CORSO DI LAUREA IN BIOLOGIA GENERALE ED APPLICATA ESERCITAZIONE DEL CORSO DI ANALISI DI MACROMOLECOLE Anno accademico 2016-2017 L esperimento consiste nella analisi di due estratti proteici frazionati
ESERCIZI 2. Spiegare il principio di.
 ESERCIZI 2 Spiegare il principio di. Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione
ESERCIZI 2 Spiegare il principio di. Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione
ESERCIZI 2. Spiegare il principio di.
 ESERCIZI 2 Spiegare il principio di. Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione
ESERCIZI 2 Spiegare il principio di. Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione
Tra nm Nucleotidi mono-fosfato
 Un estratto proteico può contenere acidi nucleici, nucleotidi Posso verificare la loro presenza mediante la misura di uno spettro di assorbimento. Gli acidi nucleici e i nucleotidi assorbono nell UV, grazie
Un estratto proteico può contenere acidi nucleici, nucleotidi Posso verificare la loro presenza mediante la misura di uno spettro di assorbimento. Gli acidi nucleici e i nucleotidi assorbono nell UV, grazie
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole 1. Elettroforesi su gel di poliacrilammide (PAGE nativa) L esperimento consiste
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole 1. Elettroforesi su gel di poliacrilammide (PAGE nativa) L esperimento consiste
ε 340 nm A 260 nm A 260nm = 1.36 A 340 nm = A 340nm A = c [NADH] = : 6.22 x 10 3 = x 10-6 M
![ε 340 nm A 260 nm A 260nm = 1.36 A 340 nm = A 340nm A = c [NADH] = : 6.22 x 10 3 = x 10-6 M ε 340 nm A 260 nm A 260nm = 1.36 A 340 nm = A 340nm A = c [NADH] = : 6.22 x 10 3 = x 10-6 M](/thumbs/59/43508258.jpg) ESERCIZI 2 Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione molare a 260 nm sono
ESERCIZI 2 Esercizio n.1 Trovare la concentrazione di NAD e NADH in una soluzione miscelata basandosi sui seguenti dati ottenuti in una cuvetta da 1 cm : I coefficienti di estinzione molare a 260 nm sono
Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole
 Laboratorio Giorno 1 Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole Docente: Dott. Andrea Strazzulli Attività: Determinazione della concentrazione proteica di un campione. Parte
Laboratorio Giorno 1 Corso di Tecniche biomolecolari applicate Modulo Analisi di Macromolecole Docente: Dott. Andrea Strazzulli Attività: Determinazione della concentrazione proteica di un campione. Parte
- determinazione colorimetrica della concentrazione di una miscela proteica
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2013/2014 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2013/2014 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
Metodi di studio delle proteine :
 Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D determinazione del peso molecolare Spettrofotometro
Metodi di studio delle proteine : determinazione della quantità determinazione della struttura primaria (sequenza a.a.) determinazione della struttura 3D determinazione del peso molecolare Spettrofotometro
WESTERN BLOTTING Tecnica che permette di valutare quanto è espressa una proteina e dove è localizzata. SDS-PAGE trasferimento saturazione
 WESTERN BLOTTING Tecnica che permette di valutare quanto è espressa una proteina e dove è localizzata. La procedura di Western Blotting consiste nei seguenti passaggi: - SDS-PAGE - trasferimento (blotting)
WESTERN BLOTTING Tecnica che permette di valutare quanto è espressa una proteina e dove è localizzata. La procedura di Western Blotting consiste nei seguenti passaggi: - SDS-PAGE - trasferimento (blotting)
Analisi quantitative
 Analisi quantitative Diversi metodi per la quantificazione delle proteine totali (reazioni generali delle proteine): 1. Dosaggio spettrofotometrico diretto 2. Metodi colorimetrici 1. Dosaggio spettrofotometrico
Analisi quantitative Diversi metodi per la quantificazione delle proteine totali (reazioni generali delle proteine): 1. Dosaggio spettrofotometrico diretto 2. Metodi colorimetrici 1. Dosaggio spettrofotometrico
- determinazione colorimetrica della concentrazione di una miscela proteica. - Determinazione spettrofotometrica dell attività enzimatica
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2012/2013 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2012/2013 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
- determinazione colorimetrica della concentrazione di una miscela proteica
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2017/2018 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2017/2018 Laboratorio di Chimica Biologica - Cromatografia a scambio ionico - determinazione colorimetrica della concentrazione di una miscela
Metodologie di microbiologia e genetica
 Metodologie di microbiologia e genetica Modulo Metodologie di Genetica Prof.ssa Antonella Furini ESERCITAZIONE N 3 PREPARAZIONE DEL FRAMMENTO E DEL taglio con enzimi di restrizione 1. Taglio del plasmide
Metodologie di microbiologia e genetica Modulo Metodologie di Genetica Prof.ssa Antonella Furini ESERCITAZIONE N 3 PREPARAZIONE DEL FRAMMENTO E DEL taglio con enzimi di restrizione 1. Taglio del plasmide
RACCOLTA E PROCESSAMENTO CAMPIONE URINE PER ANALISI OMICHE
 RACCOLTA E PROCESSAMENTO CAMPIONE URINE PER ANALISI OMICHE 00049 Autori Vittorio Sirolli Contenuti Abstract raccolta e processamento dei campioni urinari Abstract release 1 pubblicata il 20 luglio 2012
RACCOLTA E PROCESSAMENTO CAMPIONE URINE PER ANALISI OMICHE 00049 Autori Vittorio Sirolli Contenuti Abstract raccolta e processamento dei campioni urinari Abstract release 1 pubblicata il 20 luglio 2012
ε (M -1 cm -1 ) Composto 260 nm 340 nm NAD ~ 0 NADPH
 1 Una soluzione contenente una miscela di NADPH e NAD + viene analizzata spettrofotometricamente. L assorbanza di tale miscela, misurata in una cuvetta da 1 cm di lunghezza, è pari a: A260 nm = 0,85 A340nm
1 Una soluzione contenente una miscela di NADPH e NAD + viene analizzata spettrofotometricamente. L assorbanza di tale miscela, misurata in una cuvetta da 1 cm di lunghezza, è pari a: A260 nm = 0,85 A340nm
Esercitazioni di Chimica Biologica AA PURIFICAZIONE DI UNA PROTEINA DI FUSIONE GST-CATECOLO 1,2 DIOSSIGENASI
 Esercitazioni di Chimica Biologica AA 2005-06 PURIFICAZIONE DI UNA PROTEINA DI FUSIONE GST-CATECOLO 1,2 DIOSSIGENASI Viene utilizzato il pellet cellulare congelato ottenuto dalla crescita di cellule di
Esercitazioni di Chimica Biologica AA 2005-06 PURIFICAZIONE DI UNA PROTEINA DI FUSIONE GST-CATECOLO 1,2 DIOSSIGENASI Viene utilizzato il pellet cellulare congelato ottenuto dalla crescita di cellule di
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2006/2007. Corso di: Laboratorio di Biologia II Chimica Biologica
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2006/2007 Corso di: Laboratorio di Biologia II Chimica Biologica Dott. Marcello MEROLA Parziale purificazione dell enzima Alcool Deidrogenasi
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2006/2007 Corso di: Laboratorio di Biologia II Chimica Biologica Dott. Marcello MEROLA Parziale purificazione dell enzima Alcool Deidrogenasi
Acido 3,5-dinitrosalicilico Glucosio Acido 3-amino-5-nitrosalicilico Acido gluconico (Incolore-giallo leggero)
 1 Determinazione di zuccheri riducenti e di amido in banana in relazione alle condizioni di conservazione (Corso di Fisiologia della produzione e della post-raccolta) In banana durante il processo di maturazione
1 Determinazione di zuccheri riducenti e di amido in banana in relazione alle condizioni di conservazione (Corso di Fisiologia della produzione e della post-raccolta) In banana durante il processo di maturazione
INGEZIM Gluten Quanti Test Guida all utilizzo
 INGEZIM Gluten Quanti Test Guida all utilizzo 1 - INTRODUZIONE Il kit è basato su un saggio immunoenzimatico ELISA di tipo sandwich come dettato dal Codex Alimentarius. Ogni pozzetto è rivestito sul fondo
INGEZIM Gluten Quanti Test Guida all utilizzo 1 - INTRODUZIONE Il kit è basato su un saggio immunoenzimatico ELISA di tipo sandwich come dettato dal Codex Alimentarius. Ogni pozzetto è rivestito sul fondo
Analisi quantitative
 Analisi quantitative Diversi metodi per la quantificazione delle proteine totali (reazioni generali delle proteine): 1. Dosaggio spettrofotometrico diretto 2. Metodi colorimetrici 1. Dosaggio spettrofotometrico
Analisi quantitative Diversi metodi per la quantificazione delle proteine totali (reazioni generali delle proteine): 1. Dosaggio spettrofotometrico diretto 2. Metodi colorimetrici 1. Dosaggio spettrofotometrico
La cinetica enzimatica: Calcolo della K m e V max
 La cinetica enzimatica: Calcolo della K m e V max Parametri cinetici K m (numericamente uguale alla concentrazione di substrato alla quale la velocità iniziale è pari alla metà della velocità massima)
La cinetica enzimatica: Calcolo della K m e V max Parametri cinetici K m (numericamente uguale alla concentrazione di substrato alla quale la velocità iniziale è pari alla metà della velocità massima)
Determinazione di zuccheri riducenti e di amido in banana in relazione alle condizioni di conservazione
 1 Determinazione di zuccheri riducenti e di amido in banana in relazione alle condizioni di conservazione In banana durante il processo di maturazione avviene la trasformazione di amido in zuccheri riducenti
1 Determinazione di zuccheri riducenti e di amido in banana in relazione alle condizioni di conservazione In banana durante il processo di maturazione avviene la trasformazione di amido in zuccheri riducenti
Laurea Magistrale in Biologia
 Laurea Magistrale in Biologia Laboratorio del corso di Chimica Fisica Biologica Anno accademico 2009/10 SCOPO Determinazione dei parametri termodinamici associati alla denaturazione termica di una piccola
Laurea Magistrale in Biologia Laboratorio del corso di Chimica Fisica Biologica Anno accademico 2009/10 SCOPO Determinazione dei parametri termodinamici associati alla denaturazione termica di una piccola
Schema a blocchi di uno spettrofluorimetro
 MONOCROMATORE EMISSIONE EM Schema a blocchi di uno spettrofluorimetro MONOCROMATORE ECCITAZIONE SORGENTE EXC RIVELATORE (TUBO FOTOMOLTIPLICATORE) Anche il DNA assorbe nell UV Cosa determina l assorbanza
MONOCROMATORE EMISSIONE EM Schema a blocchi di uno spettrofluorimetro MONOCROMATORE ECCITAZIONE SORGENTE EXC RIVELATORE (TUBO FOTOMOLTIPLICATORE) Anche il DNA assorbe nell UV Cosa determina l assorbanza
1. Cromatografia per gel-filtrazione
 1. Cromatografia per gel-filtrazione La gel filtrazione è un metodo cromatografico che separa le sostanze in base alle loro dimensioni molecolari. I materiali che costituiscono il supporto per la gel-filtrazione
1. Cromatografia per gel-filtrazione La gel filtrazione è un metodo cromatografico che separa le sostanze in base alle loro dimensioni molecolari. I materiali che costituiscono il supporto per la gel-filtrazione
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI
 VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
VALUTAZIONE DELLA BIODIVERSITÀ MICROBICA MEDIANTE L UTILIZZO DI TECNICHE MOLECOLARI Laboratorio di Genetica dei Microrganismi (Responsabile attività: Prof. Giovanni Salzano; Tutor: Dr.ssa Maria Grazia
Strategie per la purificazione delle proteine
 Strategie per la purificazione delle proteine In una cellula ci possono essere molte proteine (ad esempio, ci sono circa 4300 geni in E. coli) Una singola proteina può rappresentare una frazione tra lo
Strategie per la purificazione delle proteine In una cellula ci possono essere molte proteine (ad esempio, ci sono circa 4300 geni in E. coli) Una singola proteina può rappresentare una frazione tra lo
LISATI DA COLTURA. GRAM-: crescita overnight su nutrient agar a 28 c singole colonie sospese in 50µl di tampone di lisi (0.05m NAOH, 0.
 LISATI DA COLTURA. GRAM-: crescita overnight su nutrient agar a 28 c singole colonie sospese in 50µl di tampone di lisi (0.05m NAOH, 0.25% SDS) agitate su vortex per 60s trattate a 95 c per 15 min centrifugate
LISATI DA COLTURA. GRAM-: crescita overnight su nutrient agar a 28 c singole colonie sospese in 50µl di tampone di lisi (0.05m NAOH, 0.25% SDS) agitate su vortex per 60s trattate a 95 c per 15 min centrifugate
5- Estrazione ed analisi di DNA
 5- Estrazione ed analisi di DNA Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU) 16 ore) Marco Scocchi ( BIO/10-11) La struttura del DNA La traduzione dell informazione
5- Estrazione ed analisi di DNA Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU) 16 ore) Marco Scocchi ( BIO/10-11) La struttura del DNA La traduzione dell informazione
PROTOCOLLI del Lab di BIOLOGIA MOLECOLARE
 PROTOCOLLI del Lab di BIOLOGIA MOLECOLARE a.a. 2013/2014 GIORNO 1: A) Rivitalizzione dei batteri precedentemente trasformati con il vettore plasmidico di interesse B) Induzione con IPTG dell'espressione
PROTOCOLLI del Lab di BIOLOGIA MOLECOLARE a.a. 2013/2014 GIORNO 1: A) Rivitalizzione dei batteri precedentemente trasformati con il vettore plasmidico di interesse B) Induzione con IPTG dell'espressione
Biochimica Analitica Esperienze di Laboratorio
 Corso di Laurea in Biotecnologie Insegnamento di Biochimica e Biochimica Analitica Dipartimento di Biotecnologie Università degli Studi di Verona Anno Accademico 2017-2018 Biochimica Analitica Esperienze
Corso di Laurea in Biotecnologie Insegnamento di Biochimica e Biochimica Analitica Dipartimento di Biotecnologie Università degli Studi di Verona Anno Accademico 2017-2018 Biochimica Analitica Esperienze
Prof. Maria Nicola GADALETA
 Prof. Maria Nicola GADALETA E-mail: m.n.gadaleta@biologia.uniba.it Facoltà di Scienze Biotecnologiche Corso di Laurea in Biotecnologie Sanitarie e Farmaceutiche Biochimica e Tecnologie Biochimiche DISPENSA
Prof. Maria Nicola GADALETA E-mail: m.n.gadaleta@biologia.uniba.it Facoltà di Scienze Biotecnologiche Corso di Laurea in Biotecnologie Sanitarie e Farmaceutiche Biochimica e Tecnologie Biochimiche DISPENSA
Elettroforesi = migrazione in campo elettrico
 Elettroforesi = migrazione in campo elettrico Molte molecole di interesse biologico possiedono gruppi ionizzabili. In opportune condizioni di ph presentano una carica elettrica + o e quindi sono in grado
Elettroforesi = migrazione in campo elettrico Molte molecole di interesse biologico possiedono gruppi ionizzabili. In opportune condizioni di ph presentano una carica elettrica + o e quindi sono in grado
VALUTAZIONE DELLA INTERAZIONE TRA ANTIGENE E ANTICORPO MEDIANTE TECNICA IMMUNOCHIMICA WESTERN BLOTTING
 VALUTAZIONE DELLA INTERAZIONE TRA ANTIGENE E ANTICORPO MEDIANTE TECNICA IMMUNOCHIMICA WESTERN BLOTTING Laboratorio di Biochimica Cellulare (Responsabile attività: Prof. Angela Ostuni; Tutor: Dr.ssa Agata
VALUTAZIONE DELLA INTERAZIONE TRA ANTIGENE E ANTICORPO MEDIANTE TECNICA IMMUNOCHIMICA WESTERN BLOTTING Laboratorio di Biochimica Cellulare (Responsabile attività: Prof. Angela Ostuni; Tutor: Dr.ssa Agata
LABORATORIO DI GENETICA SUMMER SCHOOL 2014
 P a g i n a 1 LABORATORIO DI GENETICA SUMMER SCHOOL 2014 PROTOCOLLO DI ESTRAZIONE DEL DNA DA TESSUTI VEGETALI GenElute Plant Genomic DNA Miniprep Kit (Sigma-Aldrich) https://www.google.it/url?sa=t&rct=j&q=&esrc=s&source=web&cd=2&cad=rja&uact=8&ved=0c
P a g i n a 1 LABORATORIO DI GENETICA SUMMER SCHOOL 2014 PROTOCOLLO DI ESTRAZIONE DEL DNA DA TESSUTI VEGETALI GenElute Plant Genomic DNA Miniprep Kit (Sigma-Aldrich) https://www.google.it/url?sa=t&rct=j&q=&esrc=s&source=web&cd=2&cad=rja&uact=8&ved=0c
Determinazioni quantitative in farmacologia
 Determinazioni quantitative in farmacologia 1 Dosaggio biologico determinazione quantitativa della concentrazione o della potenza di una sostanza per mezzo della valutazione della risposta biologica che
Determinazioni quantitative in farmacologia 1 Dosaggio biologico determinazione quantitativa della concentrazione o della potenza di una sostanza per mezzo della valutazione della risposta biologica che
XVII ZOLFO. Metodo XVII.1 DETERMINAZIONE DELLO ZOLFO TOTALE. Metodo XVII.2 DETERMINAZIONE DELLO ZOLFO DA SOLFATI
 XVII ZOLFO Metodo XVII.1 DETERMINAZIONE DELLO ZOLFO TOTALE Metodo XVII.2 DETERMINAZIONE DELLO ZOLFO DA SOLFATI XVII - ZOLFO Metodo XVII.1 DETERMINAZIONE DELLO ZOLFO TOTALE 1. Principio Il campione viene
XVII ZOLFO Metodo XVII.1 DETERMINAZIONE DELLO ZOLFO TOTALE Metodo XVII.2 DETERMINAZIONE DELLO ZOLFO DA SOLFATI XVII - ZOLFO Metodo XVII.1 DETERMINAZIONE DELLO ZOLFO TOTALE 1. Principio Il campione viene
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli. I nostri inquilini invisibili
 Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
Laboratorio di Microbiologia Referente: Prof. Carla Vignaroli I nostri inquilini invisibili Viviamo quotidianamente in compagnia di una moltitudine invisibile di microrganismi, che si trova nell ambiente
Analisi spettrofotometrica dell Albumina di siero bovino: applicazione della legge di Lambert-Beer:
 Analisi spettrofotometrica dell Albumina di siero bovino: applicazione della legge di Lambert-Beer: Introduzione: La spettrofotometria o fotometria è una tecnica analitica che sfrutta il fenomeno dell'assorbimento
Analisi spettrofotometrica dell Albumina di siero bovino: applicazione della legge di Lambert-Beer: Introduzione: La spettrofotometria o fotometria è una tecnica analitica che sfrutta il fenomeno dell'assorbimento
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI
 METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
METODI DI BASE PER L ANALISI DEGLI ACIDI NUCLEICI SPETTRI UV E QUANTIZZAZIONE SPETTROFOTOMETRICA DEGLI ACIDI NUCLEICI FIGURA 7.6A METODOLOGIA BIOCHIMICA (WILSON) a a = a 1 Assorbimento = a b a 1 b FIGURA
Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa
 WESTERN BLOTTING Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa Tecnica di trasferimento delle proteine da un gel ad una membrana associata ad utilizzo di anticorpi
WESTERN BLOTTING Permette l identificazione di specifiche proteine antigeniche in una soluzione complessa Tecnica di trasferimento delle proteine da un gel ad una membrana associata ad utilizzo di anticorpi
ELETTROFORESI DI PROTEINE
 ELETTROFORESI DI PROTEINE ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni
ELETTROFORESI DI PROTEINE ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni
Analisi delle Proteine
 Analisi delle Proteine Elementi che costituiscono le proteine: idrogeno, carbonio, azoto, ossigeno e zolfo; tra cui l azoto è l elemento più distintivo nei cibi, l azoto varia da 13,4% a 19,1%. L analisi
Analisi delle Proteine Elementi che costituiscono le proteine: idrogeno, carbonio, azoto, ossigeno e zolfo; tra cui l azoto è l elemento più distintivo nei cibi, l azoto varia da 13,4% a 19,1%. L analisi
Scientists with lab coats: Corso di laboratorio Chimico-Biologico. Dott.ssa Valeria Berton Università di Verona
 Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Come «vedere» il DNA: la PCR PCR = Polymerase Chain Reaction (reazione a catena della polimerasi)
Scientists with lab coats: Corso di laboratorio Chimico-Biologico Dott.ssa Valeria Berton Università di Verona Come «vedere» il DNA: la PCR PCR = Polymerase Chain Reaction (reazione a catena della polimerasi)
SAFE - Scuola di Scienze Agrarie, Forestali, Alimentari ed Ambientali. School of Agriculture, forest, Food and Environmental sciences.
 Protocollo SAFE Protocollo di estrazione DNA da colture di fermenti lattici (metodo del salting-out; Dr. Lara Aponte) Reagenti: Tampone STE (per lavaggio delle cellule) Tampone di lisi Tampone TE (per
Protocollo SAFE Protocollo di estrazione DNA da colture di fermenti lattici (metodo del salting-out; Dr. Lara Aponte) Reagenti: Tampone STE (per lavaggio delle cellule) Tampone di lisi Tampone TE (per
Espressione eterologa, purificazione e saggio di attività della Fe-idrogenasi di Enterobacter cloacae
 Università degli studi di Padova - Facoltà di scienze MM.FF.NN. Laurea di primo livello in biologia molecolare Espressione eterologa, purificazione e saggio di attività della Fe-idrogenasi di Enterobacter
Università degli studi di Padova - Facoltà di scienze MM.FF.NN. Laurea di primo livello in biologia molecolare Espressione eterologa, purificazione e saggio di attività della Fe-idrogenasi di Enterobacter
Scheda n.15. Vania Bresolin Cinetica enzimatica. Determinazione dell attività specifica di un enzima
 Scheda n.15 Vania Bresolin Cinetica enzimatica Determinazione dell attività specifica di un enzima ITAS Santorre di Santarosa Istituto Tecnico, settore Tecnologico Indirizzo Chimica, Materiali e Biotecnologie
Scheda n.15 Vania Bresolin Cinetica enzimatica Determinazione dell attività specifica di un enzima ITAS Santorre di Santarosa Istituto Tecnico, settore Tecnologico Indirizzo Chimica, Materiali e Biotecnologie
Analisi omiche su sangue e urine
 Analisi omiche su sangue e urine 00051 Autori Vittorio Sirolli Massimo Papale (redattore) Gianluigi Zaza (redattore) Francesco Trepiccione (collaboratore) Contenuti 1. Abstract 2. Sangue Separazione Plasma
Analisi omiche su sangue e urine 00051 Autori Vittorio Sirolli Massimo Papale (redattore) Gianluigi Zaza (redattore) Francesco Trepiccione (collaboratore) Contenuti 1. Abstract 2. Sangue Separazione Plasma
CINETICA ENZIMATICA - velocità di reazione
 [P] Velocità iniziale (V 0 ) CINETICA ENZIMATICA - velocità di reazione + + k 1 E + S ES ES E + P k 2 k 3 k 4 V = d[p]/dt = k 3 [ES] Velocità iniziale (V 0 ) quando [S] >> [P] V max t CINETICA ENZIMATICA:
[P] Velocità iniziale (V 0 ) CINETICA ENZIMATICA - velocità di reazione + + k 1 E + S ES ES E + P k 2 k 3 k 4 V = d[p]/dt = k 3 [ES] Velocità iniziale (V 0 ) quando [S] >> [P] V max t CINETICA ENZIMATICA:
MA + H + MAH + Materiale: 500 cc di soluzione di NaOH 0.01 M 500 cc di soluzione di HCl 0.01 M fresca Metil-arancio (MA) Etanolo Acqua distillata
 Cambiamenti nell aspetto degli spettri di assorbimento elettronico del metilarancio in funzione del ph a seguito della reazione di protonazione del metilarancio. Scopo dell esperimento è osservare la variazione
Cambiamenti nell aspetto degli spettri di assorbimento elettronico del metilarancio in funzione del ph a seguito della reazione di protonazione del metilarancio. Scopo dell esperimento è osservare la variazione
PROGETTO di ALTERNANZA SCUOLA LAVORO
 PROGETTO di ALTERNANZA SCUOLA LAVORO Formazione sul rischio specifico chimico e biologico Progettazione e sintesi di composti organici di interesse farmaceutico; determinazione quali-quantitativa Attività
PROGETTO di ALTERNANZA SCUOLA LAVORO Formazione sul rischio specifico chimico e biologico Progettazione e sintesi di composti organici di interesse farmaceutico; determinazione quali-quantitativa Attività
Analisi quantitativa dell interazione proteina-proteina
 Analisi quantitativa dell interazione proteina-proteina Legame recettore-ligando (proteina-proteina) Il legame per il ligando deve essere: reversibile ad alta affinità saturabile deve portare ad una risposta
Analisi quantitativa dell interazione proteina-proteina Legame recettore-ligando (proteina-proteina) Il legame per il ligando deve essere: reversibile ad alta affinità saturabile deve portare ad una risposta
Quantizzazione del DNA con spettrofotometro
 Quantizzazione del DNA ed elettroforesi 1 Quantizzazione del DNA con spettrofotometro Al termine della metodica di estrazione, occorre quantificare il DNA isolato. Un metodo per quantificare in modo preciso
Quantizzazione del DNA ed elettroforesi 1 Quantizzazione del DNA con spettrofotometro Al termine della metodica di estrazione, occorre quantificare il DNA isolato. Un metodo per quantificare in modo preciso
PROTOCOLLO RACCOLTA SANGUE PER ANALISI OMICHE
 PROTOCOLLO RACCOLTA SANGUE PER ANALISI OMICHE 00028 Autori Vittorio Sirolli Gianluigi Zaza Contenuti Premesse - Introduzione separazione del plasma Premesse - Introduzione separazione del plasma release
PROTOCOLLO RACCOLTA SANGUE PER ANALISI OMICHE 00028 Autori Vittorio Sirolli Gianluigi Zaza Contenuti Premesse - Introduzione separazione del plasma Premesse - Introduzione separazione del plasma release
+ (ph < pi) 0/- (ph pi)
 Pianificare la separazione delle seguenti proteine utilizzando una colonna cromatografica contenente lo scambiatore di cationi CM-cellulosa. A quale valore di ph dobbiamo equilibrare la FS? Stabilire la
Pianificare la separazione delle seguenti proteine utilizzando una colonna cromatografica contenente lo scambiatore di cationi CM-cellulosa. A quale valore di ph dobbiamo equilibrare la FS? Stabilire la
WESTERN BLOT o IMMUNOFISSAZIONE
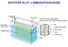 WESTERN BLOT o IMMUNOFISSAZIONE BLOTTING Trasferimento di macromolecole su una membrana immobilizzante. Southern DNA - da Edward Southern 1970 Northern Western RNA Proteine SCOPO DEL WESTERN BLOTTING Consente
WESTERN BLOT o IMMUNOFISSAZIONE BLOTTING Trasferimento di macromolecole su una membrana immobilizzante. Southern DNA - da Edward Southern 1970 Northern Western RNA Proteine SCOPO DEL WESTERN BLOTTING Consente
O (PM:266,45), resina Dowex AG50W-X2, acido perclorico 65%.
 Parte sperimentale Reagenti CrCl 3 x 6H 2 O (PM:266,45), resina Dowex AG50W-X2, acido perclorico 65%. Procedura sperimentale Preparazione delle soluzioni eluenti Preparare le seguenti soluzioni madre di
Parte sperimentale Reagenti CrCl 3 x 6H 2 O (PM:266,45), resina Dowex AG50W-X2, acido perclorico 65%. Procedura sperimentale Preparazione delle soluzioni eluenti Preparare le seguenti soluzioni madre di
Spettroscopia nell ultravioletto e nel visibile
 Spettroscopia nell ultravioletto e nel visibile Sono le regioni dello spettro elettromagnetico maggiormente utilizzate nelle ricerche in campo biomedico Le radiazioni elettromagnetiche e lo spettro Una
Spettroscopia nell ultravioletto e nel visibile Sono le regioni dello spettro elettromagnetico maggiormente utilizzate nelle ricerche in campo biomedico Le radiazioni elettromagnetiche e lo spettro Una
v = Δ[P] = _ Δ[S] S P
![v = Δ[P] = _ Δ[S] S P v = Δ[P] = _ Δ[S] S P](/thumbs/98/137173050.jpg) CINETICA ENZIMATICA Gli esperimenti di cinetica enzimatica si basano sulla determinazione della velocità di reazione, misurata come variazione della concentrazione del prodotto o del reagente in un dato
CINETICA ENZIMATICA Gli esperimenti di cinetica enzimatica si basano sulla determinazione della velocità di reazione, misurata come variazione della concentrazione del prodotto o del reagente in un dato
CFU. Nome e cognome. Metodi Biochimici in Biotecnologie 19 luglio 2AA7- Test B
 Metodi Biochimici in Biotecnologie 19 luglio 2AA7- Test B Nome e cognome CFU 1B) (3 punti max) L'enzima YZ contiene un atomo di rame nel suo sito attivo. Lo spettro di assorbimento del visibile di questo
Metodi Biochimici in Biotecnologie 19 luglio 2AA7- Test B Nome e cognome CFU 1B) (3 punti max) L'enzima YZ contiene un atomo di rame nel suo sito attivo. Lo spettro di assorbimento del visibile di questo
GENERALITA SULLA CROMATOGRAFIA
 GENERALITA SULLA CROMATOGRAFIA COSA SONO LE TECNICHE CROMATOGRAFICHE? La cromatografia consiste nello sfruttare la diversa attitudine di ogni molecola o ione nel distribuirsi fra 2 fasi differenti. Una
GENERALITA SULLA CROMATOGRAFIA COSA SONO LE TECNICHE CROMATOGRAFICHE? La cromatografia consiste nello sfruttare la diversa attitudine di ogni molecola o ione nel distribuirsi fra 2 fasi differenti. Una
Estrazione di acido salicilico con acetato di n-butile
 S.A.G.T. Anno Accademico 2009/2010 Laboratorio Ambientale II A Dottoressa Valentina Gianotti Marco Soda Matricola num. 10015062 Estrazione di acido salicilico con acetato di n-butile L estrazione liquido-liquido
S.A.G.T. Anno Accademico 2009/2010 Laboratorio Ambientale II A Dottoressa Valentina Gianotti Marco Soda Matricola num. 10015062 Estrazione di acido salicilico con acetato di n-butile L estrazione liquido-liquido
Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva.
 OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
OBIETTIVO DEL LAVORO SPERIMENTALE: TIPIZZAZIONE DEI CROMOSOMI SESSUALI X e Y Amplificazione mediante PCR (Polymerase Chain Reaction) del DNA estratto dalla saliva. Cromosoma X Cromosoma y La tipizzazione
concentrazione finale rispettivamente di 0.2 M e?%,a 55.c 60 minuti.
 SEPARAZIONE DELLE SUBUNITA' DELLA RIBULOSIO-1,5-DIFOSFATO CARBOSSILASI DA FOGLIE DI CARCIOFO. Cardinali A.,Miceli A.,Pacoda D. e Gallerani R. Centro di Biologia,Laboratorio di Chimica Biologica,Universita'
SEPARAZIONE DELLE SUBUNITA' DELLA RIBULOSIO-1,5-DIFOSFATO CARBOSSILASI DA FOGLIE DI CARCIOFO. Cardinali A.,Miceli A.,Pacoda D. e Gallerani R. Centro di Biologia,Laboratorio di Chimica Biologica,Universita'
LABORATORIO DI CHIMICA FISICA I. Dr Ester Chiessi
 LABORATORIO DI CHIMICA FISICA I. Dr Ester Chiessi STUDIO CINETICO DELLA REAZIONE DI IDROLISI DELL ACETATO DI p- NITROFENILE IN SOLUZIONE ACQUOSA ALCALINA La reazione di idrolisi dell acetato di p-nitrofenile
LABORATORIO DI CHIMICA FISICA I. Dr Ester Chiessi STUDIO CINETICO DELLA REAZIONE DI IDROLISI DELL ACETATO DI p- NITROFENILE IN SOLUZIONE ACQUOSA ALCALINA La reazione di idrolisi dell acetato di p-nitrofenile
3. ELETTROFORESI DI PROTEINE
 3. ELETTROFORESI DI PROTEINE Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU-16 ore) Marco Scocchi ( BIO/10-11) ELETTROFORESI PRINCIPI GENERALI Migrazione di particelle
3. ELETTROFORESI DI PROTEINE Corso 240/350: Didattica di biochimica e biologia molecolare con laboratorio (2 CFU-16 ore) Marco Scocchi ( BIO/10-11) ELETTROFORESI PRINCIPI GENERALI Migrazione di particelle
La Spettroscopia in Biologia
 La Spettroscopia in Biologia Linda Avesani Dip. Scientifico e Tecnologico Università di Verona Spettroscopia e Proprietà della luce La spettroscopia in biologia studia la struttura e la dinamica delle
La Spettroscopia in Biologia Linda Avesani Dip. Scientifico e Tecnologico Università di Verona Spettroscopia e Proprietà della luce La spettroscopia in biologia studia la struttura e la dinamica delle
FOSFORO REATTIVO. metodo colorimetrico 0,45 0,40 0,35 0,30 0,25 0,20 0,15 0,10 0,05
 1 Consiglio Nazionale delle Ricerche Istituto per lo Studio degli Ecosistemi Verbania Pallanza Laboratorio di idrochimica - metodi analitici ad uso interno a cura di Gabriele TARTARI FOSFORO REATTIVO metodo
1 Consiglio Nazionale delle Ricerche Istituto per lo Studio degli Ecosistemi Verbania Pallanza Laboratorio di idrochimica - metodi analitici ad uso interno a cura di Gabriele TARTARI FOSFORO REATTIVO metodo
cognome e nome turno banco data Indicare i CALCOLI per ESTESO Nei RIQUADRI TRATTEGGIATI riportare i dati con le CIFRE SIGNIFICATIVE appropriate
 PREPARAZIONE e STANDARDIZZAZIONE di una SOLUZIONE di ACIDO CLORIDRICO densità HCl (aq) conc. (g/ml) % peso prelievo HCl (aq) conc. (ml) mmoli HCl = volume matraccio (ml) titolo previsto HCl (aq) diluito
PREPARAZIONE e STANDARDIZZAZIONE di una SOLUZIONE di ACIDO CLORIDRICO densità HCl (aq) conc. (g/ml) % peso prelievo HCl (aq) conc. (ml) mmoli HCl = volume matraccio (ml) titolo previsto HCl (aq) diluito
Elettroforesi di Tamara Benincasa
 Elettroforesi di Tamara Benincasa L elettroforesi è una metodologia utilizzata in laboratorio attraverso la quale molecole biologiche (ammino acidi, peptidi, proteine, nucleotidi ed acidi nucleici), dotate
Elettroforesi di Tamara Benincasa L elettroforesi è una metodologia utilizzata in laboratorio attraverso la quale molecole biologiche (ammino acidi, peptidi, proteine, nucleotidi ed acidi nucleici), dotate
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2007/2008. Corso di: Laboratorio di Biologia II Chimica Biologica
 CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2007/2008 Corso di: Laboratorio di Biologia II Chimica Biologica Dott. Marcello MEROLA Parziale purificazione dell enzima Alcool Deidrogenasi
CORSO DI LAUREA IN BIOLOGIA GENERALE E APPLICATA Anno Accademico 2007/2008 Corso di: Laboratorio di Biologia II Chimica Biologica Dott. Marcello MEROLA Parziale purificazione dell enzima Alcool Deidrogenasi
Estrazione del DNA. A)Resine (Chelex 100) B) Solven: organici. C) Sali (sal:ng out)
 Estrazione del DNA A)Resine (Chelex 100) B) Solven: organici C) Sali (sal:ng out) La Chelex 100 è una resina cara4erizzata da biglie a carica nega:va (ioni imminoaceta: contenu: nei copolimeri di s:rene
Estrazione del DNA A)Resine (Chelex 100) B) Solven: organici C) Sali (sal:ng out) La Chelex 100 è una resina cara4erizzata da biglie a carica nega:va (ioni imminoaceta: contenu: nei copolimeri di s:rene
Dosaggio delle proteine
 Laboratorio Integrato 4 Lezione n.2 Dosaggio delle proteine Dott.ssa Francesca Zazzeroni METODI PER EFFETTUARE UN DOSAGGIO PROTEICO 1-Metodo del Biureto - il reattivo del biureto consiste in una soluzione
Laboratorio Integrato 4 Lezione n.2 Dosaggio delle proteine Dott.ssa Francesca Zazzeroni METODI PER EFFETTUARE UN DOSAGGIO PROTEICO 1-Metodo del Biureto - il reattivo del biureto consiste in una soluzione
IL BINDING RECETTORIALE VIENE EFFETTUATO ATTRAVERSO L UTILIZZO DI LIGANDI SELETTIVI MARCATI CON UN RADIOISOTOPO E CI PERMETTE:
 IL BINDING RECETTORIALE VIENE EFFETTUATO ATTRAVERSO L UTILIZZO DI LIGANDI SELETTIVI MARCATI CON UN RADIOISOTOPO E CI PERMETTE: Ø VALUTAZIONE DELLA PRESENZA DI SITI DI RICONOSCIMENTO SPECIFICI PER UN LIGANDO
IL BINDING RECETTORIALE VIENE EFFETTUATO ATTRAVERSO L UTILIZZO DI LIGANDI SELETTIVI MARCATI CON UN RADIOISOTOPO E CI PERMETTE: Ø VALUTAZIONE DELLA PRESENZA DI SITI DI RICONOSCIMENTO SPECIFICI PER UN LIGANDO
BENZILPENICILLINA BENZATINICA PREPARAZIONE INIETTABILI. Benzilpenicillina Benzatinica polvere sterile per preparazioni iniettabili
 1 1 1 1 1 1 1 1 0 1 0 1 0 1 1 01/FU Maggio 00 Commenti entro il 0 Settembre 00 NOTA: Corretta in seguito ai commenti della Dott.ssa Mozzetti La monografia è stata revisionata per armonizzarla con le altre
1 1 1 1 1 1 1 1 0 1 0 1 0 1 1 01/FU Maggio 00 Commenti entro il 0 Settembre 00 NOTA: Corretta in seguito ai commenti della Dott.ssa Mozzetti La monografia è stata revisionata per armonizzarla con le altre
Determinazione del contenuto di licopene
 Determinazione del contenuto di licopene (Corso di Fisiologia della produzione e della post-raccolta) Il colore arancione, giallo e rosso di foglie, fiori e frutti è dovuto principalmente alla presenza
Determinazione del contenuto di licopene (Corso di Fisiologia della produzione e della post-raccolta) Il colore arancione, giallo e rosso di foglie, fiori e frutti è dovuto principalmente alla presenza
Laboratorio 29.1 CINETICA DI IDROLISI ALCALINA DELL ACETATO DI ETILE
 2 Analisi chimica strumentale Laboratorio 29.1 CINETICA DI IDROLISI ALCALINA DELL ACETATO DI ETILE SCOPO Determinazione della costante di velocità, dell energia di attivazione e del fattore di frequenza
2 Analisi chimica strumentale Laboratorio 29.1 CINETICA DI IDROLISI ALCALINA DELL ACETATO DI ETILE SCOPO Determinazione della costante di velocità, dell energia di attivazione e del fattore di frequenza
Estrazione rapida di DNA plasmidico da Escherichia coli. (QIAscreen) ESERCITAZIONE DI LAB. N.1. Corso di INGEGNERIA GENETICA, Prof.
 Estrazione rapida di DNA plasmidico da Escherichia coli (QIAscreen) Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.1 PLASMIDI IN NUMEROSE SPECIE BATTERICHE SONO PRESENTI I PLASMIDI,
Estrazione rapida di DNA plasmidico da Escherichia coli (QIAscreen) Corso di INGEGNERIA GENETICA, Prof. Renato Fani, ESERCITAZIONE DI LAB. N.1 PLASMIDI IN NUMEROSE SPECIE BATTERICHE SONO PRESENTI I PLASMIDI,
cromatografia Elettroforesi
 Cromatografia ed Elettroforesi Prof.ssa Flavia Frabetti Esplorando le proteine Un passaggio indispensabile negli studi di biochimica che necessitano di conoscere la sequenza aa di una proteina per potere
Cromatografia ed Elettroforesi Prof.ssa Flavia Frabetti Esplorando le proteine Un passaggio indispensabile negli studi di biochimica che necessitano di conoscere la sequenza aa di una proteina per potere
RIDASCREEN CIS. Test immunoenzimatico per la rilevazione del latte vaccino nel latte ovino e caprino e nel formaggio ovino e caprino. Art. No.
 RIDASCREEN CIS Test immunoenzimatico per la rilevazione del latte vaccino nel latte ovino e caprino e nel formaggio ovino e caprino Art. No.: R4302 Test in vitro Conservare a 2-8 C R-Biopharm AG, Darmstadt,
RIDASCREEN CIS Test immunoenzimatico per la rilevazione del latte vaccino nel latte ovino e caprino e nel formaggio ovino e caprino Art. No.: R4302 Test in vitro Conservare a 2-8 C R-Biopharm AG, Darmstadt,
Analisi e purificazione di proteine
 Analisi e purificazione di proteine elettroforesi ultracentrifugazione cromatografia frammenti specifici sequenziamento struttura primaria livelli superiori contesto fisiologico funzione Condizioni denaturanti
Analisi e purificazione di proteine elettroforesi ultracentrifugazione cromatografia frammenti specifici sequenziamento struttura primaria livelli superiori contesto fisiologico funzione Condizioni denaturanti
Test ELISA (enzyme-linked immunosorbent assay)
 PLS-BIOTECNOLOGIE Dipartimento di Scienze ATTIVITA LABORATORIALE 2017-2018 Test ELISA (enzyme-linked immunosorbent assay) Determinazione della presenza/assenza di un Antigene INTRODUZIONE ELISA è l'acronimo
PLS-BIOTECNOLOGIE Dipartimento di Scienze ATTIVITA LABORATORIALE 2017-2018 Test ELISA (enzyme-linked immunosorbent assay) Determinazione della presenza/assenza di un Antigene INTRODUZIONE ELISA è l'acronimo
ELETTROFORESI DI PROTEINE
 ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni di PROTEINE è più piccola
ELETTROFORESI DI PROTEINE Molto più complessa della separazione elettroforetica di DNA. forma delle proteine fortissime variazioni cariche delle proteine La > parte dei campioni di PROTEINE è più piccola
k 2 per una reazione semplice ad uno stadio corrisponde alla k cat (costante catalitica)
 Costante catalitica o n di Turnover k 1 k 2 S + E ES E + P k -1 Vmax = k 2 [ES] = k 2 [E] TOT k Vmax 2 = [E] TOT k 2 per una reazione semplice ad uno stadio corrisponde alla k cat (costante catalitica)
Costante catalitica o n di Turnover k 1 k 2 S + E ES E + P k -1 Vmax = k 2 [ES] = k 2 [E] TOT k Vmax 2 = [E] TOT k 2 per una reazione semplice ad uno stadio corrisponde alla k cat (costante catalitica)
DOSAGGIO ACIDI NUCLEICI
 DOSAGGIO ACIDI NUCLEICI l Cuvetta contenente il campione di proteina a concentrazione C Sorgente I 0 I 1 I 0 Rivelatore Quando si fa passare una luce di una particolare lunghezza d onda d
DOSAGGIO ACIDI NUCLEICI l Cuvetta contenente il campione di proteina a concentrazione C Sorgente I 0 I 1 I 0 Rivelatore Quando si fa passare una luce di una particolare lunghezza d onda d
Rapporto tra soluto e solvente o soluzione
 Programma Misure ed Unità di misura. Incertezza della misura. Cifre significative. Notazione scientifica. Atomo e peso atomico. Composti, molecole e ioni. Formula molecolare e peso molecolare. Mole e massa
Programma Misure ed Unità di misura. Incertezza della misura. Cifre significative. Notazione scientifica. Atomo e peso atomico. Composti, molecole e ioni. Formula molecolare e peso molecolare. Mole e massa
Estrazione di proteine da matrici alimentari, determinazione ed analisi
 Estrazione di proteine da matrici alimentari, determinazione ed analisi Laboratorio di Biochimica Generale (Responsabile attività: Prof. Rocco Rossano, Tutor: Dr.ssa Marilena Larocca) 1. Omogeneizzazione
Estrazione di proteine da matrici alimentari, determinazione ed analisi Laboratorio di Biochimica Generale (Responsabile attività: Prof. Rocco Rossano, Tutor: Dr.ssa Marilena Larocca) 1. Omogeneizzazione
VITTORIO EMANUELE II & SCUOLAVIVA PRESENTANO
 VITTORIO EMANUELE II & SCUOLAVIVA PRESENTANO ANTICORPI MONOCLONALI: NUOVI FARMACI A BERSAGLIO MOLECOLARE ANTICORPO PROTEINE,costituite da 4 catene polipeptidiche uguali a due a due : 2 catene pesanti e
VITTORIO EMANUELE II & SCUOLAVIVA PRESENTANO ANTICORPI MONOCLONALI: NUOVI FARMACI A BERSAGLIO MOLECOLARE ANTICORPO PROTEINE,costituite da 4 catene polipeptidiche uguali a due a due : 2 catene pesanti e
Trattamento quantitativo della complessazione e della reattività nei sistemi supramolecolari
 Trattamento quantitativo della complessazione e della reattività nei sistemi supramolecolari Assunzione di principio: i sistemi si trovano in condizioni di controllo termodinamico e non cinetico (tutti
Trattamento quantitativo della complessazione e della reattività nei sistemi supramolecolari Assunzione di principio: i sistemi si trovano in condizioni di controllo termodinamico e non cinetico (tutti
AMPICILLINA SODICA PREPARAZIONE INIETTABILE. Ampicillina sodica polvere sterile per preparazioni iniettabili
 1 0 1 0 1 0 1 0 1 001/FU Aprile 00 Commenti entro il Settembre 00 NOTA: Armonizzata con la versione revisionata della B.P. La monografia è stata completamente revisionata per armonizzarla con le corrispondenti
1 0 1 0 1 0 1 0 1 001/FU Aprile 00 Commenti entro il Settembre 00 NOTA: Armonizzata con la versione revisionata della B.P. La monografia è stata completamente revisionata per armonizzarla con le corrispondenti
Se il sito attivo fosse perfettamente complementare al substrato la reazione non procederebbe. Substrato
 Se il sito attivo fosse perfettamente complementare al substrato la reazione non procederebbe Substrato Nelson Cox, I PRINCIPI DI BIOCHIMICA DI LEHNINGER, Zanichelli editore S.p.A. Copyright 2014 6 11
Se il sito attivo fosse perfettamente complementare al substrato la reazione non procederebbe Substrato Nelson Cox, I PRINCIPI DI BIOCHIMICA DI LEHNINGER, Zanichelli editore S.p.A. Copyright 2014 6 11
RISOLUZIONE ENO 25/2004
 DICARBONATO DI DIMETILE (DMDC) L'ASSEMBLEA GENERALE, Visto l'articolo 2 paragrafo 2 iv dell'accordo del 3 aprile 2001 che istituisce l'organizzazione internazionale della vigna e del vino Su proposta della
DICARBONATO DI DIMETILE (DMDC) L'ASSEMBLEA GENERALE, Visto l'articolo 2 paragrafo 2 iv dell'accordo del 3 aprile 2001 che istituisce l'organizzazione internazionale della vigna e del vino Su proposta della
SCOPO DEL WESTERN BLOTTING
 WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING Tecnica che consente di visualizzare/individuare UNA proteina tra milioni, grazie all utilizzo di anticorpi
WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING SCOPO DEL WESTERN BLOTTING Tecnica che consente di visualizzare/individuare UNA proteina tra milioni, grazie all utilizzo di anticorpi
Lezione n. 11. Reazioni enzimatiche Michaelis-Menten Dipendenza di k da T. Antonino Polimeno 1
 Chimica Fisica Biotecnologie sanitarie Lezione n. 11 Reazioni enzimatiche Michaelis-Menten Dipendenza di k da T Antonino Polimeno 1 senza catalizzatore... con catalizzatore... Antonino Polimeno 2 Catalisi
Chimica Fisica Biotecnologie sanitarie Lezione n. 11 Reazioni enzimatiche Michaelis-Menten Dipendenza di k da T Antonino Polimeno 1 senza catalizzatore... con catalizzatore... Antonino Polimeno 2 Catalisi
